Die Komponenten der sechsten Datenfreigabe von GP2

Übersicht
Im Dezember 2023 kündigte GP2 die sechste Datenfreigabe in Kooperation mit AMP® PD auf der Terra- und der Verily®-Workbench an. Zusätzlich zu den Beständen der früheren Freigaben des Netzwerks Komplexe Krankheit und des Monogenic-Netzwerks umfasst diese Version über 20.000 zusätzliche Teilnehmende.
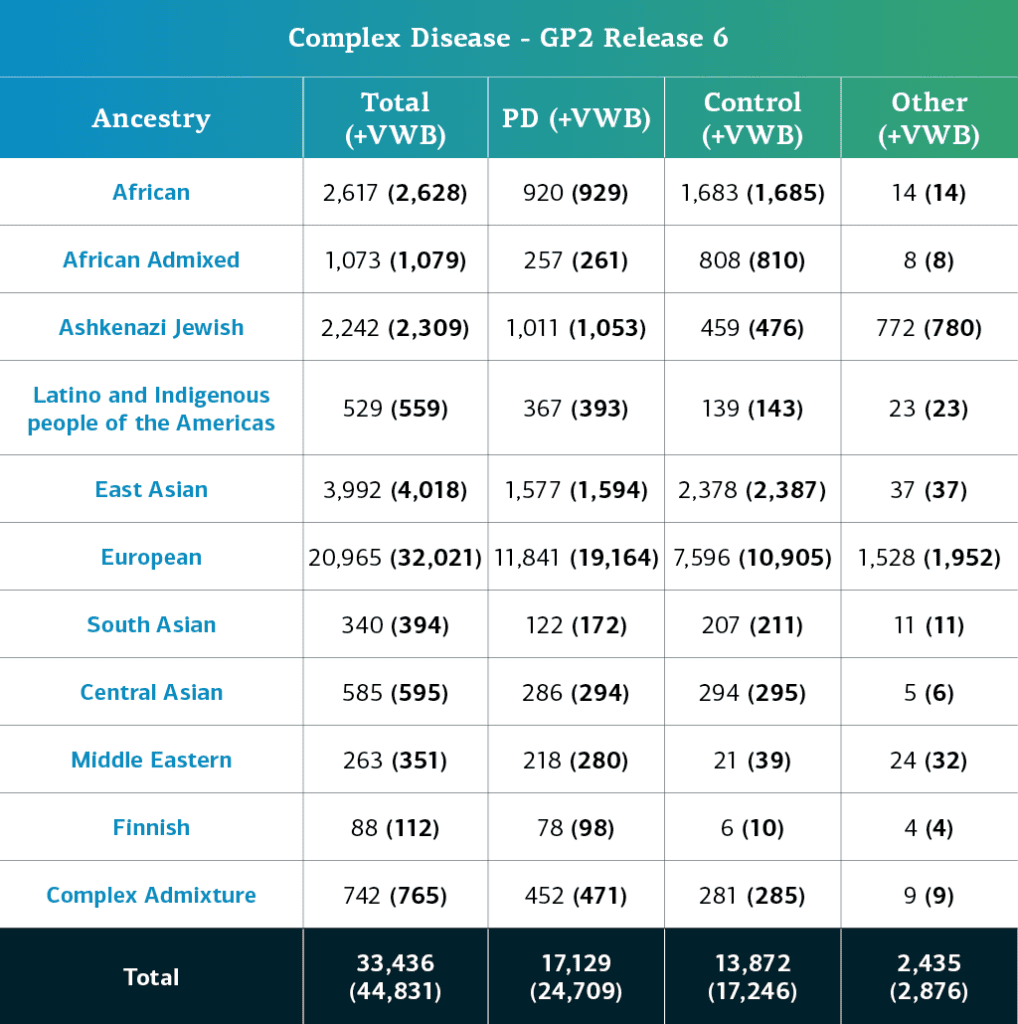
- Die Daten zur komplexen Krankheit (Genotypen), einschließlich Proben mit lokalen Einschränkungen, umfassen nun insgesamt 44.831 genotypisierte Teilnehmende (24.709 mit Parkinson-Erkrankung, 17.246 Kontrollpersonen und 2876 „sonstige“ Phänotypen).
- Abzüglich der Proben mit lokalen Einschränkungen beträgt die Zahl nun 33.436 (17.129 mit Parkinson-Erkrankung, 13.872 Kontrollpersonen und 2.435 „sonstige“ Phänotypen).
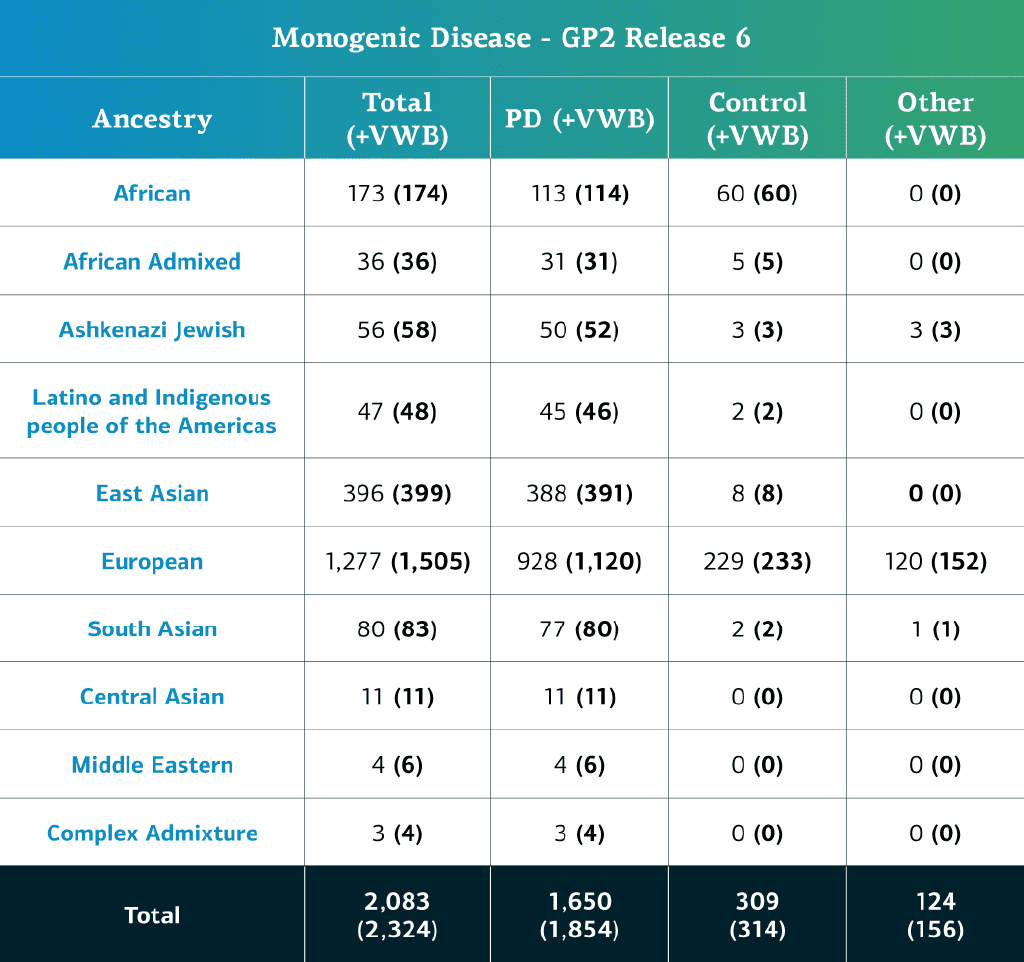
- Die Daten zu monogenen Erkrankungen (Gesamtgenomsequenzen) umfassen nun insgesamt 2.324 sequenzierte Teilnehmende (1.854 mit Parkinson-Erkrankung, 314 Kontrollpersonen und 156 „sonstige“ Phänotypen).
- Abzüglich der Proben mit lokalen Einschränkungen beträgt die Zahl nun 2.083 (1.650 mit Parkinson-Erkrankung, 309 Kontrollpersonen und 124 „sonstige“ Phänotypen).
- Zu 12.585 Personen mit tiefen klinischen Phänotypisierungsinformationen liegen auch passende genetische Informationen vor.
Was ist in dieser Freigabe neu?
- Zusätzliche Proben zu komplexer Krankheit (genotypisiert) und monogenetischer Krankheit (Ganzgenomproben)
- Einführung von DSGVO-Proben mit lokalen Einschränkungen über die Verily Viewpoint Workbench
- Einführung klinischer Daten für rund 12.000 Personen
- Einführung einer neuen Abstammungsgruppe → Complex Admixture History (CAH)
- Aktualisierungen bei den Qualitätskontrollmaßnahmen für freigegebene Genotypisierungsdaten
- Aktualisierungen bei der Variantenerkennung, jetzt mit DeepVariant, für freigegebene Ganzgenomdaten
Aktualisierungen bei der Qualitätskontrolle
Kurz gesagt umfasst die Qualitätskontrolle von GenoTools (v1.0.0) folgende Prüfschritte: Fehlzuweisungen des Geschlechts, Pruning der Call-Rate, Doubletten, Überprüfung und Meldung von verwandten Individuen und Überprüfung der Heterozygotie-Rate. Anders als bei bisherigen Freigaben nehmen wir nun die Filterung auf Variantenebene nach folgenden Kriterien nicht mehr vor: Frequenz des selteneren Allels (MAF), Hardy-Weinberg (HWE), Minor Allele Count (MAC). Wenn Sie so filtern möchten, wie wir es bei vorherigen Freigaben getan haben, empfehlen wir die entsprechende README-Datei. Dort finden Sie ausführliche Informationen und empfohlene Schwellwerte.
Complex Admixture History (CAH)
CAH, oder Complex Admixture History, ist eine neue Abstammungsgruppe, die bei GP2 für die sechste Freigabe eingeführt wurde. Sie wurde erstellt, weil in Version 5 viele südafrikanische Proben und andere Proben mit hoher Beimischung fälschlicherweise als CAS-Abstammung (zentralasiatisch) eingestuft wurden. in der sechsten Freigabe enthält die Abstammungsgruppe CAH hauptsächlich Proben von der Stellenbosch University (Kapstadt, Südafrika), The Coriell Institute (Camden, New Jersey, USA) und der Parkinson’s Foundation (Miami, Florida, USA). Wir gehen davon aus, dass alle als CAH eingeordneten Proben zu stark durchmischt sind, um sie bei Analysen mit anderen GP2-Abstammungsgruppen zu verwenden.
DSGVO-Proben mit lokalen Einschränkungen über Verily Viewpoint Workbench
Wir freuen uns sehr, bekannt geben zu können, dass einige Nutzer*innen durch unsere Zusammenarbeit mit der Verily Viewpoint Workbench Zugang zu lokal begrenzten Proben haben werden, d. h. Proben, die der Datenschutzgrundverordnung (GDPR) unterliegen. Workbench ist eine sichere Umgebung für die Verwaltung und Analyse biomedizinischer Daten zur Verbesserung der Forschungszusammenarbeit und der Reproduzierbarkeit von Daten durch Cloud-Integration. Die Lösung unterstützt die gemeinsame Nutzung von Workspaces einschließlich Python- und R-Code und bietet eine Reihe von cloud-nativen Diensten für die Datenverwaltung und -analyse. Workbench unterstützt eine sichere Integration der Datennutzung und benutzerdefinierte Authentifizierung. Dadurch ist es die perfekte, sichere und skalierbare Forschungsumgebung für GP2, um DSGVO-Proben mit lokalen Einschränkungen zu hosten.
Da GP2 aktuell weiter daran arbeitet, Lösungen für den Austausch DSGVO-geschützter Daten einzuführen, ist die sechste Freigabe nur für Mitglieder des GP2-Forschungsverbunds sowie Partner verfügbar. Im Zuge der weiteren Tests und Implementierung Anfang 2024 werden diese Lösungen der breiteren Forschungsgemeinde zur Verfügung stehen. Alle Proben der Freigabe 6 sind auf der Workbench zu finden, während alle Proben der Freigabe 6, die nicht den DSGVO-Anforderungen unterliegen, auf der Community Workbench auf Terra zu finden sind (so wie alle Vorversionen). Um Zugang zur vollständigen Freigabe auf der Verily Viewpoint Workbench zu erhalten, müssen Sie folgende Voraussetzungen erfüllen:
- über einen bewilligten GP2-Tier-2-Zugang verfügen,
- Mitglied des GP2-Forschungsverbunds sein (beitragende Kohorte, GP2-Partner oder Projektanalyse-Teammitglied),
- das Antragsformular für DSGVO-Proben ausfüllen. Nachdem Sie das Formular ausgefüllt haben, erhalten Sie Anweisungen für den Workbench-Zugang.
Klinische Daten
Wir sind auch sehr erfreut, mit dieser Freigabe umfassende tiefe klinische Phänotypdaten für 12.585 Personen bereitstellen zu können. Diese Informationen umfassen:
- Alter bei Diagnose und Ausbruch
- Primäre, aktuelle und letzte Diagnose
- Kognitive Tests wie der Mini-Mental Status-Test (MMST) und der Montreal-Cognitive-Assessment-Test (MoCa)
- Von der Movement Disorder Society gesponserte Überarbeitung der Unified Parkinson’s Disease Rating Scale (MDS-UPDRS)
- Detaillierte „sonstige“ Phänotypen wie etwa Lewy-Körper-Demenz (LBD)
In dieser Freigabe liegen zu jeder der 12.585 Personen mit klinischen Informationen auch passende genetische Informationen vor.
Individuen-spezifische Daten
Wir erfassen nun die Daten von insgesamt 74 Kohorten, 46 davon sind neu in dieser Freigabe. Weitere Informationen zu den bereitgestellten Kohorten finden Sie im GP2 Cohort Dashboard.
Die genetisch determinierte Abstammung der GP2-Teilnehmenden für komplexe Krankheiten ist in 11 Abstammungsgruppen unterteilt. Die nachstehende Tabelle enthält Angaben zur genetisch determinierten Abstammung von Teilnehmenden mit komplexer Krankheit in dieser Freigabe, für die eine erfolgreiche Qualitätskontrolle sowie Imputation erfolgt ist. Diese Zahlen beinhalten Proben aus früheren Freigaben, die unter Verwendung der neuen Cluster-Datei neu geclustert wurden und die Qualitätskontrolle durchlaufen haben, sowie die neu genotypisierten und gemeinsam genutzten Proben, die nur in dieser aktuellen Freigabe enthalten sind.
Variantenidentifizierung aus Gesamtgenomsequenzen mit DeepVariant-GLnexus
Im Gegensatz zu vorherigen Freigaben verwenden wir nun dieDeepVariant-Pipeline von Google in Kombination mit GLnexus zur Variantenerkennung auf Kohortenebene. DeepVariant ist ein Deep-Learning-basiertes Programm zur Variantenerkennung, das den bisherigen Spitzentechnologie-Tools überlegen ist, da es genetische Varianten auf individueller Ebene genau erkennt. Zudem vereinfacht es den Prozess bei gleichzeitiger Verbesserung von Präzision und Zuverlässigkeit.
Durch zukünftige Datenfreigaben wird sich die Vielfalt der verfügbaren Teilnehmenden weiter erhöhen. Auf unserem Dashboard können Sie sich über den Fortschritt unserer Arbeiten informieren. Wer bereits über einen Tier-2-Zugang verfügt, kann die Daten in unserem Kohorten-Browser ansehen, der bereits in einem Blogbeitrag vorgestellt wurde.
Wie immer finden Sie weitere Details zu Pipelines, Daten und Analysen in der README-Datei zur jeweiligen GP2-Freigabe!