GP2第六次数据发布内容

概述
2023 年 12 月,GP2 宣布与 AMP® PD 合作,在 Terra 和 Verily® Workbench 平台上发布第六次数据。除了之前发布的复杂疾病网络和单基因网络数据外,此次发布的数据又增加了超过20000名参与者。
- 复杂疾病数据(基因型),包括局部限制样本,现在总共包括 44,831个基因型参与者(24,709例PD病例、17,246例对照和 2,876例”其他 “表型)
- 去掉局部限制样本后,现在共有 33,436 人(17,129 例PD病例、13,872 例对照和 2,435例 “其他 “表型)
- 单基因疾病数据(全基因组序列)现在总共包括 2,324 个测序参与者(1,854例PD、314例对照和 156例”其他 “表型)
- 去除局部限制样本后,目前共有 2,083 人(1,650 例PD、309 例对照和124 例 “其他 “表型)。
- 有深度临床表型信息的 12,585人也有匹配的基因信息
此次发布有哪些新内容?
- 新增复杂疾病(基因分型)和单基因疾病(全基因组)样本
- 通过 Verily Viewpoint Workbench引入本地限制的 GDPR 样本
- 引入约 12,000 个体的临床数据
- 引入新的祖源组 → 复杂混杂史 (CAH)
- 对已发布基因分型数据质控措施的最新进展
- 基因组变异检测最新进展,现使用 DeepVariant,用于已发布的全基因组数据
质量控制最新进展
简言之,GenoTools(v1.0.0)执行以下质量控制步骤:性别错配、检测率调整、重复检查、检查和报告相关个体以及杂合率检查。
与之前的发布相比,我们不再执行以下变异级过滤:小等位基因频率 (MAF)、Hardy-Weinberg (HWE) 或小等位基因计数 (MAC)。如果您想按照我们之前发布的方法进行筛选,建议查阅相应的 README 以获取详细信息和建议阈值。
复杂混血史(CAH)
CAH,即复杂混血史,是 GP2第六次发布引入的全新的祖源组。是针对第五次发布中南非和其他高度混血的个体被错误地预测为CAS(中亚)血统的大量样本而创建的。在第六次发布中,CAH 祖源组主要包含来自斯泰伦博斯大学(南非开普敦)、科里尔研究所(美国新泽西州卡姆登)和帕金森基金会(美国佛罗里达州迈阿密)的样本。我们认为任何标注为 CAH 的样本混杂程度太高,无法纳入其他 GP2 祖源组一起分析。
通过 Verily Viewpoint Workbench访问受本地限制的 GDPR 样本
我们很高兴地宣布,通过与 Verily Viewpoint Workbench 的合作,部分用户将能够访问受本地限制的样本,也就是受《一般数据保护条例》(GDPR)政策管辖的样本。Workbench 是一个用于管理和分析生物医学数据的安全环境,旨在通过云端集成加强研究合作和数据可重复性。它支持工作空间共享,包括 Python 和 R 代码,并为数据管理和分析提供一套云原生服务。Workbench 支持安全数据使用集成和自定义身份验证,是 GP2 托管本地受限样本的理想、安全和可扩展的研究环境。
目前,随着 GP2 继续推出受 GDPR 保护数据的数据共享解决方案,第六次发布的内容将只提供给 GP2 联盟成员和合作伙伴。随着 2024 年初测试和实施工作的继续开展,这些解决方案将向更广泛的研究社区开放。第六次发布的所有样本都可以在Workbench上找到,而不受 GDPR 限制的所有第六次发布样本都可以在 Terra 上的社区工作台上找到(与之前的所有版本一样)。要访问 Verily Viewpoint Workbench完整版本,您须:
- 有GP2二级级访问权限
- 是GP2联盟成员(贡献了队列、GP2合作伙伴或项目分析小组成员)
- 填写 GDPR规定的样本申请表。填写表格后,您将收到访问Workbench的后续说明。
临床数据
我们还非常高兴地宣布,本次发布的数据中包含 12,585 例的全面深度临床表型数据。这些信息包括:
- 确诊和发病年龄
- 主要诊断、当前诊断和最新诊断
- 认知检查,如小型精神状态检查 (MMSE) 和蒙特利尔认知评估 (MoCA)
- 运动障碍协会赞助的统一帕金森病评分量表修订版(MDS-UPDRS)
- 详细的 “其他 “表型,如路易体痴呆 (LBD)
在这次发布中, 12,585 例有临床信息的个人都有匹配的基因信息。
个体级别的数据
我们现在总共采集了 74 个队列的数据,其中 46 个队列是本次发布的新数据。有关已共享队列的更多信息,请参阅 GP2 队列看板。 根据基因确定的GP2复杂疾病参与者的祖源分为 11 个。下表详细列出了本版本中通过质量控制和估算的复杂疾病参与者根据基因确定的祖源。这些数字包括以前版本的样本,这些样本已经使用新的集群文件重新聚集,并通过了质量控制,同时还包括本次发布的新基因型和共享样本。使用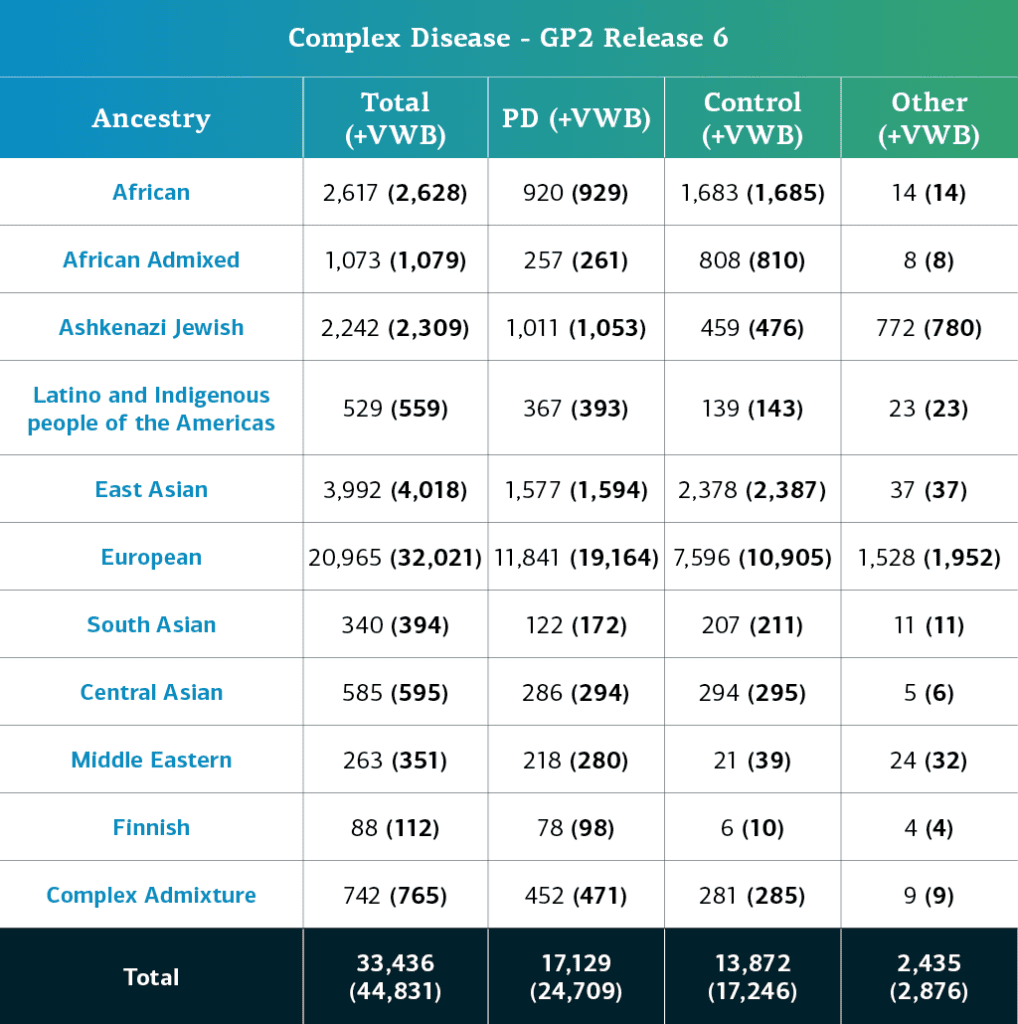
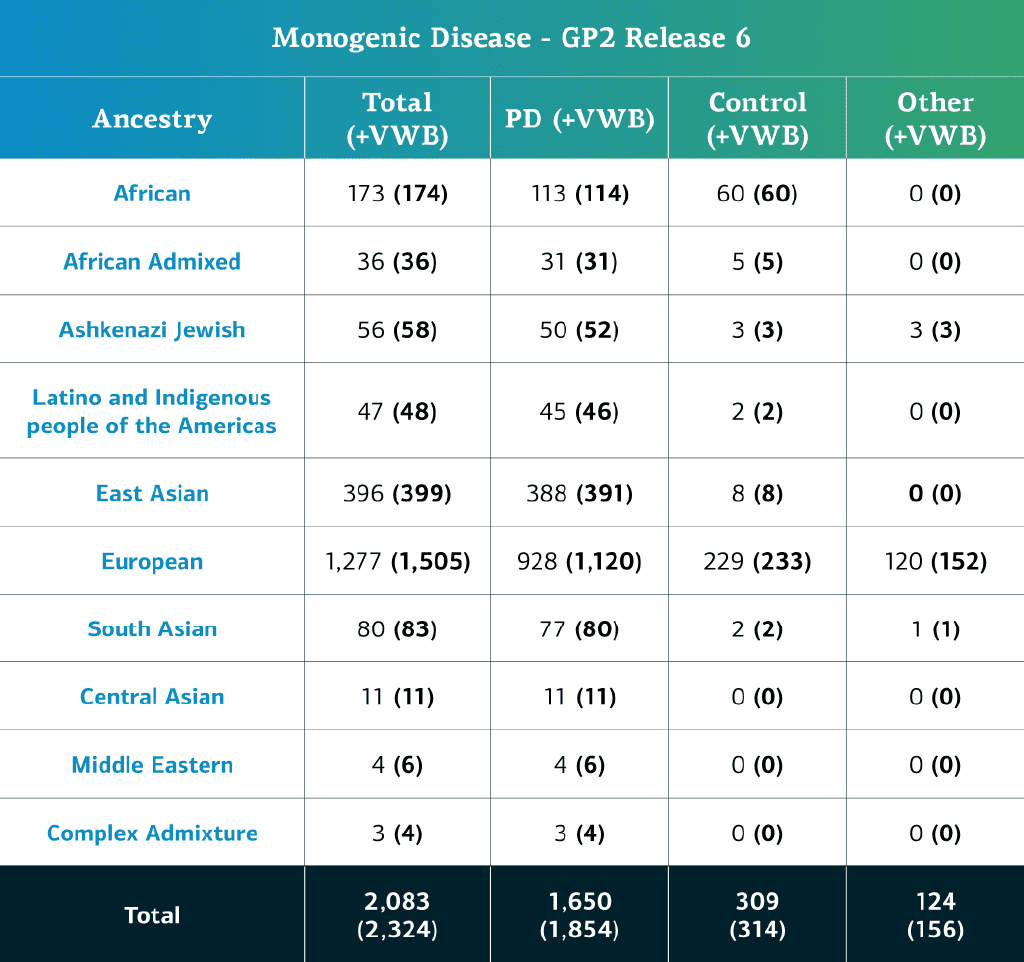
DeepVariant-GLnexusde变异检测的全基因组序列。
在这次发布中,与以前的版本不同,我们现在使用Google的DeepVariant管道和GLnexx来进行队列级别的变量调用。DeepVariant是一款基于深度学习的变体调用器,通过准确的在个体级别上检测出基因变异,其性能优于现有的最先进工具。它还简化了过程,提高了准确性和可靠性。
未来的数据发布还将继续提高参与者的多样性。可在看板上查看我们的进展。对于已经拥有二级访问权限的用户,您可以在我们的队列浏览器上进一步浏览数据,之前的 博客文章中对此进行了详细阐述。
关于管道、数据和分析的更多详细信息,请参考每期GP2发布所附带的README!











