Eléments de la première publication de données du GP2

Au mois de décembre 2021, le GP2 a annoncé sa toute première publication de données sur la plateformeAMP® PD . Les données concernent 4 908 participants (3 434 atteints de la maladie de Parkinson et 1 474 non atteints) des cohortes de l’institut CORIELL, du Baylor College of Medicine (BCM) et du BCM-University of Maryland (UMD). L’ascendance génétiquement déterminée des participants au GP2 est répartie en 8 groupes d’ascendance ; le tableau ci-dessous présente l’ascendance génétiquement déterminée des participants compris dans la première publication du GP2. 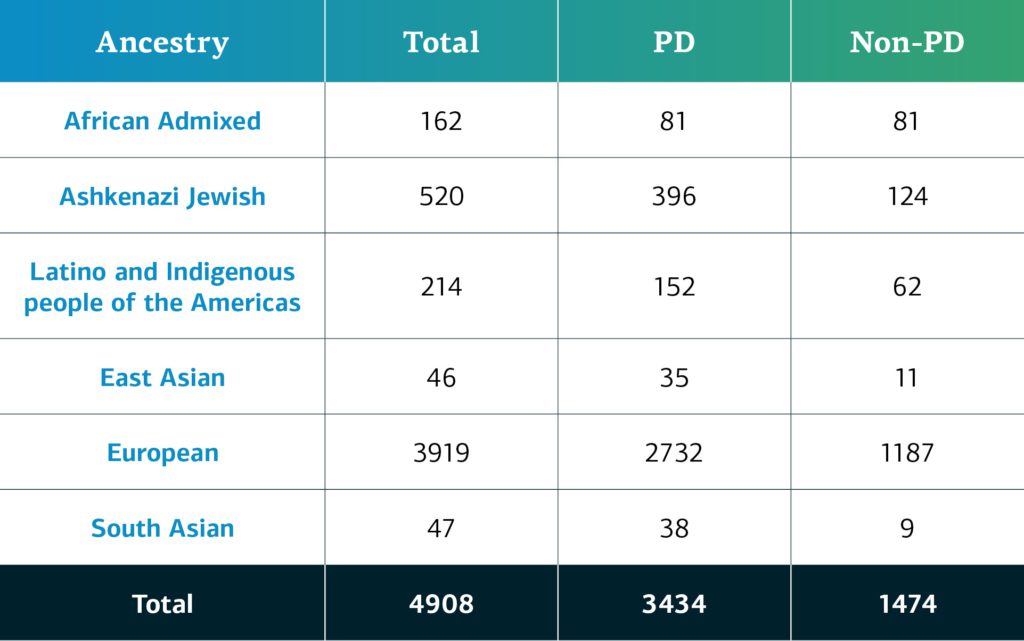 Les futures publications de données contribueront à renforcer la diversité des participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès.
Les futures publications de données contribueront à renforcer la diversité des participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès.
Données de génotype
Le génotypage de l’ensemble des données a été effectué au moyen de l’outil NeuroBooster d’Illumina qui a été spécialement conçu pour les variants des maladies neurodégénératives chez diverses populations. Le traitement des données est réalisé conformément aux protocoles standard d’Illumina et le contrôle de la qualité est effectué par le GP2 via une pipeline de CQ ouverte. Les données sont ensuite imputées au moyen de TOPMed dans chaque groupe d’ascendance distinct. Tous les doublons connus sont retirés mais les prélèvements associés sont conservés de façon à permettre une plus grande flexibilité dans les analyses des communautés. Pour plus d’informations sur les éléments de la pipeline de CQ, suivez le flux de travail indiqué dans la GitHub page correspondante.
Données cliniques :
Les données cliniques ont été harmonisées pour former un jeu de données cliniques uniformisé du GP2. Les exigences minimales pour les données cliniques sont les suivantes : données démographiques, catégorie de recrutement et antécédents familiaux. Pour plus d’informations sur les degrés d’exigence des données fondamentales du GP2, consultez le document « Ensemble de données cliniques fondamentales » dans la section du site web. La plateforme Terra permet d’accéder aux données. Pour savoir comment accéder aux données du GP2, lisez l’article de blog suivant : «Demande d’accès aux données du GP2 sur la plateforme AMP® PD». La première publication de données du GP2 comprend les éléments suivants, accessibles via Terra :
- Accès aux données de niveau 1
- Statistiques de synthèse des études d’association pangénomique de la maladie de Parkinson les plus récentes (à l’exclusion des échantillons 23andMe de Nalls et al. 2019).
- Accès aux données de niveau 2
- Génotypes brutes : Fichiers PLINK2 .pgen pour chaque groupe d’ascendance pour tous les prélèvements validés par le contrôle de qualité avant l’imputation.
- Génotypes imputés : Fichiers PLINK2 .pgen imputés à l’aide du panel de référence TOPMed (satisfaisant au filtrage minimum du CQ décrit dans le fichier readme de la publication des données).
- Méta-données : contient des informations relatives aux résultats du CQ et à la prédiction d’ascendance.
- Données cliniques : champs de données cliniques et dictionnaire de données cliniques pertinent.
Nous espérons que ces données seront utiles à la communauté de chercheurs. Alors que nous améliorons sans cesse les mécanismes de notre organisme, nous nous réjouissons à l’idée de vous communiquer d’autres actualités d’intérêt à l’avenir.  Ce blog a été co-écrit par Hampton Leonard, Mike Nalls, Yeajin Song et Dan Vitale. Veuillez consulter la page du GP2 du groupe de travail sur l’analyse des données – Maladie complexe pour en savoir plus sur les auteurs.
Ce blog a été co-écrit par Hampton Leonard, Mike Nalls, Yeajin Song et Dan Vitale. Veuillez consulter la page du GP2 du groupe de travail sur l’analyse des données – Maladie complexe pour en savoir plus sur les auteurs.



