Im Dezember 2021 kündigte GP2 die erste Freigabe von GP2-Daten auf der AMP®-PD-Plattform an. Die verfügbaren Daten stammen von 4.908 Teilnehmer*innen (3.434 Parkinson, 1.474 kein Parkinson) aus den Kohorten CORIELL, Baylor College of Medicine (BCM) und BCM-University of Maryland (UMD). Die genetisch determinierte Abstammung von GP2-Teilnehmer*innen wird in acht Abstammungsgruppen gegliedert; die Tabelle unten gibt Aufschluss über die genetisch determinierte Abstammung der Teilnehmer*innen in der Freigabe „GP2 Release 1“.
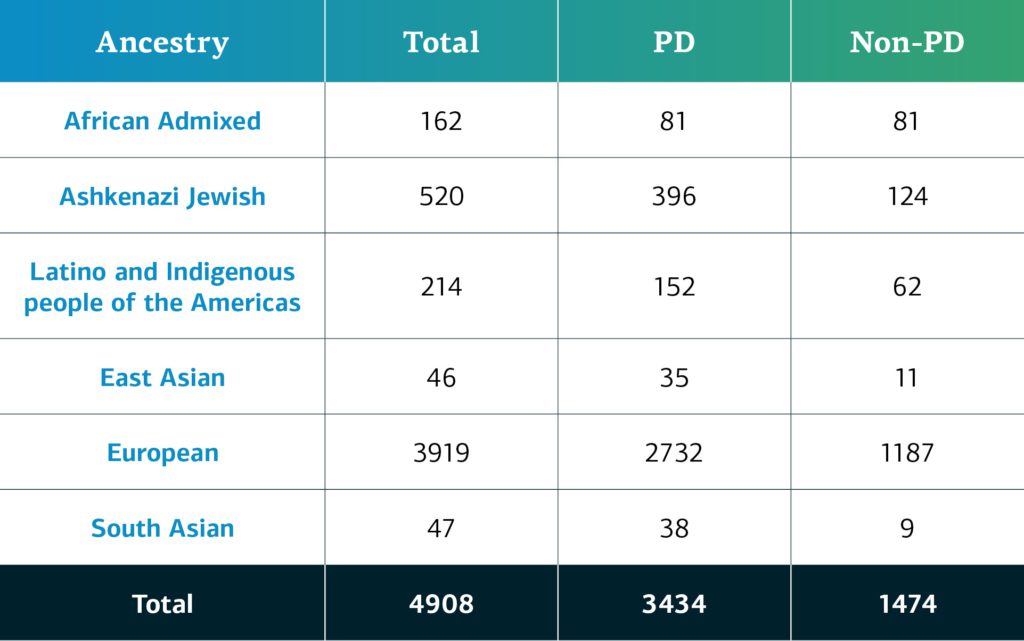
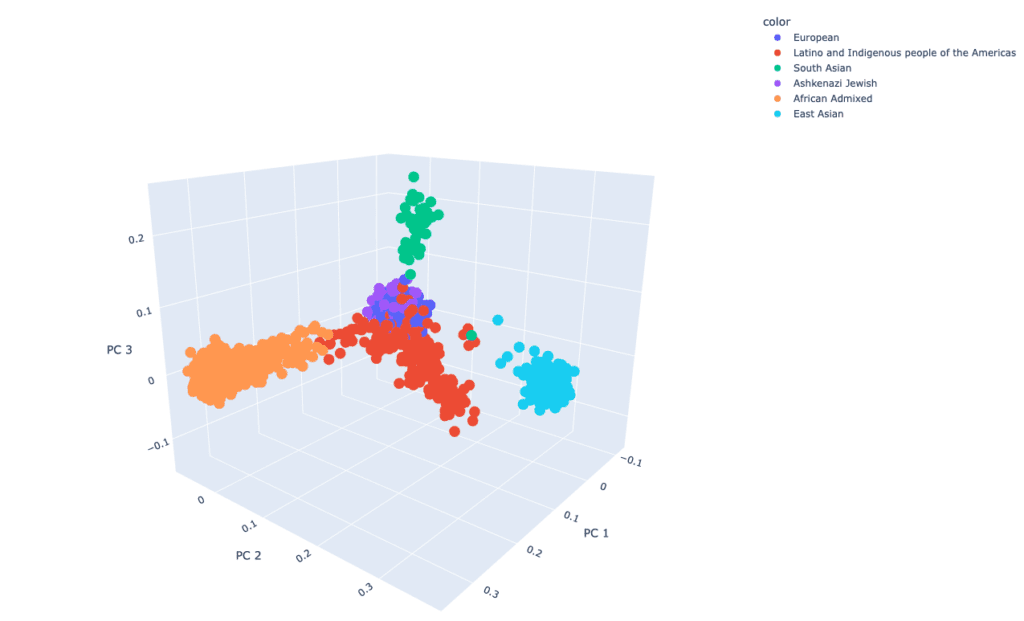
Durch zukünftige Datenfreigaben wird sich die Vielfalt der verfügbaren Teilnehmenden weiter erhöhen. Auf unserem Dashboard können Sie sich über den Stand unserer Fortschritte informieren.
Genotypdaten
Alle Daten wurden mit dem Illumina NeuroBooster Array genotypisiert – einem Array, das speziell für Varianten neurodegenerativer Erkrankungen in verschiedenen Bevölkerungsgruppen entwickelt wurde. Die Daten werden gemäß den Standardprotokollen von Illumina verarbeitet, und die Qualitätskontrolle für die Daten wird von GP2 mithilfe unserer Open-Source-QC-Pipeline durchgeführt. Die Daten werden dann unter Verwendung von TOPMed innerhalb jeder einzelnen Abstammungsgruppe separat imputiert. Alle bekannten Duplikate werden entfernt; verwandte Proben bleiben jedoch erhalten, um die Flexibilität bei Gemeinschaftsanalysen zu erhöhen.
Weitere Informationen zum Inhalt der QC-Pipeline finden Sie im ausführlichen Workflow auf der zugehörigen GitHub-Seite.
Klinische Daten
Klinische Daten wurden zu einem einheitlichen klinischen GP2-Datensatz harmonisiert. Zu den Mindestanforderungen an die klinischen Daten gehören demografische Angaben, die Rekrutierungskategorie und die Familienanamnese. Weitere Informationen zu den GP2-Kerndatenanforderungsstufen finden Sie im Dokument „ Kerndatensatz für klinische Daten “ im Abschnitt „ “ auf unserer Website.
Der Zugriff auf die Daten erfolgt über die Terra-Plattform.
Um herauszufinden, wie Sie auf GP2-Daten zugreifen können, lesen Sie bitte unseren Blog „ Beantragung des GP2-Datenzugriffs auf der AMP® PD-Plattform “. Die folgenden Datenprodukte sind auf Terra für unsere Freigabe „GP2 Release 1“ verfügbar:
- Tier-1-Datenzugriff
- Zusammenfassende Statistiken der jüngsten GWAS zur Parkinson-Krankheit (ohne 23andMe-Proben von Nalls et al. 2019).
- Tier 2-Datenzugriff
- Genotyp-Rohdaten: samples passing quality control prior to imputationPLINK2 .pgen-Dateien für jede Abstammungsgruppe für alle Proben, die die Qualitätskontrolle vor der Imputation bestanden haben
- Imputierte Genotyp-Daten: PLINK2 .pgen-Dateien, die mit dem TOPMed-Referenzpanel imputiert wurden (und die Mindest-QC-Filterung gemäß Readme-Datei zur Datenfreigabe erfüllen)
- Metadaten: enthält Informationen über QC-Ausgaben und Vorhersage der Abstammung
- Klinische Daten: klinische Datenfelder und relevantes klinisches Datenwörterbuch
Wir hoffen, dass dieser Datenbestand für die Parkinson-Forschungsgemeinschaft zu einer wertvollen Ressource wird. Wir arbeiten kontinuierlich an der Verbesserung unserer Prozesse und haben noch viele weitere spannende Dinge in Petto.
Dieser Blogbeitrag wurde gemeinschaftlich von Hampton Leonard, Mike Nalls, Yeajin Song und Dan Vitale verfasst. Wenn Sie mehr über die Autoren erfahren möchten, besuchen Sie gerne die Webseite der GP2 Complex Disease – Data Analysis Working Group.
Schauen Sie sich unsere anderen Datenveröffentlichungen an.


