GP2第一次数据发布内容

2021年12月GP2宣布在 AMP® PD平台上进行首次数据发布。发布的数据包括来自科里耶尔医学研究所(CORIELL)、贝勒医学院 (BCM) 和 BCM-马里兰大学 (UMD)队列的 4908 名参与者(3434名 PD,1474名非 PD)。GP2数据发布的参与者根据他们基因决定的祖先(GDA)分为八个祖先组;下表详细介绍了 GP2 首次数据发布中参与者的基因决定的祖先信息。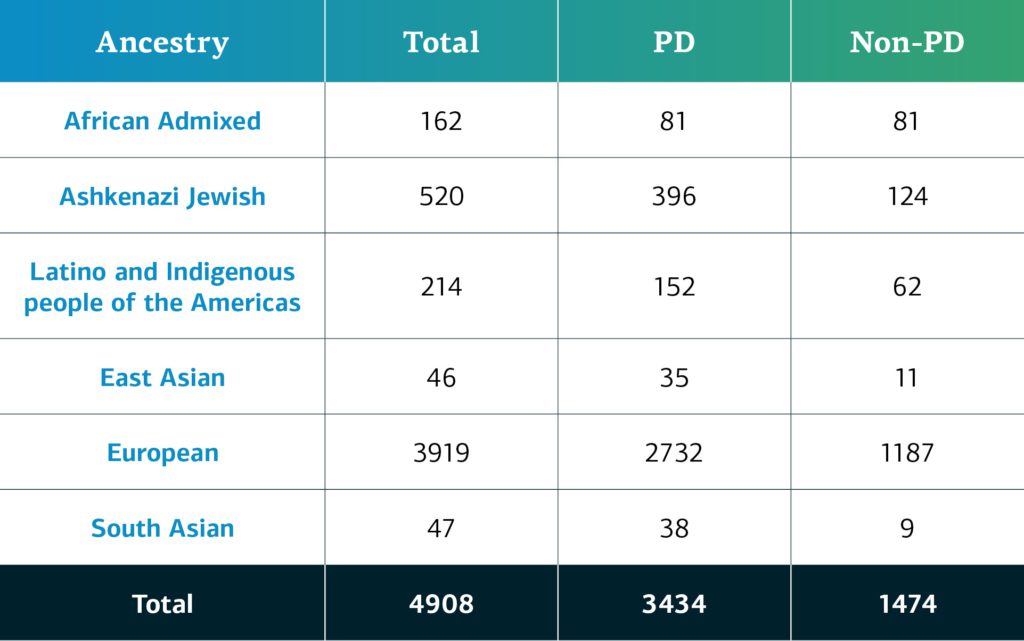 未来的数据发布还将继续提高参与者的多元性。您可以在我们的Dashboard上查看我们的进展。
未来的数据发布还将继续提高参与者的多元性。您可以在我们的Dashboard上查看我们的进展。
基因型
所有数据均在Illumina NeuroBooster Array上进行基因分型,该阵列专为不同人群中神经退行性疾病变异而设计。数据根据标准Illumina协议进行处理,数据的质量控制由GP2用我们的开源QC管道进行处理。然后在每个的祖先组中使用TOPMed进行数据填补。所有已知的重复数据都被删除,但相关的样本仍予以保留,以促进社区分析的灵活性。 如果您想了解QC管道的更多详情,请关注GitHub页面上详尽的工作流程。
临床数据
协调临床数据,以形成统一的GP2临床数据集。对临床数据的最低要求包括人口统计特征、招募类别和家族史。 如果您想进一步了解GP2核心数据集要求,请参考我们网站的中的“临床数据核心数据集” 文件。数据将通过Terra平台访问。 如果您想了解如何访问GP2数据,请阅读我们的博文《申请在AMP® PD平台上访问GP2数据 》。您可以在Terra上找到GP2首次数据发布中的以下的数据产品:
- 第一层数据访问
- 最新的帕金森病 GWAS 的汇总统计数据(不包括来自Nalls et al.2019的23andMe样本)。
- 第二层数据访问
- 原始基因型:为了在填补之前让所有样本通过质量控制,每个祖先组的PLINK2 .pgen文件
- 已填补的基因型:PLINK2.pgen文件使用美国精准化医学研究计划(TOPMed)参考面板进行填补(通过最低限度的质量控制过滤,详见随数据发布的readme文件)。
- 元数据: 包含与质量控制(QC)输出和祖先预测有关的信息
- 临床数据: 临床数据字段和相关的临床数据字典
我们希望这能成为帕金森病研究界的一个宝贵资源。我们还在不断地优化我们的流程,未来还会为大家带来更多惊喜。
本博文由Hampton Leonard、Mike Nalls、Yeajin Song和Dan Vitale共同撰写。请访问GP2的复杂疾病-数据分析工作组页面了解作者的背景。



