La maladie de Parkinson a fait l’objet de recherche génétique à grande échelle. Les chercheurs ont identifié plus de 90 variants génétiques susceptibles d’influer sur le degré de risque de développer un Parkinson, et ont ainsi révélé que c’est une maladie complexe impliquant plusieurs voies moléculaires.
Il n’a cependant pas été possible d’obtenir une vision d’ensemble, une compréhension englobante de la maladie de Parkinson en raison du manque de diversité génétique dans les études réalisées sur la maladie Parkinson, lesquelles étaient grandement axées sur les populations d’ascendance européenne. Quels seraient les variants génétiques de la maladie de Parkinson à l’origine du lien de causalité ? Quels variants seraient les plus pertinents pour toutes les populations, d’un point de vue clinique? Pour pouvoir répondre à ces questions, il nous faut nécessairement une plus grande diversité de données d’ascendance.
Une collaboration mondiale : Ascendance diversifiée et grande échelle
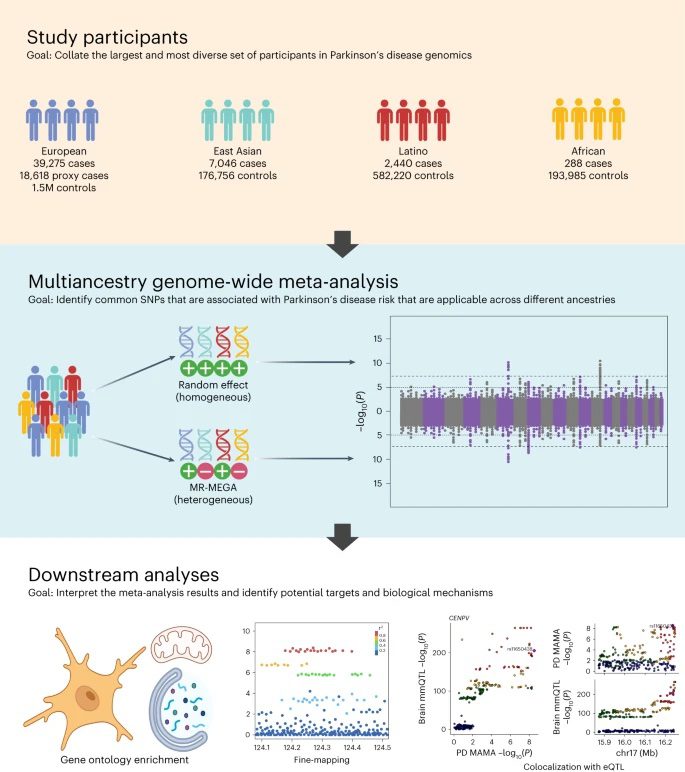
La nouvelle étude collaborative publiée dans Nature Genetics offre une compréhension plus inclusive et mondiale du paysage génétique de la maladie de Parkinson. Il s’agit de l’étude la plus étendue et la plus diversifiée en son genre grâce, en grande partie, à la collaboration mondiale et aux efforts de partage de données engagés par leProgramme mondial sur la génétique de la maladie de Parkinson (GP2). Cette étude analyse les données de 49 049 sujets atteints de parkinsonisme, 18 785 personnes dont les parents sont atteints ou ont été atteints de parkinsonisme, et plus de 2 millions de personnes neurologiquement en bonne santé, d’ascendance européenne, d’Asie de l’Est, d’Amérique latine et d’Afrique.
Les chercheurs du NIH, du GP2, du 23andMe ainsi que des collaborateurs aux Etats-Unis, au Royaume-uni, au Pérou et à Singapour ont sourcé cette diversité de données sur FinnGen, 23andMe,UK Biobank et autres bases de données en accès libre. A l’aide de ces données, ils ont pu identifier 78 positions du génome (« loci ») clairement associées à la maladie de Parkinson.
L’analyse des données à partir de multiples groupes d’ascendance représente un vrai défi, mais l’émergence de nouveaux logiciels et outils d’analyse permettent de tirer parti de la diversité grandissante des données. Grâce à ces outils et efforts de collaboration d’un bout à l’autre de la planète, les chercheurs peuvent désormais identifier des positions génétiques associées au risque de parkinsonisme sur des populations d’ascendance variée. Il est probable que ces positions contiennent des variants de causalité.
Pour cette étude, les chercheurs ont utilisé une approche dite méta-analyse pangénomique à pluri-ascendance. L’association pangénomique est une méthode qui implique de répertorier les génomes de plusieurs sujets, de chercher les séquences de variants spécifiques sur les positions génomiques (« loci ») que l’on retrouve plus fréquemment chez ceux souffrant d’une maladie spécifique comparé à ceux qui n’en souffrent pas. Les chercheurs ont récupéré les données de plusieurs études d’association pangénomiques passées et plusieurs bases de données pour ajouter de la force et de la solidité statistique à la recherche sur les facteurs de risques associés à la maladie de Parkinson.
En incluant les données de populations non-européennes, les chercheurs ont pu identifier plusieurs nouveaux loci génétiques qui n’avaient pas été observés dans les populations d’ascendance européenne. Si la majorité des 78 positions de la maladie de Parkinson identifiées dans cette étude correspond aux positions déjà associées à cette maladie, 12 d’entre elles occupent de nouvelles positions. Il semble que ces nouvelles positions impliquent des gènes qui participent à la décomposition ou à la métabolisation des protéines, au contrôle de l’activité des mitochondries et au transport des réponses immunitaires dans le cerveau.
La plupart des positions identifiées étaient associés au risque de Parkinson sur toutes les lignées étudiées, y compris les populations européennes. Ces résultats semblent indiquer que les nombreuses voies moléculaires à l’origine de la maladie de Parkinson sont communes à toutes les populations.
Vers une médecine personnalisée : Cartographie fine et implications fonctionnelles
Les chercheurs ont également pu dégager le variant génétique de causalité spécifique dans au moins 6 des positions identifiées, selon un processus dit de cartographie fine. La cartographie fine permet de faire une estimation des variants spécifiques au sein de chaque locus susceptible de déclencher un risque de maladie dans une population donnée.
« L’un des problème des études d’association pangénomique est que l’on ne sait jamais quel variant spécifique est de nature fonctionnelle ou causale sur un locus identifié » regretteIgnacio ”Nacho” Mata chercheur au Learner Research Institute, professeur assistant en médecine moléculaire à l’école de médecine Cleveland Clinic Learner et auteur de l’étude. « Pour chaque position ou point GWAS du génome clairement associé à la maladie, il peut y avoir des centaines de variants candidats potentiels.»
Identifier les variants fonctionnels sur un locus GWAS significatif et la façon dont ils affectent l’expression génétique est essentiel pour le suivi des études et le développement d’une thérapeutique ciblée de la maladie de Parkinson.
« En intégrant des séries de données de lignées diverses, nous avons pu réduire le nombre de variants fonctionnels pour certains des gènes sur le locus identifié » explique Nacho. « Les données de lignées diverses permettent, par exemple, d’identifier des variants pouvant être spécifiques à la population et vous permettent aussi d’identifier des variants qui sont significatifs d’une population à l’autre et donc susceptibles d’être porteur d’un facteur de risque plus élevé. »
Certains des loci identifiés avaient des effets différents selon les populations, soulignant l’importance des études génétiques se consacrant aux facteurs de risque d’ascendances spécifiques. Deux des loci identifiés uniquement auprès des populations latino américaines et africaines se trouvaient proches des gènesJAK1 et HS1BP3. Ces gènes sont tous deux impliqués dans les signaux inflammatoires et peuvent alimenter le rôle de l’inflammation dans la maladie de Parkinson.
Implications et orientations futures
Les conclusions des cette étude soulignent combien il est important d’inclure des ascendances variées dans la recherche génétique. Cette inclusivité renforce la précision de la prédiction du risque génétique et le développement de traitements personnalisés. Le GP2 est en partenariat avec des institutions qui travaillent avec des populations sous-représentées afin de générer des données sur ces communautés qui ont été négligées partout dans le monde.
« Nous avons vu des personnes du monde entier travailler ensemble et apprendre les unes des autres » dit Nacho. « Selon moi, cette collaboration est ce qu’il y a eu de mieux dans cette étude et ses promesses à venir ».
D’autres études de laboratoires seront nécessaires pour confirmer combien les loci identifiés dans cette étude touchent les voies de la maladie de Parkinson et les manifestations de la maladie. Ce travail n’en reste pas moins une base solide sur laquelle pourront s’appuyer les études à venir.
Ce travail a bénéficié du soutien de : Intramural Research Program of the National Institutes of Health (NIH), National Institute on Aging (NIA), NIH, Department of Health and Human Services; National Institute of Neurological Disorders and Stroke; Parkinson’s Foundation; Michael J Fox Foundation; Aligning Science Across Parkinson’s Global Parkinson’s Genetic Project (ASAP-GP2); American Parkinson’s Disease Association; National Medical Research Council Singapore; and Singapore Ministry of Education Academic Research Fund.
Cette étude pu être menée en utilisant les sources de la biobanque du Royaume-uni sous le numéro de dossier 33601. Nous tenons à remercier les participants et les chercheurs de l’étude du FinnGen. Nous remercions les participants aux travaux et les employés de 23andMe. Les données utilisées dans la préparation de cet article sont issues du Programme mondial sur la génétique de la maladie de Parkinson (GP2). Le GP2 est financé par l’initiative Aligning Science Against Parkinson’s (ASAP) et mis en œuvre par la Fondation Michael J. Fox pour la recherche sur la maladie de Parkinson (https://gp2.org).



