En mayo de 2023, el GP2 anunció la quinta aportación de datos en la plataforma Terra, en colaboración con AMP® PD. Los nuevos datos corresponden a 7,462 participantes nuevos con EP compleja y 487 participantes nuevos con EP monogénica, que se suman a las aportaciones anteriores de las Redes de la Enfermedad de Parkinson de Etiología Compleja y Monogénica.
- Los datos de EP compleja (genotipos) corresponden ahora a un total de 24,935 participantes genotipados (12,728 con EP; 10,533 de control y 1,674 «otros» fenotipos).
- Los datos de EP monogénica (secuencias de genoma completo) corresponden ahora a un total de 722 participantes.
Datos a nivel individual: Las nuevas cohortes agregadas al GP2 en esta aportación son:
- Arizona Brain Bank Brain y programa de donación de cuerpos (BBDP, por sus siglas en inglés), un banco de cerebros estadounidense
- Innate and Adaptive Immunity in Parkinson Disease (IMMUNEPD), un estudio estadounidense de la University of Alabama at Birmingham
- IPDGC Africa (IPDGCAF-NG), una cohorte nigeriana
- PDGENEration (PDGNRTN), una cohorte de la Parkinson’s Foundation
- Estudio genético de la enfermedad de Parkinson y trastornos del movimiento relacionados (STELLENBOS), una cohorte sudafricana de Stellenbosch University
- Malaysian Parkinson’s Genetics Cohort (UMKLM), una cohorte malaya de la Universidad de Malaya
- Correlaciones genotipo-fenotipo en la enfermedad de Parkinson y trastornos del movimiento relacionados (KUL), una cohorte monogénica de la Universidad de Malasia
- Parkinson Disease and Movement Disorders Center Biorepository (PDMDC), una cohorte monogénica de Northwestern University
La ascendencia determinada genéticamente de los participantes del GP2 con EP compleja se divide en diez grupos. La tabla siguiente presenta la ascendencia determinada genéticamente de los participantes con EP compleja de esta aportación de datos que pasaron el control de calidad y se imputaron. Estas cifras incluyen muestras de datos anteriores que ahora se reagruparon en un nuevo archivo clúster y que pasaron el control de calidad, junto con las nuevas muestras genotipadas y compartidas por primera vez en la aportación actual. 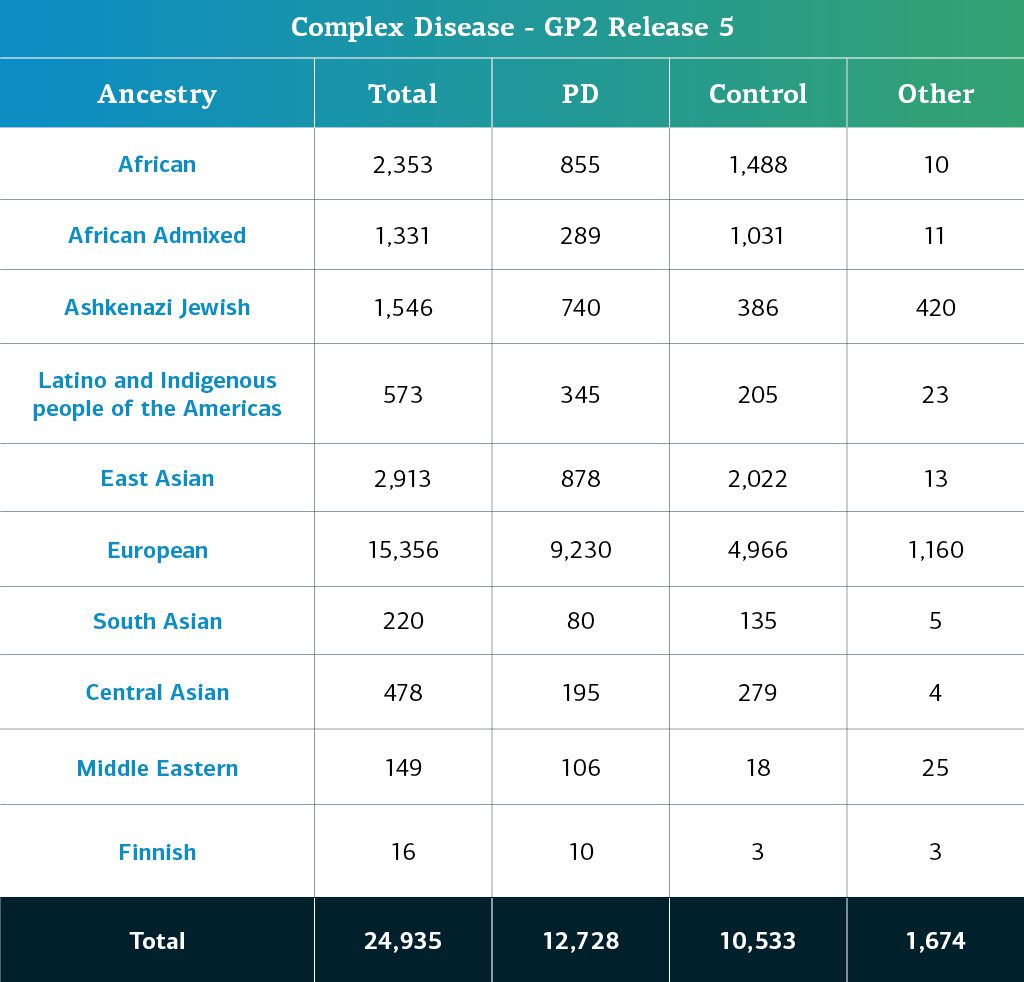 Observación importante sobre esta aportación: STELLENBOS es una cohorte sudafricana y contiene una mezcla de ascendencia genética que refleja la región y su historia. Algunas de nuestras predicciones de ascendencia para los miembros de este estudio podrían cambiar a medida que hacemos ajustes para esta nueva población en futuras aportaciones de datos. En esta aportación, hemos estimado una mezcla de ascendencia genética para todos los participantes con el fin de que los investigadores puedan tomar sus propias decisiones sobre si desean o no incluir una gran mezcla genética en sus análisis como parte de sus datos de nivel 2. También hemos incluido una lista de muestras que en estos momentos recomendamos excluir de los análisis porque sus altos niveles de ascendencia mixta reducen la confianza en las predicciones de ascendencia de nuestro modelo actual.
Observación importante sobre esta aportación: STELLENBOS es una cohorte sudafricana y contiene una mezcla de ascendencia genética que refleja la región y su historia. Algunas de nuestras predicciones de ascendencia para los miembros de este estudio podrían cambiar a medida que hacemos ajustes para esta nueva población en futuras aportaciones de datos. En esta aportación, hemos estimado una mezcla de ascendencia genética para todos los participantes con el fin de que los investigadores puedan tomar sus propias decisiones sobre si desean o no incluir una gran mezcla genética en sus análisis como parte de sus datos de nivel 2. También hemos incluido una lista de muestras que en estos momentos recomendamos excluir de los análisis porque sus altos niveles de ascendencia mixta reducen la confianza en las predicciones de ascendencia de nuestro modelo actual.
La ascendencia determinada genéticamente de los participantes del GP2 con EP monogénica se estima a través del mismo pipeline que los participantes con EP compleja. La siguiente tabla muestra la ascendencia determinada genéticamente de los participantes con EP monogénica de la quinta aportación del GP2. 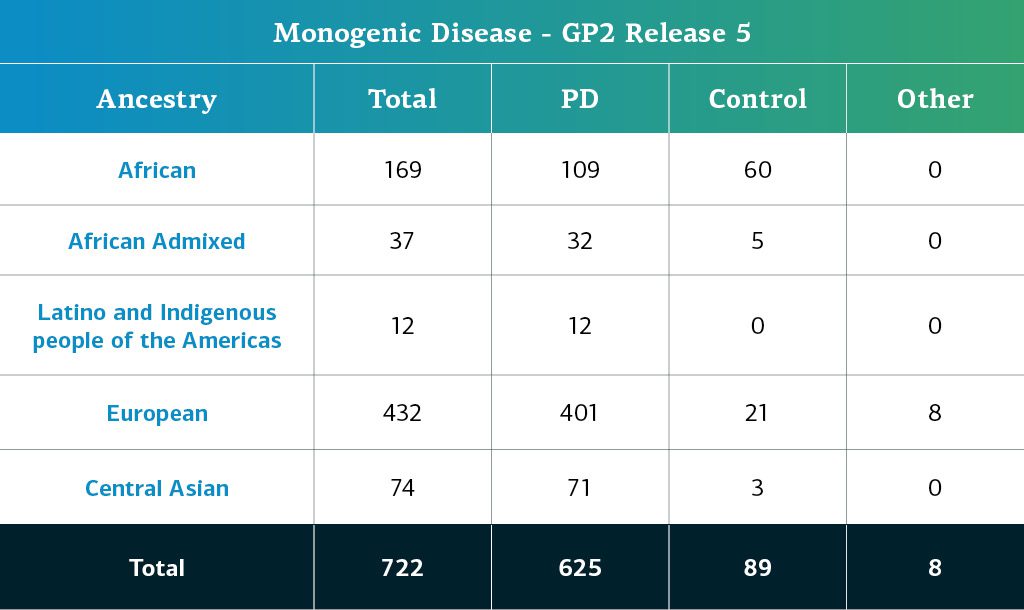 En futuras aportaciones, seguiremos ampliando la diversidad de los participantes. Los invitamos a visitar el para conocer nuestro progreso.
En futuras aportaciones, seguiremos ampliando la diversidad de los participantes. Los invitamos a visitar el para conocer nuestro progreso.
Es importante destacar que en esta aportación introdujimos un pequeño cambio en la forma de determinar qué participantes tienen lazos familiares. Ahora usamos el software KING. Este cambio podía alterar las estimaciones de algunos participantes de aportaciones anteriores. Debido a este cambio, esta aportación irá acompañada de un archivo donde se detallan los baremos para estimar relaciones familiares a fin de que los investigadores puedan personalizar sus propios filtros. Además de los componentes principales proyectados, a partir de ahora también incluimos componentes principales calculados por ascendencia, para llevar a cabo análisis corriente abajo.
Resumen estadístico: Nos complace anunciar que, con esta aportación, también pondremos a disposición de los usuarios con acceso de nivel 1 un nuevo resumen estadístico. Kim et al 2023 también está disponible como parte de nuestra colaboración con el Neurodegenerative Disease Knowledge Portal.
- Nalls et al 2019 sin 23andme en hg38 (el GWAS de EP europeo más grande del mundo)
- Loesch et al 2021 (el GWAS de EP latinoamericano/amerindio más grande del mundo)
- Kim et al 2023 sin 23andme (metanálisis multiancestrales sobre el riesgo de la EP que incluye poblaciones europeas, asiática orientales, latinoamericanas/amerindias y afroamericanas)
- Rizig et al 2023 sin 23andme en hg38 (el GWAS de EP de población africana y africana mixta más grande del mundo, con datos de la aportación 5 del GP2)
Variaciones del número de copias:
También hemos actualizado las llamadas probabilísticas a las variaciones del número de copias (CNV, por sus siglas en inglés) para todas las muestras genotipadas que pasaron el control de calidad (a nivel de gen y en las regiones flanqueantes de 250 kb) y ahora incluyen todas las muestras de la aportación 5. Dichos datos genotipados se agruparon en un archivo clúster con un formato específico del GP2 (disponible en los directorios Utils para usuarios con acceso tanto de nivel 1 como de nivel 2). Tanto el archivo clúster como el flujo (pipeline) usados para predecir las llamadas probabilísticas CNV pueden consultarse en el GP2 Github y pueden usarse con datos externos al GP2. Para más información sobre clustering utilizando el archivo clúster de genotipado específico del GP2 y las llamadas probabilísticas CNV, consulte el artículo de blog del GP2 «Los componentes de la tercera aportación de datos del GP2».
En el artículo de blog «» encontrará más información sobre la estructura de los datos clínicos y genotipados de enfermedades complejas, así como en el documento README, que se ha actualizado para incluir esta nueva aportación de datos y está disponible en los espacios de trabajo oficiales del GP2 de Terra. Los datos de WGS sobre la EP monogénica también se detallan en el mismo documento README.
Como siempre, ¡consulte el README que acompaña cada aportación de datos del GP2 para obtener más detalles sobre pipelines, datos y análisis!
En nombre de los Grupos de Trabajo de Análisis de Datos de la Enfermedad de Parkinson Genéticamente Compleja, Análisis de Datos de la Enfermedad de Parkinson Monogénica y Diseminación de Datos y Código Bioinformático.


