Im Mai 2023 kündigte GP2 die fünfte Datenfreigabe auf der Terra-Plattform in Zusammenarbeit mit AMP® PD an.
Zusätzlich zu den Beständen der früheren Freigaben des Netzwerks Komplexe Krankheit und des Monogenic-Netzwerks umfasst diese Version zusätzlich 7462 neue Teilnehmer*innen mit komplexen Erkrankungen und 487 Teilnehmer*innen mit monogenen Erkrankungen.
- Die Daten zur komplexen Krankheit (Genotypen) umfassen nun insgesamt 24.935genotypisierte Teilnehmer*innen (12.728mit Parkinson-Erkrankung, 10.533 Kontrollpersonen und 1674 „sonstige“ Phänotypen).
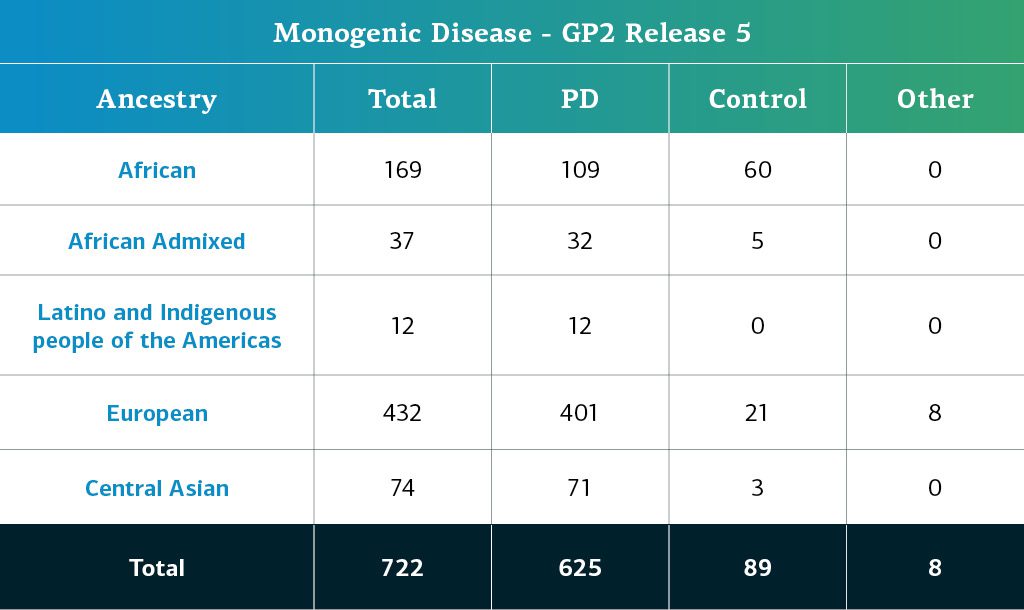
- Die Daten zu monogenen Erkrankungen (Gesamtgenomsequenzen) umfassen nun insgesamt 722 sequenzierte Teilnehmer*innen.
Individuen-spezifische Daten:
Folgende Kohorten sind mit dieser Freigabe zu GP2 hinzugekommen:
- Arizona Brain Bank Brain and Body Donation Program (BBDP), eine in den USA ansässige Gehirnbank
- Innate and Adaptive Immunity in Parkinson Disease (IMMUNEPD), eine US-amerikanische Studie der University of Alabama in Birmingham
- IPDGC Africa (IPDGCAF-NG), eine nigerianische Kohorte
- PDGENEration (PDGNRTN), eine Kohorte der Parkinson’s Foundation
- STELLENBOS Genetic study of Parkinson’s disease and related movement disorders, eine südafrikanische Kohorte der Stellenbosch University
- Malaysian Parkinson’s Genetics Cohort (UMKLM), eine malaysische Kohorte der University of Malaya
- Genotype-Phenotype Correlations in Parkinson’s Disease and Related Movement Disorders (KUL), eine monogene Kohorte der University of Malaysia
- Parkinson Disease and Movement Disorders Center Biorepository (PDMDC), eine monogenetische Kohorte der Northwestern University
Die genetisch determinierte Abstammung von GP2-Teilnehmer*innen mit komplexer Krankheit wird in zehn Abstammungsgruppen gegliedert; die Tabelle unten gibt Aufschluss über die genetisch determinierte Abstammung der Teilnehmer*innen mit komplexer Krankheit in dieser Freigabe, die die Qualitätskontrolle durchlaufen haben und imputiert wurden. Diese Zahlen beinhalten Proben aus früheren Freigaben, die unter Verwendung der neuen Cluster-Datei neu geclustert wurden und die Qualitätskontrolle durchlaufen haben, sowie die neu genotypisierten und gemeinsam genutzten Proben, die nur in dieser aktuellen Freigabe enthalten sind.
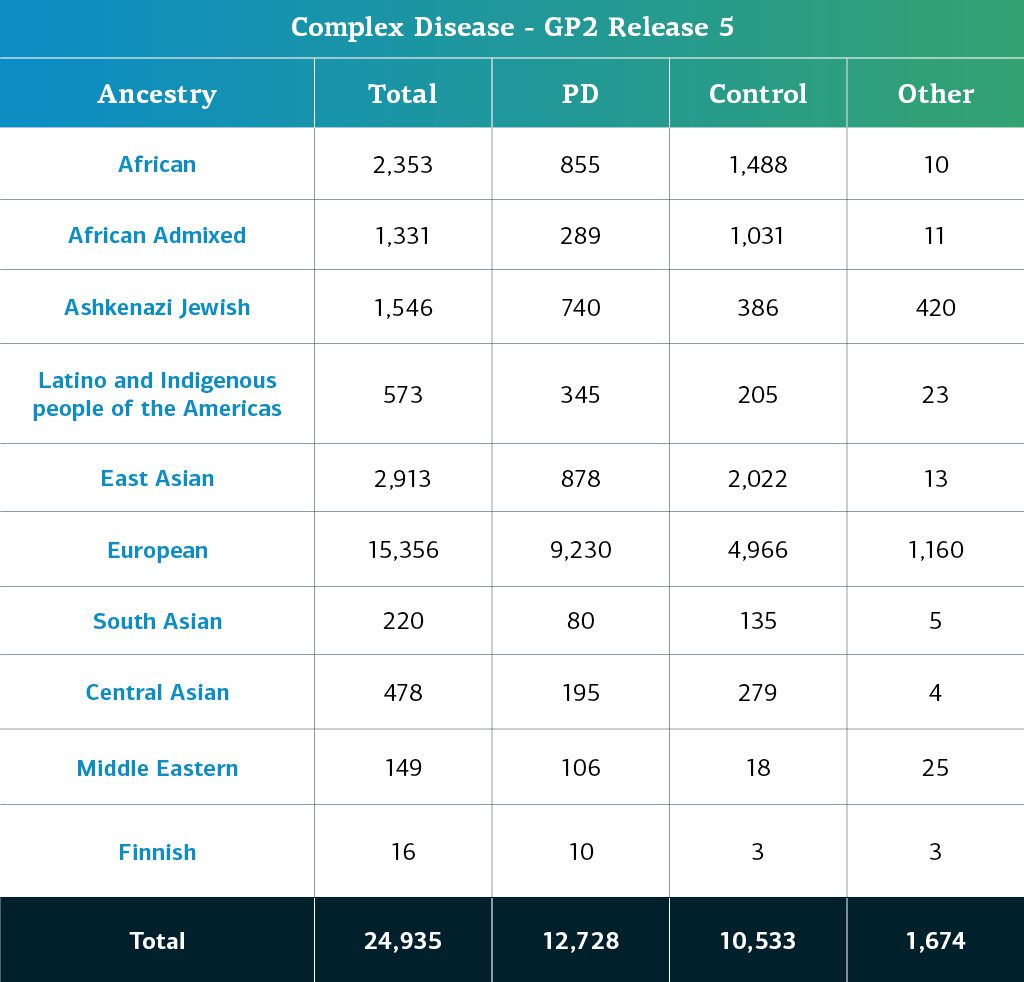
Wichtige Hinweise zu dieser Version : STELLENBOS ist eine südafrikanische Kohorte und weist genetische Beimischungen auf, die diese Region und ihre Abstammungsgeschichte widerspiegeln. Einige unserer Vorhersagen zur Abstammung der Teilnehmer*innen dieser Studie ändern sich womöglich, wenn diese neue Population in zukünftigen Veröffentlichungen berücksichtigt wird. In dieser Freigabe sind für alle Teilnehmer*innen Schätzungen zur Beimischung enthalten, damit die Forscher*innen jeweils entscheiden können, ob und wie eine hohe Beimischung als Teil der Tier-2-Daten in ihre Analyse einbezogen werden soll. Es ist auch eine Liste von Proben enthalten, für die wir aktuell von der Berücksichtigung in der Analyse abraten, da ihr hoher Grad an Beimischung zu einem geringeren Konfidenzniveau bezüglich der abstammungsspezifischen Vorhersagen durch unser aktuelles Modell führt.
Die genetisch bedingte Abstammung der GP2-Teilnehmer*innen mit monogener Erkrankung wird anhand derselben Pipeline geschätzt wie die der Teilnehmer*innen mit komplexer Erkrankung. In der nachstehenden Tabelle ist die genetisch determinierte Abstammung der Teilnehmer*innen mit monogener Erkrankung aus der fünften GP2-Freigabe dargestellt.
Durch zukünftige Datenfreigaben wird sich die Vielfalt der verfügbaren Teilnehmenden weiter erhöhen. Auf unserem können Sie sich über den Fortschritt unserer Arbeiten informieren.
Eine wichtige Neuerung in dieser Freigabe ist eine geringfügige Änderung in der Art und Weise, wie wir feststellen, welche Teilnehmer*innen verwandtschaftlich miteinander verbunden sind. Hierfür nutzen wir nun die KING Software. Mit dieser Änderung wurden möglicherweise die Schätzungen zu den Verwandtschaftsbeziehungen für einige unserer zuvor veröffentlichten Teilnehmer*innen aktualisiert. Diese Änderung umfasst nun eine Datei mit den geschätzten Schwellenwerten zu den Verwandtschaftsbeziehungen, die Forscher*innen für ihre eigenen Zwecke filtern können. Zusätzlich zu den projizierten Hauptkomponenten sind nun auch pro Abstammung berechnete Hauptkomponenten für nachgelagerte Analysen enthalten.
Zusammenfassende Statistik:
Wir freuen uns, dass wir jetzt auch neue zusammenfassende Statistiken über den Tier-1-Zugang für diese Freigabe zur Verfügung stellen können. Kim et al 2023 sind ebenfalls über unsere laufende Zusammenarbeit mit dem Neurodegenerative Disease Knowledge Portal verfügbar.
- Nalls et al 2019 without 23andme in hg38 (größte europäische genomweite Assoziationsstudie (GWAS) zu Parkinson)
- Loesch et al 2021 (größte lateinamerikanische/amerindische GWAS zu Parkinson)
- Kim et al 2023 without 23andme (Multi-Abstammungs-Metaanalyse für das Parkinson-Risiko, umfasst europäische, ostasiatische, lateinamerikanische/amerindische und afroamerikanische Teilnehmer*innen)
- Rizig et al 2023 without 23andme in hg38 (größte Parkinson-GWAS mit afrikanischer Beteiligung und Beimischung; unter Nutzung von Daten der 5. Freigabe von GP2)
Kopienzahlvarianten:
Die Aufrufe der Kopienzahlvarianten (CNV) für alle genotypisierten Proben, welche die Qualitätskontrolle durchlaufen haben (Gen-Ebene plus 250kb flankierende Abschnitte), wurden aktualisiert und alle Proben der 5. Freigabe einbezogen. Diese Daten wurden mit Hilfe einer benutzerdefinierten GP2-Genotyp-Clustering-Datei geclustert (verfügbar in den utils-Verzeichnissen beim Tier-1- wie auch beim Tier-2-Datenzugang). Die Cluster-Datei und die Pipeline für die Vorhersage der probabilistischen CNV-Aufrufe finden sich auf dem GP2 Github zur Nutzung mit Daten außerhalb von GP2. Weitere Informationen zum Clustering mit Hilfe der speziellen GP2-Genotypisierungs-Cluster-Datei und den probabilistischen CNV-Aufrufen finden sich in dem Beitrag „Die Komponenten der dritten Datenfreigabe von GP2“ auf dem GP2 Blog.
Nähere Informationen zur Struktur der Genotyp- und klinischen Daten zur komplexen Krankheit finden sich im Blogbeitrag „Die Komponenten der ersten Datenfreigabe von GP2“ sowie in der README-Datei, die für diese Freigabe aktualisiert wurde und in den offiziellen GP2-Terra-Workspaces verfügbar ist. Die WGS-Daten zur monogenen Parkinson-Erkrankung sind ebenfalls in dieser README-Datei enthalten.
Wie immer finden Sie weitere Details zu Pipelines, Daten und Analysen in der README-Datei zur jeweiligen GP2-Freigabe!
Im Namen der GP2-Arbeitsgruppen Komplexe Krankheit – Datenanalyse, Monogene Datenanalyse sowie Daten- und Code-Verbreitung.









