Im Oktober 2022 kündigte GP2 die dritte Datenfreigabe auf der Terra-Plattform in Zusammenarbeit mit AMP® PD an. Diese Version ist buchstäblich vollgepackt mit neuen Daten und Ressourcen und enthält fast doppelt so viele Proben wie die vorherige Freigabe.
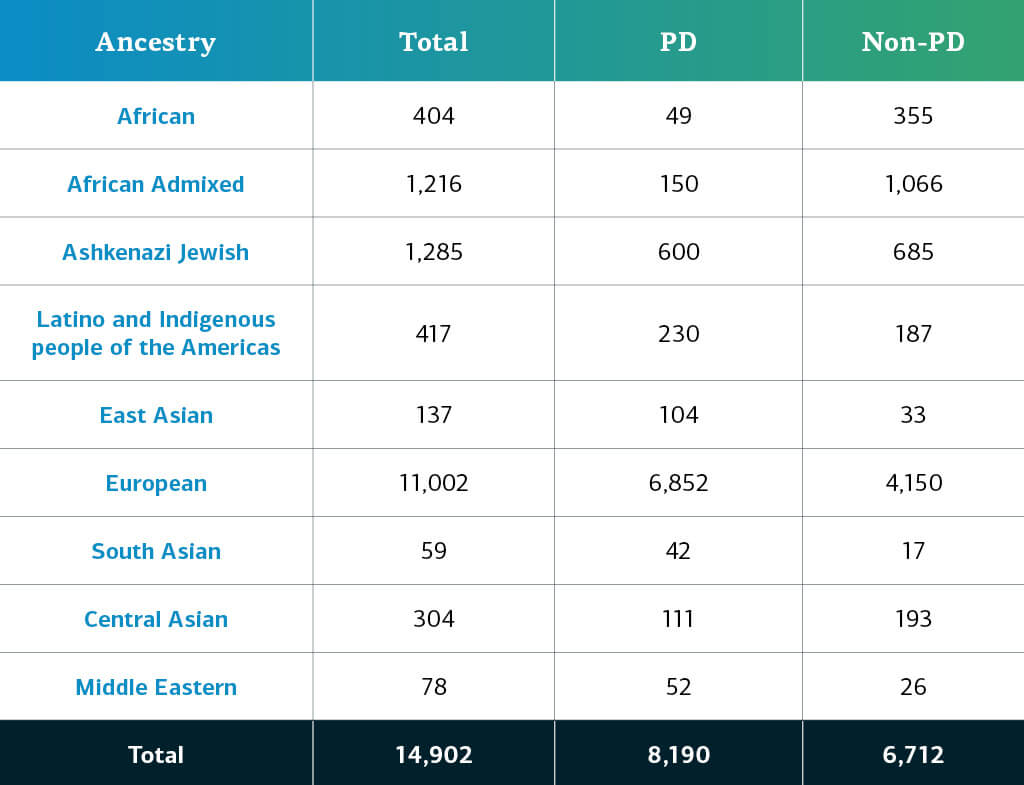
Diese Veröffentlichung umfasst 6.330 zusätzliche neue Teilnehmer zu komplexen Erkrankungen und ergänzt die vorherigen Veröffentlichungen der komplexen und monogenen Netzwerke. Die Daten zur komplexen Krankheit umfassen nun insgesamt 14.902 genotypisierte Teilnehmer*innen (8190 mit Parkinson-Erkrankung, 6712 ohne Parkinson). Neue Kohorten in dieser Freigabe sind:
- Australian Parkinson’s Genetics Study, APGS (Australische Parkinson-Genetik-Studie)
- Fox Investigation for New Discovery of Biomarkers (BioFIND), eine klinische Parkinson-Beobachtungsstudie für die Ermittlung neuer Biomarker
- Teilnehmende Standorte der Black and African American Connections to Parkinson’s Disease (BLAACPD-KPM, BLAACPD-RUSH, BLAACPD-UAB, BLAACPD-UC), einer Parkinson-Kohorte von schwarzen und afroamerikanischen Personen
- GP2-Kohorte der Universität Koc (KOC), eine in der Türkei ansässige Kohorte
- LRRK2 Cohort Consortium (LCC), eine Parkinson-Kohorte aus LRRK2-Mutationsträger*innen
- Movement Disorders Genotypes and Phenotypes – King’s College London (MDGAP-KINGS), eine im Vereinigten Königreich ansässige Gehirnbank mit Schwerpunkt Genotypen und Phänotypen von Bewegungsstörungen
- New Zealand Parkinson’s Programme, NZP3 (Parkinson-Programm Neuseeland)
- Parkinson’s Genes and Environment Study, PAGE (Parkinson-Studie zu Genen und Umwelt)
- Die Parkinson’s Progression Markers Initiative, PPMI (Initiative betreffend Parkinson-Progressionsmarker)
- Systemic Synuclein Sampling Study (S4), eine Parkinson-Biomarker-Studie zu Alpha-Synuclein
Die genetisch determinierte Abstammung von GP2-Teilnehmer*innen mit komplexer Krankheit wird in zehn Abstammungsgruppen gegliedert (die neun Gruppen unten plus eine kleine Anzahl finnischer Europäer*innen); die Tabelle unten gibt Aufschluss über die genetisch determinierte Abstammung der Teilnehmer*innen mit komplexer Krankheit in dieser Freigabe, die die Qualitätskontrolle durchlaufen haben und imputiert wurden. Diese Zahlen beinhalten Proben aus früheren Freigaben, die unter Verwendung der neuen Cluster-Datei neu geclustert wurden und die Qualitätskontrolle durchlaufen haben, sowie die neu genotypisierten und gemeinsam genutzten Proben, die nur in dieser aktuellen Freigabe enthalten sind.
Durch zukünftige Datenfreigaben wird sich die Vielfalt der verfügbaren Teilnehmenden weiter erhöhen. Auf unserem Dashboardkönnen Sie sich über den Fortschritt unserer Arbeiten informieren.
Die primäre Leistung dieses Datensatzes besteht darin, dass er mehr Proben mit array-basierten und imputierten Genotypdaten sowie zusätzliche klinische und genomische Metadaten enthält. Diese Daten wurden mit Hilfe einer benutzerdefinierten GP2-Genotyp-Clustering-Datei geclustert (verfügbar in den utils-Verzeichnissen beim Tier-1- wie auch beim Tier-2-Datenzugang). Diese Clusterdatei basiert auf 2793 Proben aus 6 Abstammungsgruppen, die repräsentativ für die verschiedenen genetischen Abstammungen dieser GP2-Datenfreigabe sind, sowie auf einer Ergänzung um 420 Fälle der Gaucher-Krankheit, um relevante Varianten im GBA-Risikogen besser zu erfassen. Die Clusterdatei kann ebenfalls über das GP2 GitHub Repository heruntergeladen werden, wenn Sie dies auf Daten außerhalb von GP2 anwenden möchten.
Wir haben auch die Aufrufe der Kopienzahlvarianten (CNV) für alle genotypisierten Proben aktualisiert, die die Qualitätskontrolle durchlaufen haben (Gen-Ebene plus 250kb flankierende Abschnitte). Die CNV beschreibt die Variation in der Anzahl der Wiederholungen eines bestimmten DNA-Abschnitts. Diese Variation kann durch Deletion, Insertion oder andere Ereignisse entstanden sein und liefert möglicherweise mehr Informationen darüber, wie strukturelle Variationen das Krankheitsrisiko beeinflussen. Die Pipeline für die probabilistischen CNV-Aufrufe findet sich auf dem GP2-Github. Diese Ergebnisse sind nach genetischer Abstammung im Arbeitsbereich der Stufe 2 stratifiziert. Diese probabilistischen CNVs sind ein hervorragender Ausgangspunkt für die Priorisierung von Proben mit potenziellen Insertionen, Deletionen oder Duplikationen in Genen, die für Folgestudien und weitere Analysen relevant sind.
Nähere Informationen zur Struktur der Genotyp- und klinischen Daten zur komplexen Krankheit finden sich im Blogbeitrag „Die Komponenten der ersten Datenfreigabe von GP2“sowie in der README-Datei, die für diese Freigabe aktualisiert wurde und in den offiziellen GP2-Terra-Workspaces verfügbar ist. Die WGS-Daten zur monogenen Parkinson-Erkrankung sind ebenfalls in dieser README-Datei enthalten. Weitere monogene WGS-Daten werden mit der nächsten Freigabe verfügbar.
Die Vorschau auf die nächste Freigabe umfasst (geplant für die Ferienzeit, Kalenderjahr Q1-2023):
- Mehr Genotypdaten
- Mehr WGS-Daten
- Mehr klinische Metadaten
- Aktualisierte Fassungen von Analysecode und Werkzeugen
- Zusätzliche zusammenfassende Statistiken für GWAS-Studien zu Risiko und Alter bei Einsetzen der Erkrankung
Dieser Blog-Beitrag stammt von Mitgliedern der Arbeitsgruppe Complex Disease – Data Analysis (Komplexe Krankheit – Datenanalyse):: Hampton Leonard, Mike Nalls, Dan Vitale und Mary Makarious; Matthew Korestsky von den National Institutes of Health; Kristin Levine von Data Tecnica International/National Institutes of Health und Zih-Hua Fang und Peter Heutink, Mitglieder der Arbeitsgruppe Monogenic – Data Analysis (Monogene Datenanalyse) .
Schauen Sie sich unsere anderen Datenveröffentlichungen an.




