GP2の第3回データリリースの内容

2022年10月、GP2は、AMP®PDと共同で、Terraプラットフォーム上で第3弾のデータリリースを発表しました。このリリースは、新しいデータとリソースが詰まっており、前回のリリースのほぼ2倍のサンプルが含まれています。
このリリースには、6330人の新たな複合疾患の参加者が追加され、複合・モノジェニックネットワークからのこれまでリリースに追加されました。複合疾患データは現在、合計14,902名の遺伝子型同定参加者(PDが8190人、非PDが6712人)で構成されています。このリリースの新しいコホートは次のとおりです:
- The Australian Parkinson’s Genetics Study (APGS)
- The Fox Investigation for New Discovery of Biomarkers (BioFIND)、PDの観察的臨床研究
- 黒人、アフリカ系アメリカ人のPDコホートである、The Black and African American Connections to Parkinson’s Disease (BLAACPD-KPM、BLAACPD-RUSH、BLAACPD-UAB、BLAACPD-UC)からの参加
- Koc University GP2 Cohort (KOC)、トルコ拠点のコホート
- The LRRK2 Cohort Consortium (LCC)、LRRK2突然変異のキャリアのPDコホート
- 英国拠点のブレインバンクであるMovement Disorders Genotypes and Phenotypes – King’s College London (MDGAP-KINGS)
- New Zealand Parkinson’s Programme (NZP3)
- Parkinson’s Genes and Environment Study (PAGE)
- The Parkinson’s Progression Markers Initiative (PPMI)
- アルファシヌクレインのPDバイオマーカー研究であるSystemic Synuclein Sampling Study (S4)
複合疾患GP2参加者の遺伝的祖先は、10の祖先グループ(以下の9つのグループと少数のフィンランドヨーロッパ人)に分けられています。以下の表は、このリリースにおける複合疾患参加者の遺伝的祖先を詳細に示し、品質管理に合格し、帰属されたものです。これらの番号には、新しいクラスターファイルを使用して再クラスター化され、現在のリリースに固有の新しくジェノタイピングおよび共有されたサンプルとともに品質管理を行った、以前のリリースサンプルが含まれています。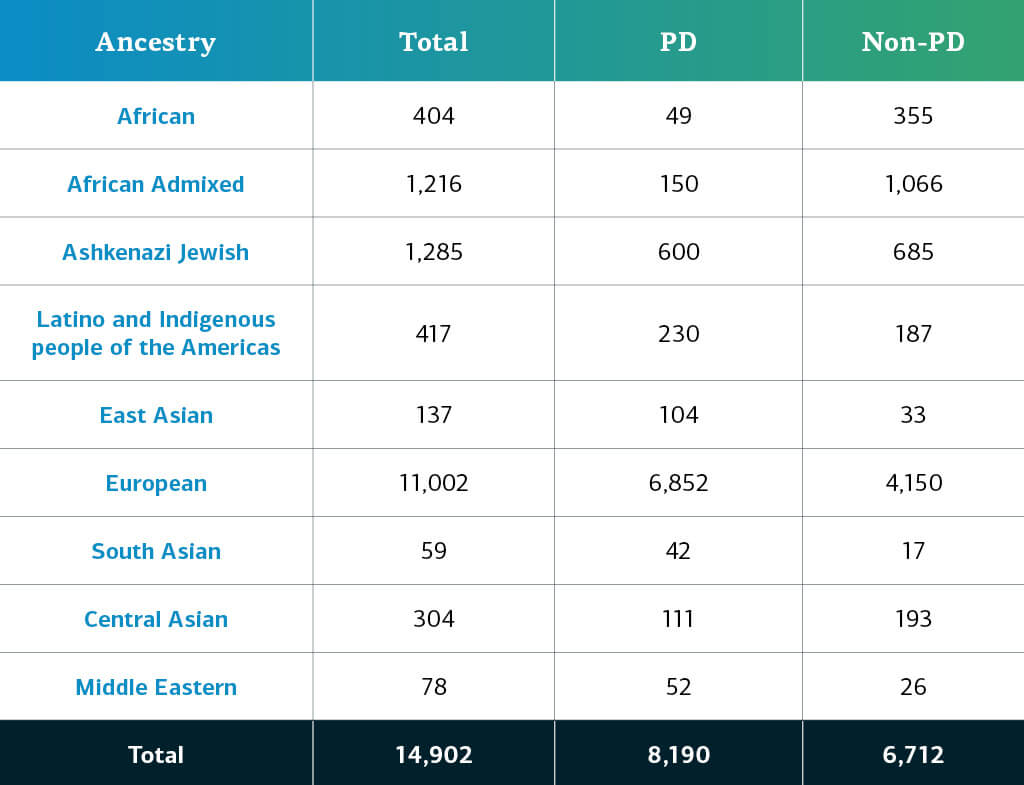 今後のデータリリースで、利用可能な参加者の多様性をさらに拡大します。ダッシュボードで進捗状況を確認していただけます。
今後のデータリリースで、利用可能な参加者の多様性をさらに拡大します。ダッシュボードで進捗状況を確認していただけます。
このデータセットの主な成果は、アレイに基づく帰属された遺伝子型データに加え、臨床的・ゲノムのメタデータが追加された、より多くのサンプルを含むことです。データは、カスタムGP2ジェノタイプクラスタリングファイルを使用してクラスタ化されています(階層1と階層2の両方のデータアクセスの下のutils ディレクトリで使用できます)。このクラスターファイルは、GP2データリリースを構成する多様な遺伝的祖先を代表する6つの祖先グループにまたがる2793のサンプルと、GBAリスク遺伝子にかかわる変異体をより良く捕捉するための420のGaucher病症例に基づいています。GP2以外のデータに適用する場合は、GP2 GitHub リポジトリからクラスタファイルをダウンロードすることもできます。
また、品質管理(遺伝子レベルプラス250kb隣接領域)に合格する全ての遺伝子型タイピングサンプルのためのコピー数変異体(CNV)コールを更新しました。CNVとは、あるDNAの伸長が繰り返される回数の変化のことです。この変異は、欠失、挿入、または他の事象によって生じた可能性があり、構造的変異が疾患リスクにどのように影響するかについてより多くの情報を提供する可能性があります。確率的CNVコールを生成するために使用されるパイプラインは、GP2 Githubにあります。 結果は階層2アクセスワークスペースの遺伝的祖先によって層別化されます。確率論的CNVは、追跡研究およびさらなる解析に関わる遺伝子における潜在的な挿入、欠失、重複を有するサンプルに優先順位を付けるための大きな出発点となります。
複合疾患の遺伝子型と臨床データの構造に関するより詳細な情報は、このリリースで更新され、公式GP2テラワークスペースで入手可能なREADMEと同様、「GP2の最初のデータリリースの構成要素」ブログ記事で詳述されています。モノジェニックPD WGSデータも同じREADMEに詳述されています。追加のモノジェニックWGSデータは、次のリリースで利用可能になります。
次回リリースのプレビュー(年末年始ターゲット、2023年Q1カレンダー):
- 遺伝型データ
- WGSデータ
- 臨床メタデータ
- アップデートされた分析コードとツール
- GWAS研究のリスクと発症年齢の追加概要統計
このブログは、複合疾患 – データ分析ワーキンググループのメンバーによって執筆されました:Hampton Leonard、Mike Nalls、Dan VitaleとMary Makarious;Matthew Korestsky the National Institutes of Health;Kristin Levine Data Tecnica International/National Institutes of Health;Zih-Hua FangとPeter Heutink、モノジェニック – データ分析ワーキンググループのメンバー。



