En octubre del 2022, el GP2 anunció la tercera aportación de datos a la plataforma Terra en colaboración con AMP® PD. Esta nueva aportación contiene nuevos datos y recursos e incluye casi el doble de muestras que la aportación anterior.
En total, los nuevos datos corresponden a 6,330 participantes nuevos con enfermedades complejas, que se suman a las aportaciones anteriores de las Redes la Enfermedad de Parkinson de Etiología Compleja y Monogénica. Los datos de enfermedades complejas corresponden ahora a un total de 14,902 participantes genotipados (8,190 con EP; 6,712 sin EP). Las nuevas cohortes de esta aportación son:
- El Estudio Australiano de Genética del Parkinson (APGS, por sus siglas en inglés)
- La Investigación Fox del Nuevo Descubrimiento de Biomarcadores (BioFIND), un estudio clínico observacional de la EP
- Datos de centros adheridos a The Black and African American Connections to Parkinson’s Disease (BLAACPD-KPM, BLAACPD-RUSH, BLAACPD-UAB, BLAACPD-UC), una cohorte de pacientes negros y afroamericanos con EP
- La cohorte del GP2 de la Universidad Koc (KOC), de procedencia turca
- El LRRK2 Cohort Consortium (LCC), una cohorte de pacientes con EP portadores de la mutación del gen LRRK2
- Genotipos y Fenotipos de Trastornos del Movimiento – King’s College London (MDGAP-KINGS), un banco de cerebros del Reino Unido
- New Zealand Parkinson’s Programme (NZP3)
- El estudio PAGE (Parkinson’s Genes and Environment)
- La Parkinson’s Progression Markers Initiative (PPMI)
- El estudio de muestreo de la sinucleína sistémica (S4), un estudio sobre biomarcadores de la EP enfocado en la alfa-sinucleína
La ascendencia determinada genéticamente de los participantes del GP2 con enfermedades complejas se divide en diez grupos (los nueve grupos que se detallan a continuación más una pequeña población de europeos finlandeses). La tabla siguiente presenta la ascendencia determinada genéticamente de los participantes con enfermedades complejas de esta aportación de datos que pasaron el control de calidad y se imputaron. Estas cifras incluyen muestras de datos anteriores que ahora se reagruparon en un nuevo archivo clúster y que pasaron el control de calidad, junto con las nuevas muestras genotipadas y compartidas por primera vez en la aportación actual. 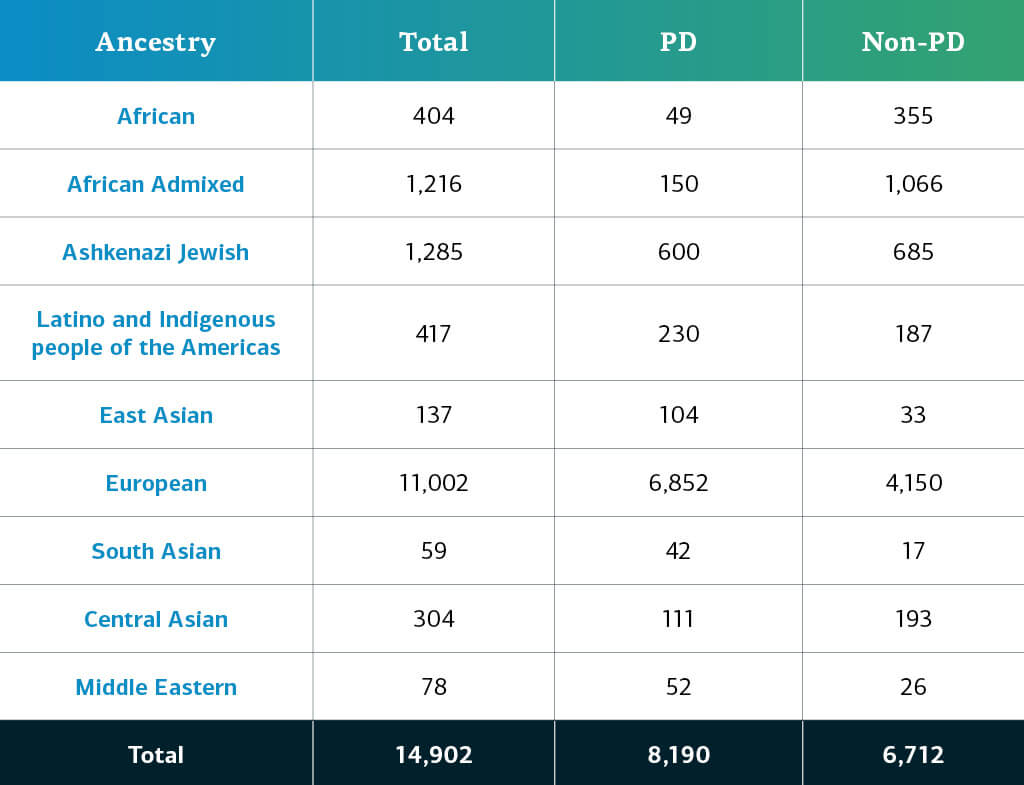 Seguiremos publicando nuevas aportaciones de datos para contribuir a la diversidad de los participantes. Los invitamos a visitar este panel para conocer nuestro progreso.
Seguiremos publicando nuevas aportaciones de datos para contribuir a la diversidad de los participantes. Los invitamos a visitar este panel para conocer nuestro progreso.
El objetivo principal de este conjunto de datos consistió en incluir más muestras con datos genotipados e imputados mediante matrices, además de metadatos clínicos y genómicos adicionales. Dichos datos genotipados se agruparon en un archivo clúster con un formato específico del GP2 (disponible en los directorios Utils para usuarios con acceso tanto de nivel 1 como de nivel 2). Este archivo clúster se basa en 2,793 muestras de 6 grupos de ascendencia representativos de las diversas ascendencias genéticas que comprenden esta aportación de datos del GP2, además de 420 casos de enfermedad de Gaucher que se incluyeron para reflejar mejor las variantes de interés del gen de riesgo GBA. El archivo clúster también se puede descargar desde el repositorio GitHub del GP2 , en caso de que desee aplicar los datos en contextos externos al GP2.
También hemos actualizado las llamadas probabilísticas a las variaciones del número de copias (CNV, por sus siglas en inglés) para todas las muestras genotipadas que pasaron el control de calidad (a nivel de gen y en las regiones flanqueantes de 250 kb). Las CNV hacen referencia a las variaciones del número de veces que se repite un tramo determinado de ADN. Dichas variaciones pueden deberse a supresiones, inserciones u otros factores, y pueden proporcionar información acerca de cómo la variación estructural afecta al riesgo de enfermedad. Encontrará una explicación sobre el pipeline empleado para producir las llamadas probabilísticas de CNV en el Github del GP2. Los resultados están estratificados por ascendencias genéticas en el espacio de trabajo de acceso de nivel 2. Estas CNV probabilísticas son un excelente punto de partida para priorizar las muestras portadoras de posibles inserciones, supresiones o duplicaciones de genes de interés para estudios de seguimiento y análisis posteriores.
En el artículo de blog «Los componentes de la primera aportación de datos del GP2» encontrará más información sobre la estructura de los datos clínicos y genotipados de enfermedades complejas, así como en el documento README, que se ha actualizado para incluir esta nueva aportación de datos y está disponible en los espacios de trabajo oficiales del GP2 de Terra. Los datos de WGS sobre la EP monogénica también se detallan en el mismo documento README. La próxima aportación de datos incluirá más datos de WGS monogénicos.
La próxima aportación de datos (prevista para el primer trimestre del 2023), incluirá:
- Más datos genotipados
- Más datos de WGS
- Más metadatos clínicos
- Código y herramientas de análisis actualizados
- Estadísticas de resumen adicionales basadas en estudios GWAS de riesgo y edad de aparición de la enfermedad
Este artículo de blog está redactado por los siguientes miembros del Grupo de Trabajo de Análisis de Datos de la Enfermedad de Parkinson Genéticamente Compleja : Hampton Leonard, Mike Nalls, Dan Vitale y Mary Makarious; Matthew Korestsky, de los Institutos Nacionales de Salud; Kristin Levine, de Data Tecnica International/Institutos Nacionales de Salud; y Zih-Hua Fang y Peter Heutink, miembros del Grupo de Trabajo de Análisis de Datos Monogénicos.




