En mai 2023, le GP2 a annoncé la diffusion de la cinquième édition de données sur la plateforme Terra en collaboration avec AMP® PD.
Cette édition recense 7 462 nouveaux participants dans la catégorie des maladies complexes et 487 dans celle des maladies monogéniques qui viennent s’ajouter au données des éditions précédentes issues du réseau des maladies complexes.
- Les données sur les maladies complexes comptent désormais un total de 24,935 participants génotypés (12 728 atteints de MP, 10 533 cas témoins et 1 674 « autres » phénotypes).
- Les données sur les maladies monogéniques portent maintenant sur un total de 722 participants dont le séquençage de génome complet a été effectué.
Données individuelles:
Les nouvelles cohortes ajoutées au GP2 dans cette diffusion sont:
- La « Arizona Brain Bank » et le programme « Brain and body donation program » (BBDP) sont des banques de cerveaux basées aux États-Unis
- L’étude IMMUNEPD (Immunité innée et adaptative dans la maladie de Parkinson) est menée aux États-Unis par l’Université d’Alabama à Birmingham
- {IPDGC Afrique (IPDGCAF-NG), une cohorte du Nigéria
- PDGENEration (PDGNRTN), une cohorte de la La Parkinson’s Foundation
- Étude génétique de la maladie de Parkinson et des troubles connexes du mouvement (STELLENBOS), une cohorte sud-africaine de l’Université de Stellenbosch
- La cohorte malaisienne de la maladie génétique de Parkinson (UMKLM), une cohorte malaisienne de l’Université de Malaya.
- Corrélations génotype-phénotype dans la maladie de Parkinson et les troubles connexes du mouvement (KUL), une cohorte monogénique de l’Université de Malaisie
- « Parkinson Disease and Movement Disorders Center Biorepository » (PDMDC), une cohorte monogénique de l’Université Northwestern
Les participants au GP2 atteints de maladies complexes ont leur ascendance génétiquement déterminée et répartie en dix groupes ; le tableau ci-dessous détaille l’ascendance des participants à cette édition qui ont passé le contrôle qualité et ont été comptabilisés. Ces chiffres prennent en compte les données des éditions précédentes réorganisées selon une nouvelle méthode de cluster et soumises à un contrôle qualité, ainsi que de nouveaux échantillons génotypés et spécifiques à cette édition.
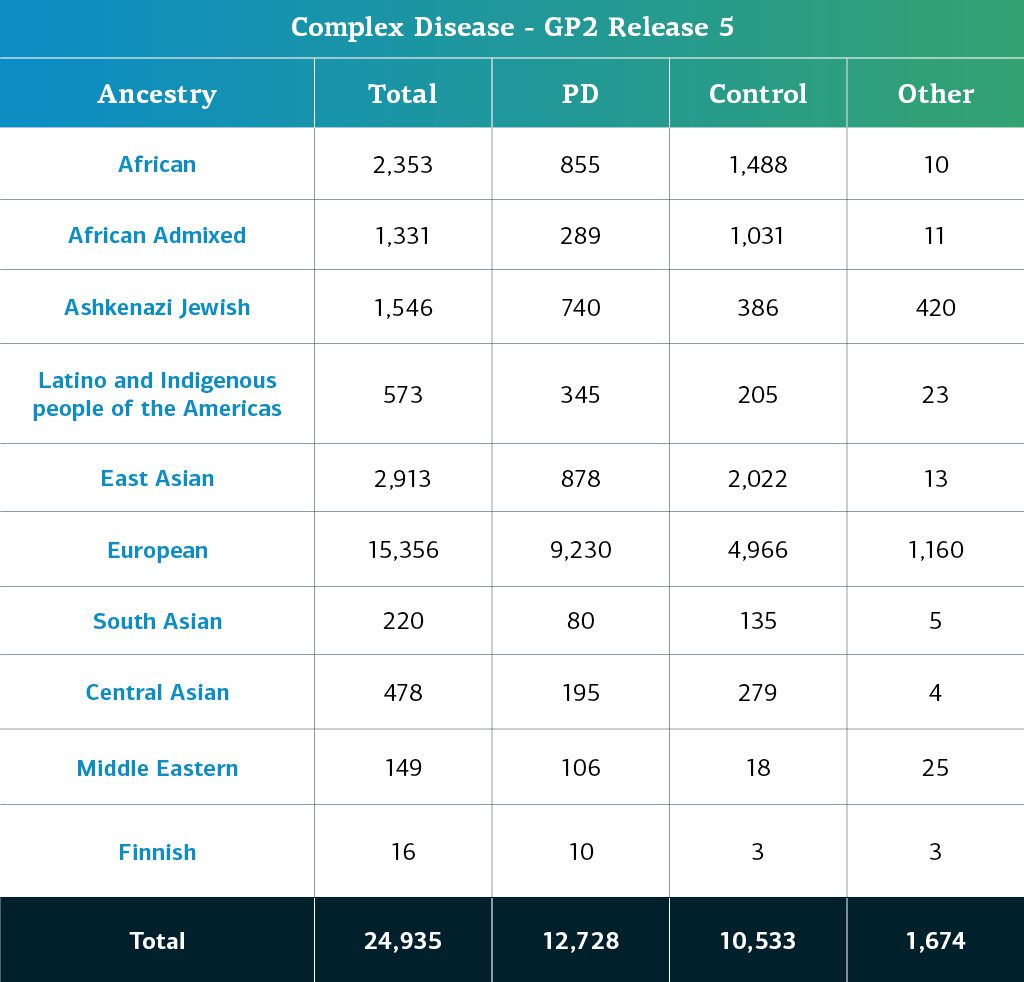
Points important de cette édition : STELLENBOS est une cohorte sud-africaine qui présente des brassages d’ascendance génétique reflétant cette région et son histoire. Certaines prédictions d’ascendance concernant les membres de cette étude pourront évoluer lorsque cette nouvelle population sera prise en compte dans les futures éditions. Dans cette édition nous fournissons des estimations de brassage pour tous les participants afin que les chercheurs puissent choisir si et comment ils doivent inclure un taux de brassage élevé dans leur analyse des données du niveau 2. Nous publions également la liste des échantillons que nous recommandons d’éliminer à ce stade de l’analyse car leurs niveaux de brassage élevés conduisent à un indice de confiance plus faible dans les prédictions d’ascendance de notre modèle actuel.
L’ascendance génétiquement déterminée des participants au GP2 sur les maladies monogéniques est estimée à partir des mêmes données que l’ascendance des participants sur les maladies complexes. Le tableau ci-dessous détaille l’ascendance génétiquement déterminée des participants aux maladies monogéniques dans la cinquième édition du GP2.
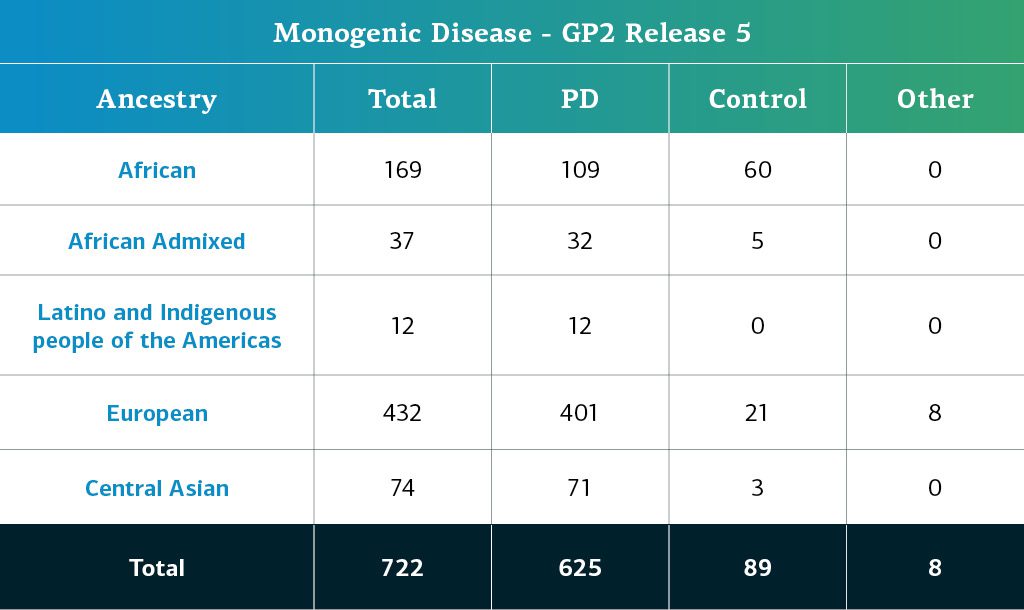
Les futures diffusions de données contribueront à renforcer la diversité du groupe des participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès. Dans cette nouvelle édition, une mise à jour importante concerne une modification à la marge de la méthode utilisée pour déterminer les liens de parenté des participants. Nous utilisons maintenant le logiciel KING. Ce changement peut avoir affecté des estimations de la parenté précédemment diffusées pour certains de nos participants. À la suite de cette modification un fichier détaillant les degrés de parenté estimés sera désormais mis à la disposition des chercheurs lors de la diffusion afin qu’ils puissent l’utiliser pour leur propre filtrage. En plus des composantes principales estimées, nous calculons maintenant les composantes principales par ascendance et les mettons à disposition pour des analyses en aval.
Synthèse statistique :
Nous sommes fiers d’annoncer que de nouvelles synthèses statistiques sont également disponibles en accédant au premier tiers de données de cette édition. Kim « et coll. » 2023 est également disponible grâce à notre partenariat en cours, en accédant le portailNeurodegenerative Disease Knowledge Portal.
- Nalls « et coll. » 2019 without 23andme in hg38 (la plus grande étude d’association pangénomique européenne sur la maladie de Parkinson) (PD GWAS).
- Loesch « et coll. » 2021 (la plus grande étude d’association pangénomique latino-américaine/amérindienne sur la maladie de Parkinson)
- Kim « et coll. » 2023 without 23andme (méta-analyse de multi-ascendance sur les risques de la maladie de Parkinson incluant les Européens, les Asiatiques de l’Est, les Latino-Américains/Amérindiens et les Afro-Américains)
- Rizig « et coll. » 2023 sans 23andme dans hg38 (la plus grande étude d’association pangénomique africaine et africaine mixte ; utilise les données de la cinquième édition du GP2)
Variation du nombre de copies :
La variation du nombre de copies (CNV) pour tous les échantillons génotypés ayant passé le contrôle de qualité (au niveau du gène plus 250kb de séquençage des régions flanquantes) a été mise à jour pour inclure tous les échantillons dans la cinquième édition du GP2. Ces données ont été regroupées par génotype dans un fichier de classement propre au GP2 (disponible dans les paramètres des répertoires sous accès aux données de niveau 1 et 2). Le fichier de classement et la procédure utilisée pour les appels probabilistes de la variation du nombre de copies sont disponibles sur le Github du GP2 pour l’utilisation des données hors du GP2. Pour plus d’informations concernant le regroupement par génotype dans le fichier de classement propre au GP2 et les appels probabilistes de la variation du nombre de copies, veuillez consulter « Les éléments de la troisième publication de données du GP2 » sur le blog de GP2. Des informations complémentaires sur la structure du génotype des maladies complexes et sur les données cliniques sont détaillées sur le blog « Les éléments de la première publication des données de GP2 » ainsi que dans le README qui a été mis à jour pour cette nouvelle édition et qui est disponible sur les espaces de travail officiels de GP2 Terra. Les données monogéniques sur la maladie de Parkinson-séquençage de génome complet (PD WGS) sont également disponibles dans ce même document README.
{Comme toujours, veuillez-vous référer au README qui accompagne chaque nouvelle édition du GP2 pour plus de détails concernant les procédures, les données et les analyses!
Au nom des groupes de travail du GP2 sur l’analyse des données relatives aux maladies complexes, sur l’analyse des données relatives aux maladies monogéniques et sur la diffusion des données et des codes.








