Resumen
En septiembre de 2024, el GP2 anunció su octava publicación de datos en las plataformas Workbench de Terra y Verily® en colaboración con AMP® PD.<//span> Esta aportación cuenta con 5,481 secuencias de genoma completo adicionales y 10,454 secuencias clínicas de exoma. Se incluirán genotipados adicionales de cara a la siguiente publicación.
- Los datos de secuencias de genoma completo (WGS) corresponden ahora a un total de 7,734 participantes secuenciados (6,113 con EP, 617 de control y 1,004 «otros» fenotipos).
- Si restamos las muestras localmente restringidas, el total asciende a 4,713 participantes (4,098 con EP; 390 de control y 225 «otros» fenotipos).
- Cabe destacar que los casos reclutados por la Red monogénica están clasificados como «otros»
- Asimismo, esta aportación de WGS es una publicación parcial de secuencias de genoma completo de dos cohortes de AMP® PD (BioFind y PPMI) denominadas, conjuntamente, «GP2 WGS». Las muestras publicadas están asociadas a sus ID originales en AMP® PD a través de un archivo de referencias cruzadas que se incluye en esta aportación de datos.
- Esta edición también incluye 10,454 participantes de llamado conjunto de secuenciación clínica de exoma facilitados por la Parkinson’s Foundation.
- Esta publicación incluye un total de 62,087 personas de las cuales disponemos de datos clínicos principales. De entre ellas, 16,800 cuentan con datos clínicos fenotipados profundos y datos genéticos.
¿Qué hay de nuevo en esta aportación de datos?
- Nuevas muestras de secuenciación del genoma completo del GP2 y conjuntos de variantes de llamado conjunto, incluidas muestras de dos cohortes de AMP® PD (BioFind y PPMI)
- Datos clínicos de exoma de la Parkinson’s Foundation
- Nuevos datos clínicos; el número total de participantes de quienes disponemos de datos clínicos principales ahora asciende a 62,087
Introducción de muestras localmente restringidas vía Verily Viewpoint Workbench
Seguimos haciendo pruebas para brindar acceso a muestras localmente restringidas, es decir, muestras sujetas al Reglamento General de Protección de Datos (RGPD), mediante nuestra colaboración con Verily Viewpoint Workbench. Por ahora, y mientras el GP2 sigue desarrollando soluciones para la compartición de datos protegidos por el RGPD, los datos de la octava aportación con restricciones regionales solo están disponibles para miembros y socios del consorcio del GP2. A medida que las pruebas y la implementación sigan avanzando en 2024, esta solución estará también disponible para la comunidad de investigación en general. Todas las muestras de la octava aportación pueden encontrarse en Workbench, mientras que todas las muestras de la octava aportación no sujetas a los requisitos del RGPD pueden encontrarse en el Workbench comunitario de Terra (como en las publicaciones anteriores). Para obtener acceso a la aportación completa en VWB, usted debe:
- Contar con acceso de nivel 2 autorizado por el GP2
- Llenar el formulario de solicitud para muestras sujetas al RGPD
- Ser miembro del consorcio del GP2 (contribuir cohortes, ser socio del GP2 o ser miembro de un equipo de análisis de proyectos)
Datos clínicos
Esta aportación contiene datos clínicos de un total de 62,087 participantes de quienes disponemos de datos genéticos y datos clínicos principales. Esta edición incluye datos clínicos fenotipados profundos y datos genéticos de 16,800 participantes. Esta información contiene los siguientes datos:
- Edad en el momento del diagnóstico y de la aparición de síntomas
- Diagnóstico principal, actual y más reciente
- Exámenes cognitivos, como el Miniexamen Cognoscitivo y la Evaluación Cognitiva de Montreal
- Revisión patrocinada por la Movement Disorder Society de la Escala de Valoración Unificada de la Enfermedad de Parkinson
- «Otros» fenotipos detallados, como la demencia de cuerpos de Lewy
- Los casos reclutados por la Red monogénica están clasificados como «otros»
Secuenciación clínica de exoma
Esta aportación cuenta con la secuenciación clínica de exoma de 10,454 muestras facilitadas por Parkinson’s Foundation, con análisis de las regiones de codificación y empalmes de ARN de 4,717 genes. Esta secuenciación dirigida tiene el propósito de identificar e informar acerca de variantes que podrían tener relevancia clínica, con un enfoque en aquellas que se alinean con la información clínica del paciente y sus antecedentes familiares. Para obtener más información, consulte la página de Fulgent Genetics Clinical Exome.
Secuencias de genoma completo detectadas por DeepVariant-GLnexus
Usamos el pipeline DeepVariant de Google conjuntamente con GLnexus para las llamadas de variante a nivel de cohorte. DeepVariant es un programa de deep learning de llamado de variantes con un mayor rendimiento que las herramientas de vanguardia existentes a la hora de llamar variantes genéticas a nivel individual. También simplifica el proceso, mejora la precisión y aumenta la confiabilidad. La ascendencia determinada genéticamente de los participantes genotipados del GP2 se divide en once grupos. La tabla siguiente presenta la ascendencia determinada genéticamente de los participantes genotipados de esta aportación de datos que pasaron el control de calidad y se imputaron. Estas cifras incluyen muestras de datos anteriores que ahora se reagruparon en un nuevo archivo clúster y que pasaron el control de calidad, junto con las nuevas muestras genotipadas y compartidas por primera vez en la aportación actual. 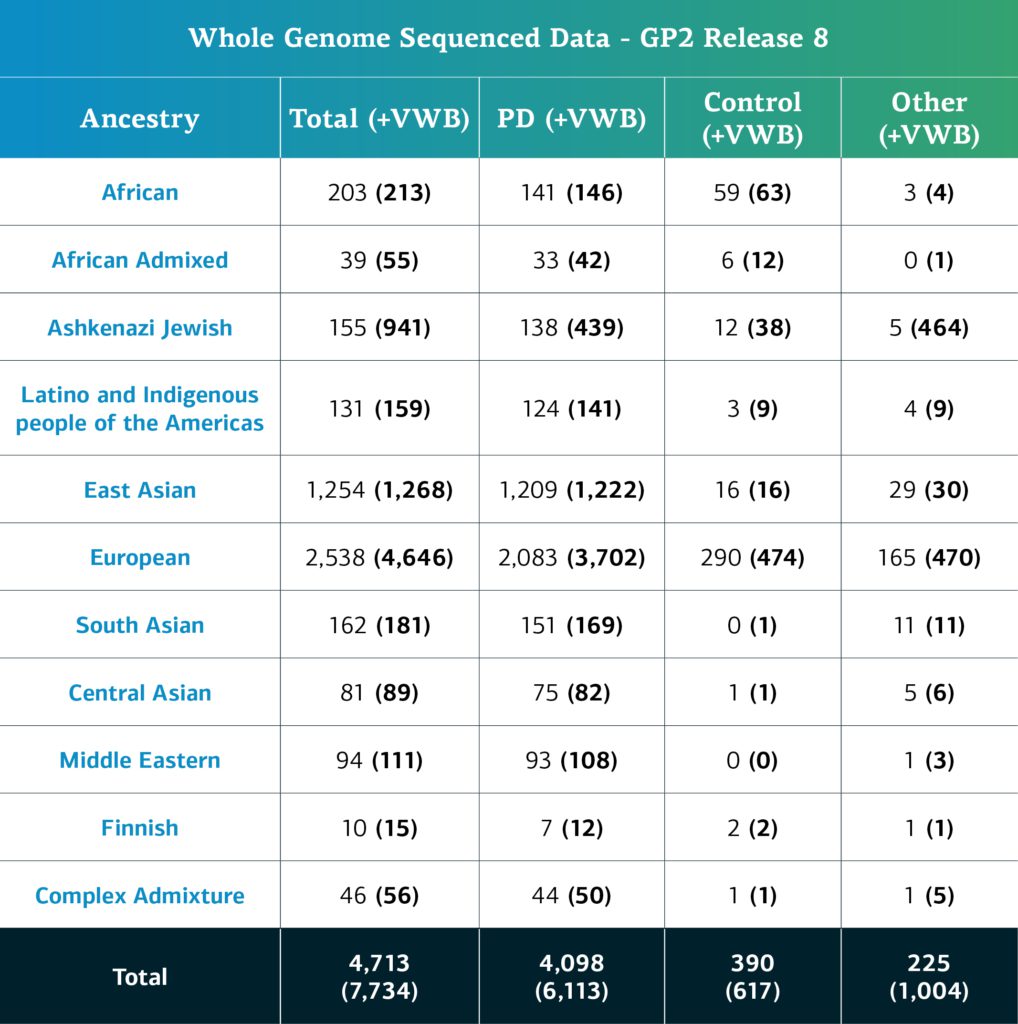 En futuras aportaciones, seguiremos ampliando la diversidad de los participantes. Los invitamos a visitar nuestro Panel de Cohortes para conocer nuestro progreso. Los usuarios que ya tengan acceso de nivel 2 pueden explorar los datos en más profundidad en nuestro Navegador de Cohortes, que presentamos en un artículo de blog anterior.
En futuras aportaciones, seguiremos ampliando la diversidad de los participantes. Los invitamos a visitar nuestro Panel de Cohortes para conocer nuestro progreso. Los usuarios que ya tengan acceso de nivel 2 pueden explorar los datos en más profundidad en nuestro Navegador de Cohortes, que presentamos en un artículo de blog anterior.
Como siempre, ¡consulte el README que acompaña cada aportación de datos del GP2 para obtener más detalles sobre recomendaciones de control de calidad, pipelines, datos y análisis!






