Aperçu général
En septembre 2024, le GP2 a annoncé la publication de la huitième édition des données sur les plateformes Terra et Verily® Workbench , en collaboration avec l’AMP® PD. Cette édition comprend 5 481 séquençages du génome entier et 10 454 séquençages d’exomes cliniques. La prochaine édition proposera d’autres génotypages.
- Les données du séquençage du génome entier comprennent désormais un total de 7 734 participants séquencés (6 113 atteints de Parkinson, 617 cas témoins et 1004 « autres » phénotypes).
- Si l’on soustrait les échantillons restreints, le nombre total atteint 4 713 participants (soit 4 098 cas de Parkinson, 390 cas témoins et 225 « autres » phénotypes).
- Il convient de noter que les cas recrutés par l’intermédiaire du réseau monogénique sont classés dans la catégorie « Autres »
- Par ailleurs, cette édition du WGS comprend une publication partielle des séquences du génome entier de deux cohortes de l’AMP® PD (BioFind et PPMI), lesquelles ont été regroupées avec celles du WGS du GP2 à des fins d’analyse. Au moyen d’un fichier de concordance d’identifiants inclus dans cette édition, il est possible de faire le lien entre les échantillons publiés et leurs identifiants de départ issus de l’AMP® PD.
- Dans cette édition, le séquençage de l’exome clinique de 10 454 participants, issus de la Fondation pour la maladie de Parkinson, a été réalisé en cohortes regroupées.
- Cette édition comprend les données cliniques de base de 62 087 personnes. Sur ce total, l’accès aux données de phénotype clinique profond et génétiques ets disponible pour 16 800 personnes.
Quoi de neuf dans cette publication?
- Nouveaux échantillons de séquençage du génome entier du GP2 et ensembles de variants identifiés en cohortes, y compris les échantillons de deux cohortes d’AMP®PD (BioFind et PPMI).
- Données cliniques sur l’exome de la Fondation pour la maladie de Parkinson
- Données cliniques supplémentaires concernant les individus, portant le total à 62 087 individus pour lesquels des données cliniques de base sont disponibles.
Echantillons restreints localement par le RGPD via Verily Viewpoint Workbench
Nous continuons à essayer, grâce à notre collaboration avec Verily Viewpoint Workbench, d’obtenir l’accès aux échantillons restreints localement, connus également comme étant des échantillons soumis au Règlement général sur la protection des données (RGPD). A ce stade, alors que le GP2 continue de déployer ses solutions de partage de données pour les données protégées par le RGPD, la huitième édition ne sera disponible que pour les membres du consortium GP2 et ses partenaires. Au fur et à mesure de la poursuite des tests et de la mise en œuvre, en 2024, ces solutions seront mises à la disposition d’un plus grand nombre de chercheurs. Tous les échantillons de la huitième édition sont disponibles sur Workbench. Cependant, les échantillons de la huitième édition non régis par le RGPD sont disponibles sur Terra (comme pour les éditions passées). Pour avoir accès à l’intégralité des publications sur VWB, vous devez :
- Disposer d’une autorisation d’accès GP2 de catégorie 2.
- Remplir le formulaire de demande d’échantillons régis par le RGPD.
- Être membre du consortium du GP2 (contribution de cohorte, partenaire GP2 ou équipe de projet d’analyses).
Données cliniques
Cette édition contient les données cliniques de 62 087 personnes dont les données génétiques et les données cliniques de base sont accessibles. Cette édition comprend les données précises du phénotype et des données génétiques de 16 800 personnes. Ces données incluent :
- Âge au moment du diagnostique
- Diagnostique primaire, actuel et suivant
- Examens cognitifs tels que le Mini-Mental State Examination et le Montreal Cognitive Assessment
- Révision de l’échelle UPDRS parrainée par la Movement Disorder Society
- Détail des « autres » phénotypes, tels que la démence à corps de Lewy
- Cas recrutés par l’intermédiaire du réseau monogénique classés dans la catégorie « Autres »
Séquençages d’exomes cliniques
Le séquençage de l’exome clinique, fourni par la Fondation pour la maladie de Parkinson, est disponible pour 10 454 échantillons dans cette édition et offre ainsi une analyse des régions codantes et des sites d’épissage de 4 717 gènes. Ce séquençage ciblé a pour but d’identifier et de signaler les variants susceptibles de présenter une importance clinique, en se concentrant sur ceux qui correspondent aux informations cliniques et aux antécédents familiaux du patient. Pour de plus amples informations, consultez la page Exome clinique de Fulgent Genetics..
Séquençage entier du génome appelé DeepVariant-GLnexus
L’outil DeepVariant de Google associé au GLnexus a été utilisé pour l’identification des variants en cohortes. DeepVariant est un outil d’appel des variants à apprentissage profond qui dépasse les outils les plus sophistiqués actuels en faisant un appel de variant génétique ciblé. Cela simplifie également le processus, tout en en renforçant la précision et la fiabilité. Les participants au GP2 atteints de maladies complexes ont leur ascendance génétiquement déterminée et répartie en onze groupes ; le tableau ci-dessous détaille l’ascendance des participants à cette édition qui ont passé le contrôle de qualité et ont été comptabilisés. Ces chiffres inclus les échantillons des éditions précédentes qui ont été réorganisés selon une nouvelle méthode de classement des dossiers et ont été soumis à un contrôle qualité, ainsi que de nouveaux échantillons de génotypes, spécifiques à cette édition. 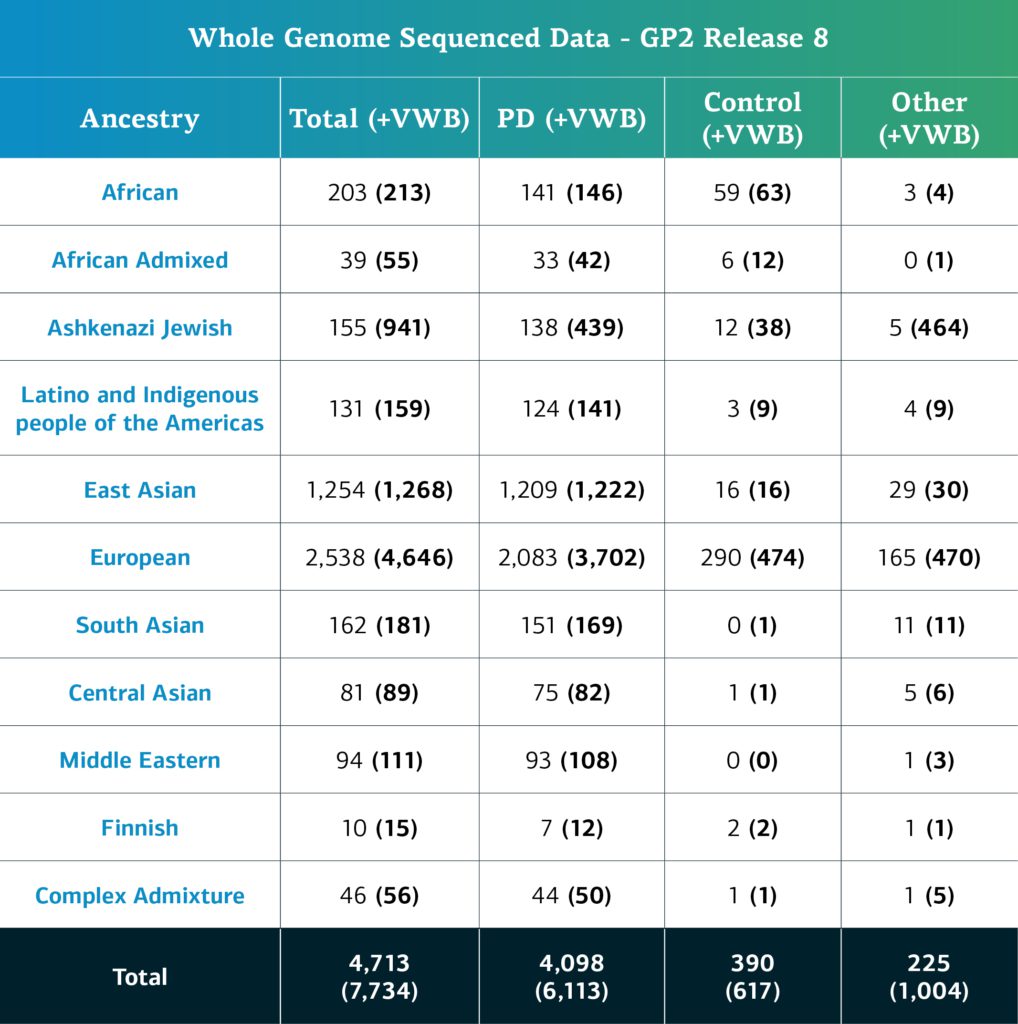 Les futures diffusions de données contribueront à renforcer la diversité du groupe des participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès. Les utilisateurs ayant un accès de catégorie 2 peuvent d’ores et déjà explorer les données sur notre navigateur de cohortes, dont nous avons parlé dans un précédent blog.
Les futures diffusions de données contribueront à renforcer la diversité du groupe des participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès. Les utilisateurs ayant un accès de catégorie 2 peuvent d’ores et déjà explorer les données sur notre navigateur de cohortes, dont nous avons parlé dans un précédent blog.
Comme toujours, veuillez consulter le document README qui accompagne chaque édition du GP2 pour obtenir plus d’informations sur le contrôle de qualité, les canaux, les données et les analyses !










