Übersicht
Im September 2024 kündigte GP2 in Kooperation mit AMP® PD die achte Datenfreigabe auf den Plattformen Terra und Verily® Workbench an. Diese Version enthält 5.481 zusätzliche Gesamtgenomsequenzen (WGS) und 10.454 klinische Exomsequenzen. Weitere Genotypisierungen werden mit der nächsten Freigabe zur Verfügung gestellt.
- Die WGS-Daten umfassen nun insgesamt 7.734 sequenzierte Teilnehmende (6.113 mit Parkinson-Erkrankung, 617 Kontrollpersonen und 1.004 „sonstige“ Phänotypen).
- Abzüglich der Proben mit lokalen Einschränkungen sind nun 4.713 Teilnehmende umfasst (4.098 mit Parkinson-Erkrankung, 390 Kontrollpersonen und 225 „sonstige“ Phänotypen).
- Anmerkung: Über das Monogenic Network rekrutierte Fälle werden als „sonstige“ kodiert.
- Zusätzlich ist in dieser WGS-Freigabe eine Teilfreigabe von WGS aus zwei AMP® Parkinson-Kohorten (BioFind und PPMI) enthalten, die per Joint-Calling mit GP2 WGS umgesetzt wurden. Freigegebene Proben können zu den ursprünglichen AMP® PD-IDs über eine ID-Crosswalk-Datei rückverknüpft werden, die in der Freigabe enthalten ist.
- Diese Freigabe enthält auch 10.454 klinische Exomsequenzierungsdaten (die einem Joint-Calling unterzogen wurden) von Teilnehmenden der Parkinson’s Foundation.
- Diese Freigabe umfasst insgesamt 62.087 Personen, für die klinische Kerndaten verfügbar sind. Hiervon stehen für 16.800 Personen tiefe klinische Phänotypisierungs- und genetische Daten zur Verfügung
Was ist in dieser Freigabe neu?
- Zusätzliche GP2-Ganzgenomsequenzierungsproben und die Joint-Calling-Variantensätze, einschließlich der Proben aus zwei AMP® Parkinson-Kohorten (BioFind und PPMI)
- Klinische Exom-Daten von der Parkinson’s Foundation
- Zusätzliche klinische Individuendaten, wodurch sich die Gesamtzahl der Personen, für die klinische Kerndaten verfügbar sind, auf 62.087 erhöht
DSGVO-Proben mit lokalen Einschränkungen über die Verily Viewpoint Workbench
Im Rahmen unserer Zusammenarbeit mit der Verily Viewpoint Workbench setzen wir das Pilotprojekt zur Gewährung eines Zugangs zu lokal eingeschränkten Proben (d. h. Proben, die unter die Allgemeine Datenschutzgrundverordnung, DSGVO, fallen) fort.
Während GP2 weiter an Lösungen für den Austausch DSGVO-geschützter Daten arbeitet, sind Daten der 8. Freigabe mit regionalen Einschränkungen nur für Mitglieder des GP2-Forschungsverbunds sowie Partner verfügbar. Im Zuge der weiteren Test- und Implementierungsphase im Jahr 2024 wird diese Lösung dann der breiteren Forschungsgemeinschaft zur Verfügung gestellt. Sämtliche Proben der 8. Freigabe sind auf der Workbench zu finden. Sämtliche Proben der 8. Freigabe, die nicht den DSGVO-Anforderungen unterliegen, sind auf der Community Workbench auf Terra zu finden (so wie alle vorherigen Freigaben). Wenn Sie Zugang zur gesamten Freigabe auf VWB erhalten möchten, müssen Sie:
- über einen bewilligten GP2-Tier-2-Zugang verfügen,
- das Antragsformular für DSGVO-Proben ausfüllen,
- Mitglied des GP2-Forschungsverbunds sein (beitragende Kohorte, GP2-Partner oder Projektanalyse-Teammitglied).
Klinische Daten
Diese Freigabe enthält klinische Daten von insgesamt 62.087 Personen, für die genetische und klinische Kerndaten verfügbar sind. Die Freigabe umfasst tiefe klinische Phänotypisierungs- und genetische Daten für weitere 16.800 Personen. Diese Informationen umfassen:
- Alter bei Diagnose und Ausbruch
- Primäre, aktuelle und letzte Diagnose
- Kognitive Tests wie den Mini-Mental-Status-Test und den Montreal-Cognitive-Assessment-Test
- Von der Movement Disorder Society gesponserte Überarbeitung der Verlaufsbeobachtungsskala „Unified Parkinson’s Disease Rating Scale“
- Detaillierte „sonstige“ Phänotypen wie etwa Lewy-Körper-Demenz
- Über das Monogenic Network rekrutierte Fälle werden als „sonstige“ kodiert.
Klinische Exomsequenzen
Die von der Parkinson’s Foundation zur Verfügung gestellten klinischen Exomsequenzierungen sind in dieser Freigabe für 10.454 Proben der Parkinson’s Foundation verfügbar und bieten Analysen zu codierenden Regionen und Spleißverbindungen von 4.717 Genen. Mit dieser gezielten Sequenzierung sollen potenziell klinisch relevante Varianten identifiziert und gemeldet werden, wobei der Schwerpunkt auf den Varianten liegt, die zu den jeweiligen klinischen Patientendaten und der Familiengeschichte passen. Ausführlichere Informationen finden Sie auf der Seite Fulgent Genetics Clinical Exome.
Gesamtgenomsequenzen mit Aufruf durch DeepVariant-GLnexus
Wir verwenden Googles DeepVariant-Pipeline gekoppelt mit GLnexus für die Variantenaufrufe auf Kohortenebene. DeepVariant ist ein Deep-Learning-basiertes Programm für das Varianten-Calling, das den bisherigen Tools im Stand der Technik überlegen ist, da es bei genetischen Varianten auf individueller Ebene sehr akkurate Aufrufe leistet. Zudem vereinfacht es den Prozess bei gleichzeitiger Verbesserung von Präzision und Zuverlässigkeit.
Die genetisch determinierte Abstammung von Array-genotypisierten GP2-Teilnehmenden wird in 11 Abstammungsgruppen gegliedert; die Tabelle unten gibt Aufschluss über die genetisch determinierte Abstammung der genotypisierten Teilnehmenden in dieser Freigabe, welche die Qualitätskontrolle durchlaufen haben und imputiert wurden. Diese Zahlen beinhalten Proben aus früheren Freigaben, die unter Verwendung der neuen Cluster-Datei neu geclustert wurden und die Qualitätskontrolle durchlaufen haben, sowie die neu genotypisierten und gemeinsam genutzten Proben, die nur in dieser aktuellen Freigabe enthalten sind.
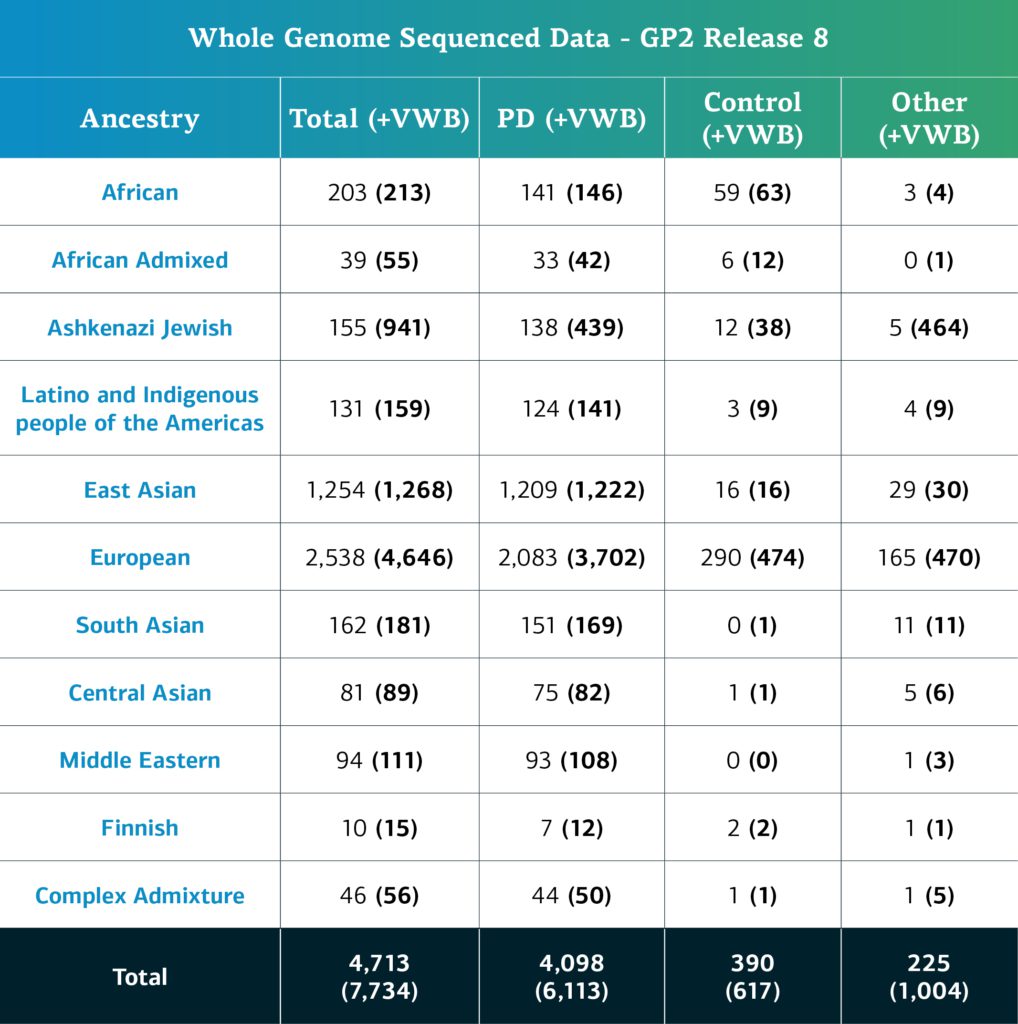
Durch zukünftige Datenfreigaben wird sich die Vielfalt der verfügbaren Teilnehmenden weiter erhöhen. Sie können sich unseren Fortschritt auf unserem Kohorten-Dashboard ansehen . Benutzer mit Tier-2-Zugriff können die Daten in unserem Cohort-Browser weiter untersuchen. , ausführlicher in einem früheren Blog-Beitrag .
Wie immer finden Sie weitere Details zu den Themen Qualitätskontrolle, Pipelines, Daten und Analysen in der README-Datei zur jeweiligen GP2-Freigabe!









