2021年12月、GP2はAMP® PDプラットフォームで初のGP2データリリースを発表しました。このリリースで利用可能になるデータは、CORIELL、ベイラー医科大学(BCM)、BCM-メリーランド大学(UMD)のコホートから4,908人の参加者(3,434 PD、1,474非PD)によって構成されています。遺伝的に決定されたGP2参加者の祖先は、8つの祖先グループに分類されます。以下の表はGP2リリース1の参加者の遺伝的に決定された祖先の内訳を示しています。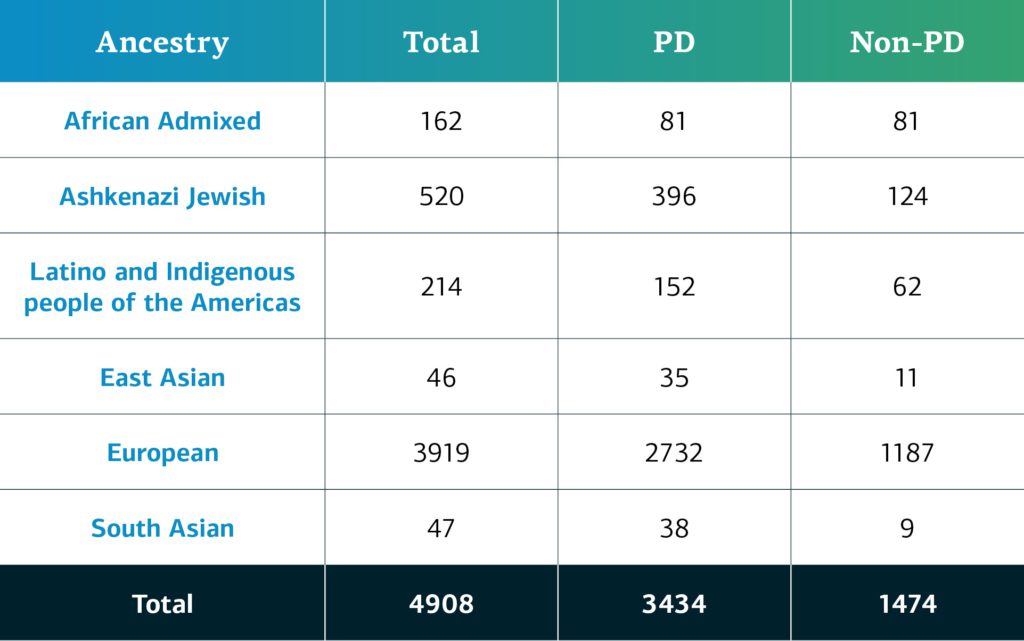 今後のデータリリースで、利用可能な参加者の多様性をさらに拡大します。ダッシュボードで進捗状況を確認していただけます。
今後のデータリリースで、利用可能な参加者の多様性をさらに拡大します。ダッシュボードで進捗状況を確認していただけます。
遺伝子型データ
すべてのデータの遺伝子型はイルミナ・ニューロブースターアレイで特定されました。このアレイは多様な人口集団の神経変性疾患変異用に特別に設計されました。データは標準イルミナプロトコルに基づいて処理され、品質管理はオープンソースのQCパイプラインを使ってGP2が行います。その後データは個別の祖先グループ内でTOPMedを使って入力されます。既知の重複はすべて削除されますが、コミュニティ分析の柔軟性を高めるために関連サンプルは残します。QCパイプラインに含まれるものの詳細については、関連のGitHubページで詳説のワークフローをお読みください。
臨床データ
臨床データを取り入れ、統一されたGP2臨床データセットを構築しました。臨床データの最低要件には人口統計、採用カテゴリー、家族歴が含まれます。GP2コアデータ要件レベルの詳細については、 リソースセクションウェブサイトにある「臨床データ コアデータセット」の資料を参照してください。データはTerraプラットフォームからアクセスできます。 GP2データにアクセスする方法については、ブログ「AMP® PDプラットフォームのGP2データアクセスの申請について」をお読みください。次のデータ製品はGP2リリース1のTerraで利用可能です:
- Tier 1 データアクセス
- 最新のパーキンソン病GWAS(Nalls et al. 2019の23andMeサンプルを除く)からの要約統計
- Tier 2 データアクセス
- 未処理の遺伝子型:インピュテーション前に品質管理に合格したすべてのサンプルの各祖先グループのPLINK2 .pgenファイル
- インピュテーション遺伝子型:TOPMed参照パネルを使ってインピュテーションしたPLINK2.pgenファイル(データリリースに付随するreadmeファイルに詳述されている最低QCフィルタリングに合格)
- メタデータ:QC出力と祖先予測に関する情報を含む
- 臨床データ:臨床データフィールドおよび関連臨床データ辞書
この私たちはこれがパーキンソン病研究コミュニティにとって貴重なリソースになることを願っています。継続的なプロセスの改善を通して、今後のさらなる進展にご期待下さい。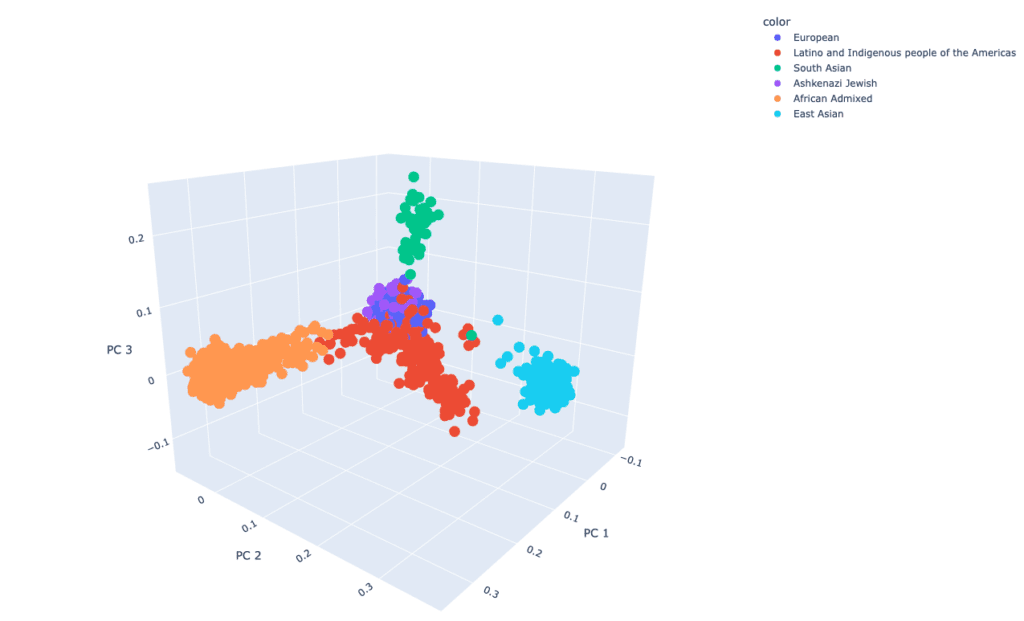 このブログはHampton Leonard、Mike Nalls、Yeajin Song、Dan Vitaleが共同で執筆しました。執筆者の経歴については、GP2複合疾患 データ分析ワーキンググループのページをご覧ください。
このブログはHampton Leonard、Mike Nalls、Yeajin Song、Dan Vitaleが共同で執筆しました。執筆者の経歴については、GP2複合疾患 データ分析ワーキンググループのページをご覧ください。