Resumen
En diciembre de 2023, el GP2 anunció su sexta publicación de datos en las plataformas Workbench de Terra y Verily® en colaboración con AMP® PD. Esta publicación incluye >20,000 participantes nuevos, que se suman a las aportaciones anteriores de las Redes de la Enfermedad de Parkinson de Etiología Compleja y Monogénica.
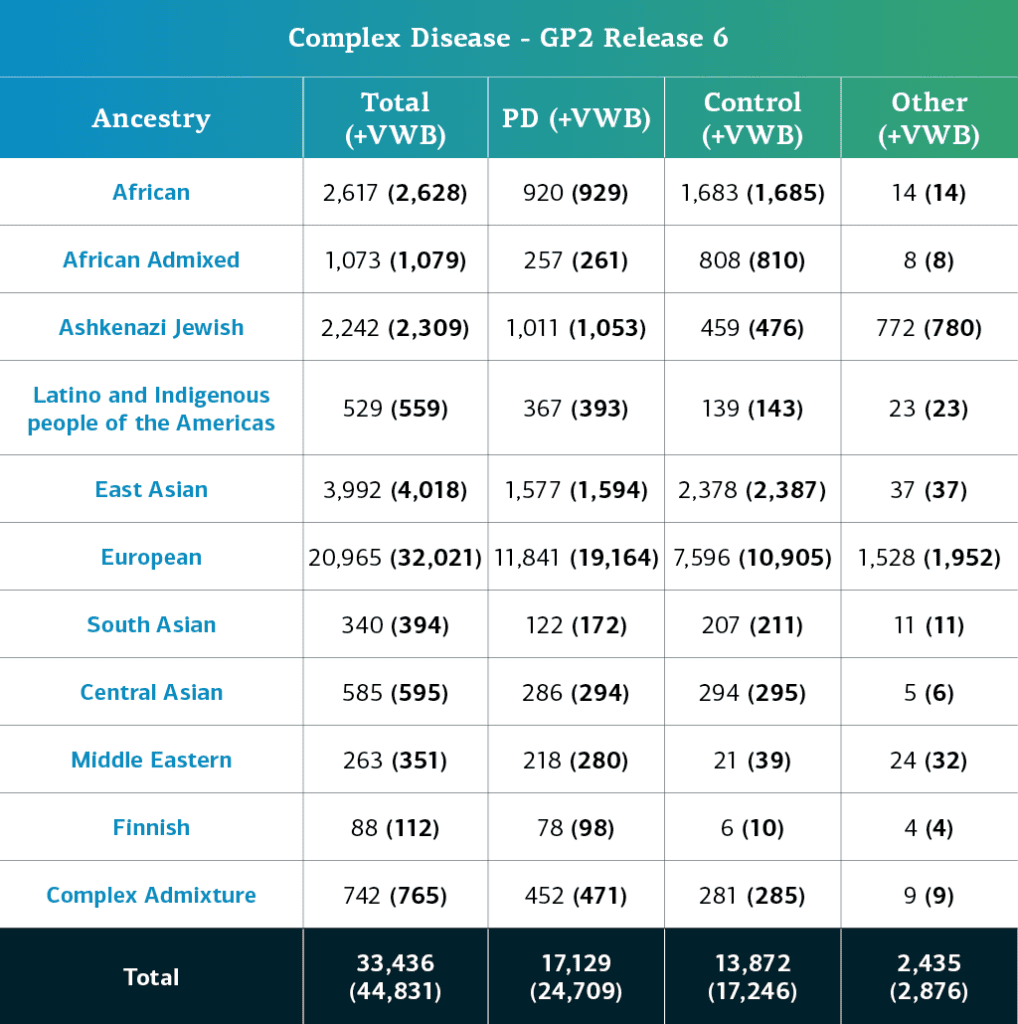
- Los datos de EP compleja (genotipos), incluidas muestras localmente restringidas, corresponden ahora a un total de 44,831 participantes genotipados (24,709 con EP; 17,246 de control y 2,876 «otros» fenotipos)
- Si restamos las muestras localmente restringidas, el total asciende a 33,436 participantes (17,129 con EP; 13,872 de control y 2,435 «otros» fenotipos)
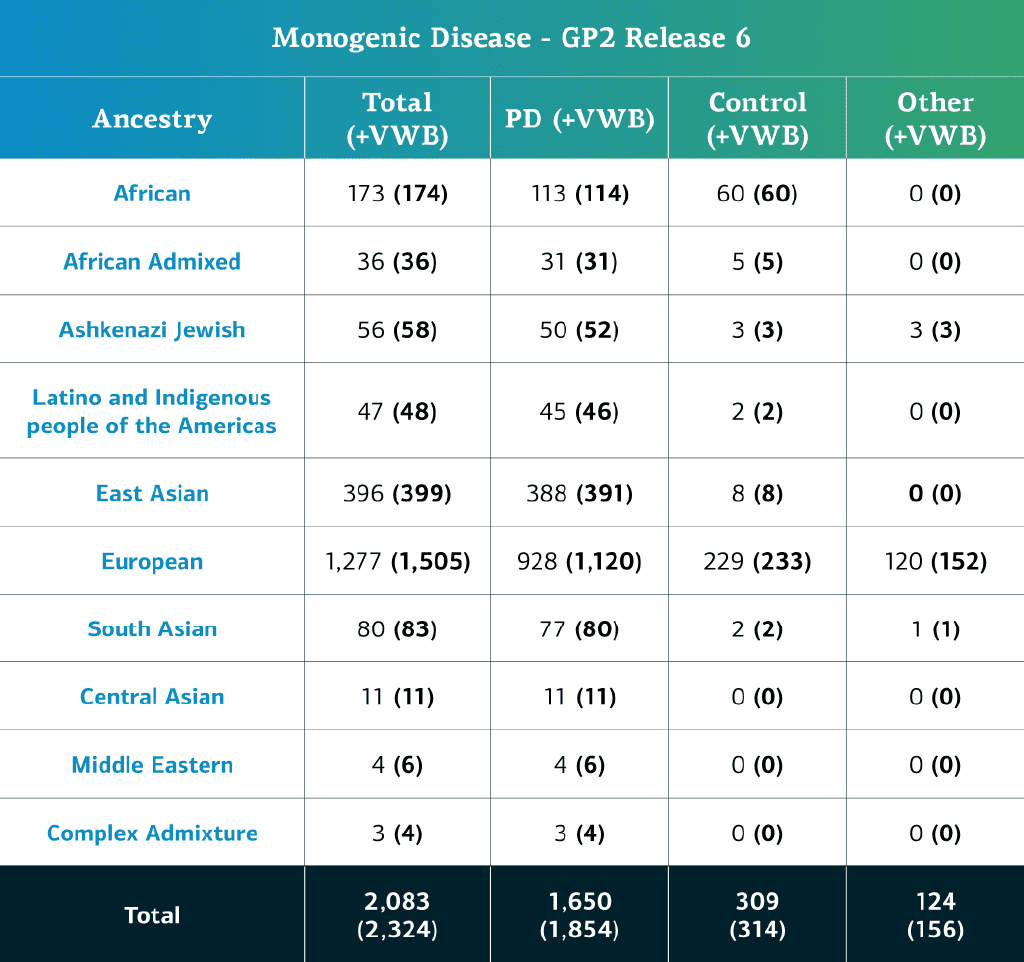
- Los datos de EP monogénica (secuencias de genoma completo) corresponden ahora a un total de 2,324 participantes secuenciados (1,854 con EP, 314 de control y 156 «otros» fenotipos)
- Si restamos las muestras localmente restringidas, el total asciende a 2,083 participantes (1,650 con EP; 309 de control y 124 «otros» fenotipos)
- 12,585 participantes que cuentan con información fenotipada clínica profunda también tienen información genética correspondiente
¿Qué hay de nuevo en esta aportación de datos?
- Nuevas muestras de enfermedad de Parkinson de etiología compleja (genotipadas) y monogénica (genoma completo)
- Introducción de muestras localmente restringidas sujetas al RGPD, vía Verily Viewpoint Workbench
- Introducción de datos clínicos sobre ~12,000 participantes
- Introducción de un nuevo grupo de ascendencia → Ascendencia mixta compleja (Complex Admixture History, CAH)
- Mejoras en las medidas de control de calidad para datos genotipados publicados
- Novedades en la llamada de variantes, ahora con DeepVariant, para los datos de genoma completo publicados
Mejoras de control de calidad
En resumen, GenoTools (v1.0.0) lleva a cabo los siguientes pasos de control de calidad: discrepancias de sexo, recorte de la tasa de éxito, comprobar que no haya duplicados, verificar y notificar si hay familiares y comprobar la tasa de heterocigosis.
A diferencia de las publicaciones anteriores, ya no aplicamos los siguientes filtros a nivel de variante: por frecuencia de alelos menores (MAF), Hardy-Weinberg (HWE) o recuento de alelos menores (MAC). Si desea filtrar los resultados como hacíamos en aportaciones de datos anteriores, le recomendamos que consulte el README correspondiente para obtener información detallada y sugerencias de umbrales.
Ascendencia mixta compleja (CAH)
CAH, o ascendencia mixta compleja, es un nuevo grupo de ascendencia introducido en la sexta aportación de datos del GP2. Se creó para dar respuesta a un gran número de muestras de Sudáfrica y otras poblaciones altamente mixtas que en la quinta aportación de datos se predijeron erróneamente como ascendencia CAS (Asia Central). En la sexta aportación, el grupo de ascendencia CAH contiene principalmente muestras de Stellenbosch University (Ciudad del Cabo, Sudáfrica), The Coriell Institute (Camden, Nueva Jersey, Estados Unidos) y Parkinson’s Foundation (Miami, Florida, Estados Unidos). Consideramos que las muestras categorizadas como CAH son demasiado mixtas para incluirlas en análisis con otros grupos de ascendencia del GP2.
Muestras localmente restringidas sujetas al RGPD, vía Verily Viewpoint Workbench
Nos complace anunciar que algunos usuarios podrán acceder a muestras localmente restringidas, es decir, muestras sujetas al Reglamento General de Protección de Datos (RGPD), gracias a nuestra colaboración con Verily Viewpoint Workbench. Workbench es un entorno seguro para la gestión y el análisis de datos biomédicos, cuyo objetivo es impulsar la colaboración en la investigación y la reproducción de datos mediante la integración en la nube. Es compatible con espacios de trabajo compartidos, como Python y código R, y ofrece una suite de servicios nativos en la nube de gestión y análisis de datos. Workbench facilita la integración del uso seguro de datos y la autenticación personalizada, por lo que es un espacio de investigación ideal, seguro y escalable donde el GP2 puede albergar sus muestras localmente restringidas.
Por ahora, y mientras el GP2 sigue desarrollando soluciones para la compartición de datos protegidos por el RGPD, la sexta aportación solo está disponible para miembros y asociados del consorcio del GP2. De cara a principios de 2024 y a medida que las pruebas y la implementación sigan avanzando, estas soluciones estarán también disponibles para la comunidad de investigación en general. Todas las muestras de la sexta aportación pueden encontrarse en Workbench, mientras que todas las muestras de la sexta aportación no sujetas a los requisitos del RGPD pueden encontrarse en el Workbench comunitario de Terra (como en las publicaciones anteriores). Para obtener acceso a la publicación completa en Verily Viewpoint Workbench necesitará:
- Contar con acceso de nivel 2 autorizado por el GP2
- Ser miembro del consorcio del GP2 (contribuir cohortes, ser socio del GP2 o ser miembro de un equipo de análisis de proyectos)
- Llenar el formulario de solicitud para muestras sujetas al RGPD. Tras cumplimentar el formulario, recibirá instrucciones adicionales para acceder al Workbench.
Datos clínicos
También nos complace anunciar la publicación detallada de datos fenotipados clínicos profundos de 12,585 participantes. Esta información contiene los siguientes datos:
- Edad en el momento del diagnóstico y de la aparición de síntomas
- Diagnóstico principal, actual y más reciente
- Exámenes cognitivos, como el Miniexamen Cognoscitivo (MMSE) y la Evaluación Cognitiva de Montreal (MoCA)
- Revisión patrocinada por la Movement Disorder Society de la Escala de Valoración Unificada de la Enfermedad de Parkinson (MDS-UPDRS)
- «Otros» fenotipos detallados, como la demencia de cuerpos de Lewy
En esta aportación de datos, cada uno de los 12,585 participantes que cuentan con información clínica también tienen información genética correspondiente
Datos a nivel individual
Ahora recopilamos datos de un total de 74 cohortes, de las cuales 46 son nuevas de esta publicación. Consulte el Panel de cohortes del GP2 para obtener más información sobre las cohortes compartidas.
La ascendencia genéticamente determinada de los participantes del GP2 con EP compleja se divide en 11 grupos. La tabla siguiente presenta la ascendencia determinada genéticamente de los participantes con EP compleja de esta aportación de datos que pasaron el control de calidad y se imputaron. Estas cifras incluyen muestras de datos anteriores que ahora se reagruparon en un nuevo archivo clúster y que pasaron el control de calidad, junto con las nuevas muestras genotipadas y compartidas por primera vez en la aportación actual.
Secuenciación del genoma completo de llamadas de variantes con DeepVariant-GLnexus
A diferencia de las versiones anteriores, en esta publicación usamos el pipeline DeepVariant de Google junto con GLnexus para la llamada de variantes a nivel de cohorte. DeepVariant es un programa de deep learning de llamado de variantes con un mayor rendimiento que las herramientas de vanguardia existentes a la hora de llamar variantes genéticas a nivel individual. También simplifica el proceso, mejora la precisión y aumenta la confiabilidad.
En futuras aportaciones, seguiremos ampliando la diversidad de los participantes. Los invitamos a visitar este panel para conocer nuestro progreso. Los usuarios que ya tengan acceso de nivel 2 pueden explorar los datos en más profundidad en nuestro navegador de cohortes, que presentamos en un artículo de blog anterior.
Como siempre, ¡consulte el README que acompaña cada aportación de datos del GP2 para obtener más detalles sobre pipelines, datos y análisis!






