En abril del 2022, el GP2 anunció la segunda aportación de datos en la plataforma Terra en colaboración con AMP® PD. Esta aportación contiene datos de las redes del GP2 tanto de etiología compleja como monogénica. Los datos relativos a enfermedades de etiología compleja ahora ascienden a un total de 8644 participantes genotipados (5249 EP, 3395 no EP). Las novedades de esta versión son Movement Disorders Genotypes and Phenotypes – Queen Square Brain Bank (MDGAP-QSBB), un banco de cerebros del Reino Unido, y el Estudio SYNAPS – Kazajstán (SYNAPS-KZ), una cohorte de EP de Kazajstán. También se han agregado muestras adicionales de CORIELL. Los datos relativos a enfermedades monogénicas ascienden a 235 participantes con EP con secuenciación del genoma completo (WGS) de la cohorte de PDGENEration, que fueron seleccionados a partir de criterios establecidos por tener una causa monogénica sospechosa de EP de la red de enfermedades monogénicas.
La diferencia clave entre los datos de genotipado de microarrays y los de WGS radica en el número de marcadores genéticos que se detectan durante el proceso de genotipado. La WGS proporciona una visión exhaustiva del genoma al analizar potencialmente los 3200 millones de pares de bases del genoma humano, mientras que el genotipado con un microarray dirigido (como el array NeuroBooster del GP2) analiza un número más acotado de hasta 1,9 millones de marcadores de región por muestra. La WGS es más adecuada para los análisis dirigidos a investigar variaciones genéticas raras, mientras que los datos genotipados imputados con un panel de referencia bien emparejado son una solución escalable y eficiente para estudiar variaciones genéticas más comunes.
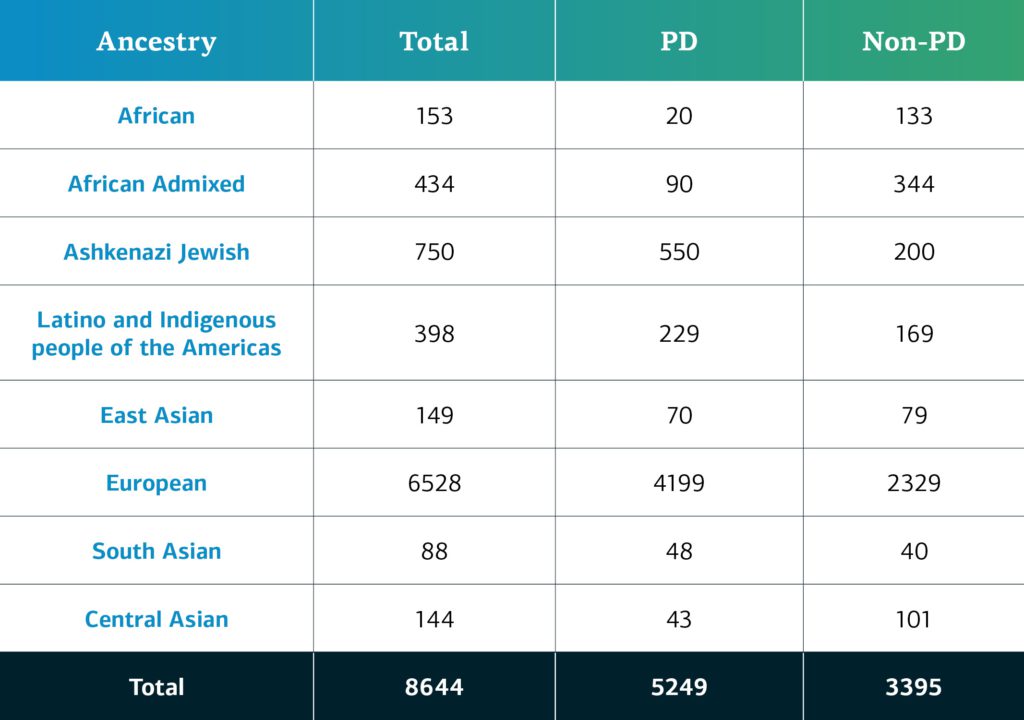
La ascendencia determinada genéticamente de los participantes del GP2 con enfermedades de etiología compleja se divide en nueve grupos. La siguiente tabla detalla la ascendencia determinada genéticamente de los participantes con enfermedades de etiología compleja de la segunda aportación del GP2 que pasaron el control de calidad y se incluyeron en el análisis.
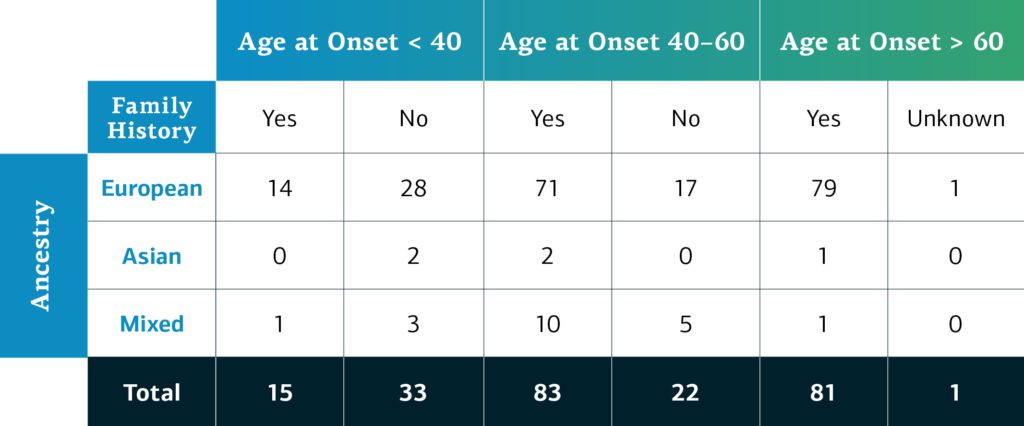
La ascendencia determinada genéticamente de los participantes del GP2 con enfermedades monogénicas se divide en cuatro grupos principales. Cabe destacar que se trata de los primeros datos disponibles de la red monogénica. A medida que procesemos más datos, dispondremos de muestras más diversas. La siguiente tabla detalla la ascendencia determinada genéticamente de los participantes con enfermedades monogénicas de la segunda aportación del GP2 por edad de aparición de la enfermedad y antecedentes familiares.
En las próximas aportaciones de datos, la diversidad de participantes seguirá aumentando. Le invitamos a seguir nuestro progreso en nuestro panel.
Además de incluir los primeros datos de WGS, incluimos también un nuevo tipo de datos adicionales: llamadas probabilísticas de variantes del número de copias (CNV) para todas las muestras genotipadas que hayan pasado el control de calidad (a nivel de genes, más las regiones flanqueantes de 250kb). La CNV se refiere a la variación en el número de veces que se repite un determinado tramo de ADN. Esta variación puede producirse a través de deleciones, inserciones u otros eventos y puede proporcionar más información sobre cómo la variación estructural afecta al riesgo de enfermedad. El proceso utilizado para producir las llamadas probabilísticas de CNV se puede encontrar en la página de Github del GP2. Se trata de un trabajo en curso, que se irá mejorando a medida que incluyamos más datos y ajustemos los pipelines. Por lo tanto, los datos CNV deben considerarse como una fuente de «generación de hipótesis». Las notas de uso se incluirán en el artículo de blog sobre la primera versión estable prevista para el próximo trimestre.
Esta versión contiene datos de WGS de la red de enfermedades monogénicas, además de los datos de enfermedades de etiología compleja genotipados por el array NeuroBooster. Encontrará más información sobre la estructura de los datos clínicos y genotipados de enfermedades de etiología compleja en el artículo de blog Los componentes de la primera aportación de datos del GP2 así como en el README, que se actualiza en cada aportación de datos y que está disponible en los espacios de trabajo oficiales de Terra del GP2. Los datos de WGS de la EP monogénica también se detallan en el mismo README.
Estamos muy contentos de poner esta versión beta a disposición de la comunidad de investigadores de la EP, ¡y aprovechamos para informarles de que pronto publicaremos más novedades!



