GP2第二次数据发布内容

2022年4月,GP2与AMP® PD一起在Terra平台进行了第二次数据发布。此次发布包含来自GP2复杂疾病数据网络和单基因数据网络的数据。复杂疾病数据现包含总共8644名基因型参与者的数据(5249名PD,3395名非PD)。此次发布增加了运动障碍基因型和表型,来自英国的皇后广场神经病学大脑库 (MDGAP-QSBB)以及SYNAPS 研究 -哈萨克斯坦 (SYNAPS-KZ) -一个来自哈萨克斯坦的PD队列。这次还一并发布了CORIELL样本。这组单基因疾病数据包含235个来自PDGENEration队列的全基因组测序(WGS)PD参与者 ,从单基因疾病网络中筛选而出,筛选标准为怀疑有PD单基因病因 (筛选标准链接) 。
基因分型微阵列和 WGS 数据之间的主要区别在于基因分型过程中检测到的遗传标记的数量。通过询问人类基因组中的几乎所有32亿个碱基对,WGS提供了基因组的全面视图,而使用靶向微阵列(例如GP2的定制NeuroBooster阵列)进行基因分型可以对每个样本更有针对性地询问多达190万个标有区域的标记数量。WGS更适合用于分析罕见基因变异,而基因型-得益于其匹配良好的参考面板-是研究更为常见的基因变异的可扩展的有效解决方案。
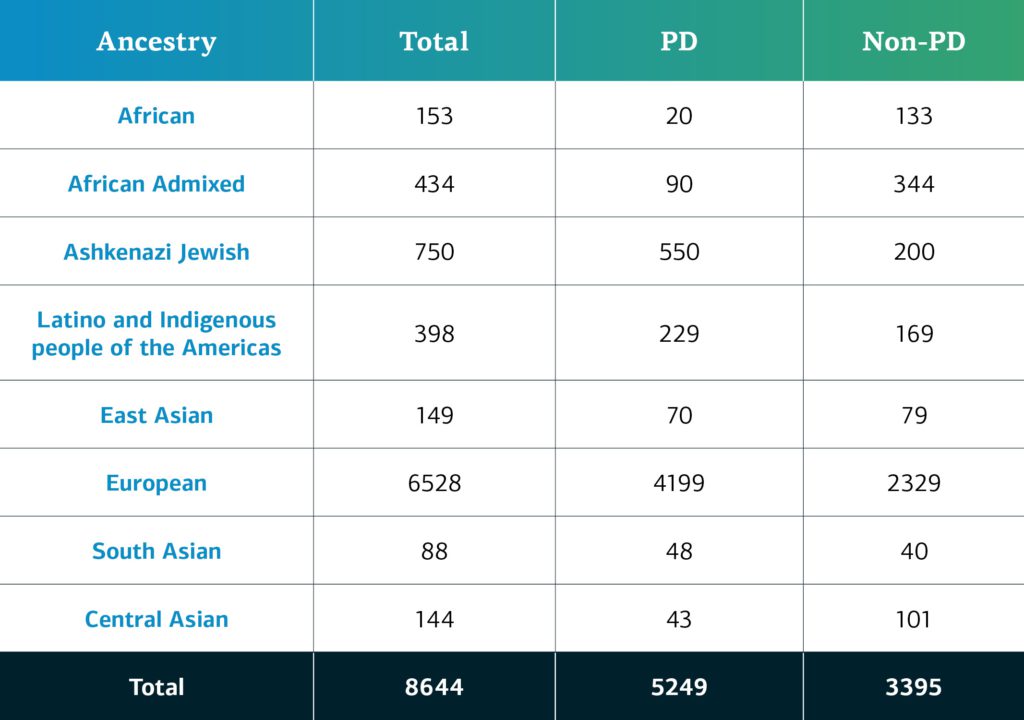
GP2复杂疾病参与者根据他们基因决定的血统分为九个血统组;下表详细介绍了GP2第二次数据发布中复杂疾病参与者的基因决定的血统,这些参与者已通过质量控制并被推算。
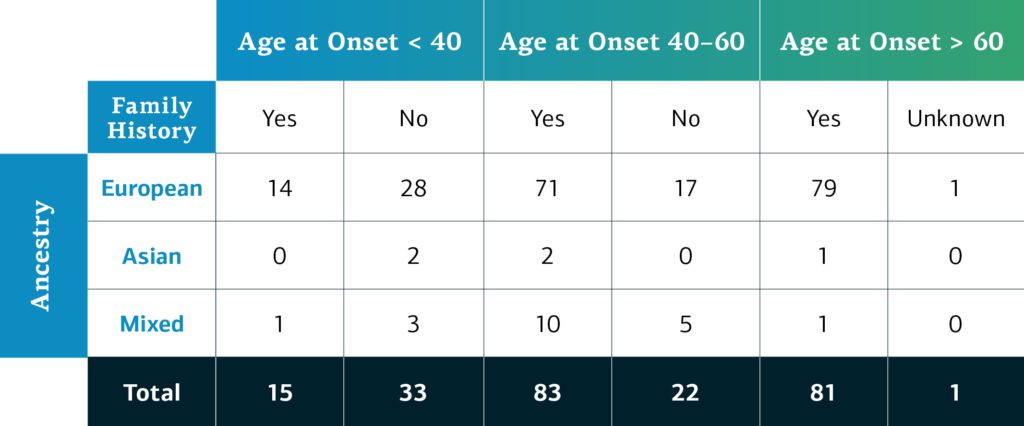
单基因疾病GP2参与者目前按其基因决定的血统分为四个主要血统组,因为这是来自单基因网络的第一组可用数据。随着更多的数据被处理,更多不同的样本将变得可用。下表按发病年龄和家族史详细介绍了GP2第2次发布中单基因疾病参与者的基因决定的血统。
未来的数据发布中,可用的参与者数据将更加多元。您可以在我们的操作面板上查看我们的进展。
除了包括第一个WGS数据之外,我们还加入了一个新的数据类型:对所有通过质量控制的基因分型样本进行概率拷贝数变异(CNV)分析(基因水平加上250kb侧翼区域)。CNV是指某段DNA重复次数的变化。这种变异可能是通过缺失、插入或其它事件产生的,并且可能提供更多关于结构变异如何影响疾病风险的信息。用于概率拷贝数变异(CNV)分析的管道可在GP2 Github上找到 。这项工作尚未完成,会随着我们包含更多数据并对管道进行调整而改善。可将这些CNV数据视为“假设生成”。报道下个季度进行的第一批稳定发布的博客文章中将包含使用说明。
此次发布包含来自单基因疾病网络的WGS数据以及 NeuroBooster阵列基因分型的复杂疾病数据。更多关于复杂疾病基因型和临床数据详见题为“GP2第一次数据发布内容”的博客文章 ,README格式在每次发布时会予以更新,在GP2官方Terra工作空间中可获取。同一个README文件中也包含单基因PD WGS数据的详细信息。
我们很高兴向PD研究社区提供这个beta版本,在不久的将来还会有更多发布!



