En febrero de 2023, el GP2 anunció la cuarta aportación de datos en la plataforma Terra, en colaboración con AMP® PD. Esta publicación está llena de nuevos datos y recursos.
En total, los nuevos datos corresponden a 2,583 participantes nuevos con enfermedades complejas, que se suman a las aportaciones anteriores de las Redes de la Enfermedad de Parkinson de Etiología Compleja y Monogénica. Los datos de enfermedades complejas corresponden ahora a un total de 17,485 participantes genotipados (9,429 con EP; 6,648 de control y 1,408 «otros»). Las cohortes agregadas al GP2 en esta aportación son:
- Canadian Open Parkinson Network (C-OPN), una red de colaboración compuesta por nueve universidades y centros de investigación de Canadá
- Genotipos y Fenotipos de Trastornos del Movimiento – Edinburgh Brain Bank (MDGAP-EBB), un banco de cerebros del Reino Unido
- Sydney Brain Bank (SBB), un banco de cerebros de Australia
La ascendencia determinada genéticamente de los participantes del GP2 con enfermedades complejas se divide en diez grupos (los nueve grupos que se detallan a continuación más una pequeña población de europeos finlandeses). La tabla siguiente presenta la ascendencia determinada genéticamente de los participantes con enfermedades complejas de esta aportación de datos que pasaron el control de calidad y se imputaron. Estas cifras incluyen muestras de datos anteriores que ahora se reagruparon en un nuevo archivo clúster y que pasaron el control de calidad, junto con las nuevas muestras genotipadas y compartidas por primera vez en la aportación actual. 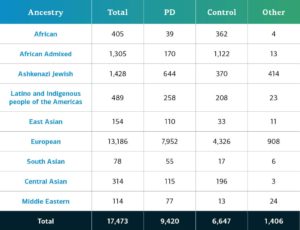 En futuras aportaciones, seguiremos ampliando la diversidad de los participantes. Los invitamos a visitar este panel para conocer nuestro progreso.
En futuras aportaciones, seguiremos ampliando la diversidad de los participantes. Los invitamos a visitar este panel para conocer nuestro progreso.
Un cambio importante de esta aportación es la pequeña modificación de uno de los umbrales usados en nuestros procesos de control de calidad. Para compartir más muestras, hemos eliminado las variantes genotipadas que tenían un rendimiento deficitario en repetidas ocasiones antes del proceso de control de calidad. La lista de variantes de rendimiento deficitario puede consultarse en el directorio de metadatos de los cubos de Nivel 2 y en Github. También hemos usado una tasa de llamada de muestreo menos restrictiva respecto a aportaciones anteriores: pasamos del 0.98 al 0.95. Con este cambio, podrá usar más muestras en sus análisis sin sacrificar la calidad.
Otra novedad de esta aportación son los cambios respecto a los datos clínicos. La columna de fenotipo («Phenotype») ahora solo incluye las etiquetas «PD», «Control» y «Other». Se ha agregado una nueva columna: «other_pheno», que ofrece una etiqueta de diagnóstico más detallado para los participantes etiquetados como «Otros/Other». Algunas de estas etiquetas de diagnóstico detallado incluyen personas no afectadas y afectadas vía el reclutamiento genético de EP, SWEDDs, y EP prodrómica, además de otros trastornos como el temblor esencial, la parálisis supranuclear progresiva, la demencia con cuerpos de Lewy y la atrofia multisistémica.
También hemos actualizado las llamadas probabilísticas a las variaciones del número de copias (CNV, por sus siglas en inglés) para todas las muestras genotipadas que pasaron el control de calidad (a nivel de gen y en las regiones flanqueantes de 250 kb) y ahora incluyen todas las muestras de la aportación 4. Dichos datos genotipados se agruparon en un archivo clúster con un formato específico del GP2 (disponible en los directorios Utils para usuarios con acceso tanto de nivel 1 como de nivel 2). Tanto el archivo clúster como el flujo (pipeline) usados para predecir las llamadas probabilísticas CNV pueden consultarse en el GP2 Github y pueden usarse con datos externos al GP2. Para más información sobre clustering utilizando el archivo clúster de genotipado específico del GP2 y las llamadas probabilísticas CNV, consulte el artículo de blog «Los componentes de la tercera aportación de datos del GP2».
En el artículo de blog «Los componentes de la primera aportación de datos del GP2» encontrará más información sobre la estructura de los datos clínicos y genotipados de enfermedades complejas, así como en el documento README, que se ha actualizado para incluir esta nueva aportación de datos y está disponible en los espacios de trabajo oficiales del GP2 de Terra. Los datos de WGS sobre la EP monogénica también se detallan en el mismo documento README.


