2022年10月,GP2与AMP® PD一起在Terra平台进行了第三次数据发布。本次发布包含了大量的新数据和新资源,样本数量几乎是上次发布的两倍。
本次发布包括6330个新增的复杂疾病参与者,对之前发布的复杂基因和单基因网络进行了补充。复杂疾病数据现在包括总共14,902个基因型参与者(8,190名PD;6,712名非PD)。此次发布的新队列包括:
- 澳大利亚帕金森病遗传学研究(APGs)
- 针对帕金森病的观察性临床研究——福克斯生物标志物新发现调查(BioFIND)
- 黑人和非裔美国人与帕金森病的联系(BLAACPD-KPM、BLAACPD-RUSH、BLAACPD-UAB、BLAACPD-UC)的参与站点,这是一个由黑人和非裔美国人构成的帕金森病队列
- 位于土耳其的Koc大学GP2队列(KOC)
- LRRK2突变携带者的PD队列——LRRK2队列小组(LCC)
- 运动障碍的基因型和表型–英国脑库伦敦国王学院(MDGAP-Kings)。
- 新西兰帕金森病计划(NZP3)
- 帕金森病基因与环境研究(PAGE)
- 帕金森病进展标志物倡议(PPMI)
- α-突触核蛋白的PD生物标志物研究——系统性突触核蛋白抽样研究(S4)
GP2复杂疾病参与者根据他们基因决定的血统分为十个血统组(以下九个组增加了少量的芬兰欧洲人);下表详细介绍本次数据发布中复杂疾病参与者的基因决定的血统,这些参与者已通过了质量控制并被推算。这些数字包括以前版本的样本,这些样本已经使用新的集群文件重新聚集,并通过了质量控制,同时还包括本次发布的新基因型和共享样本。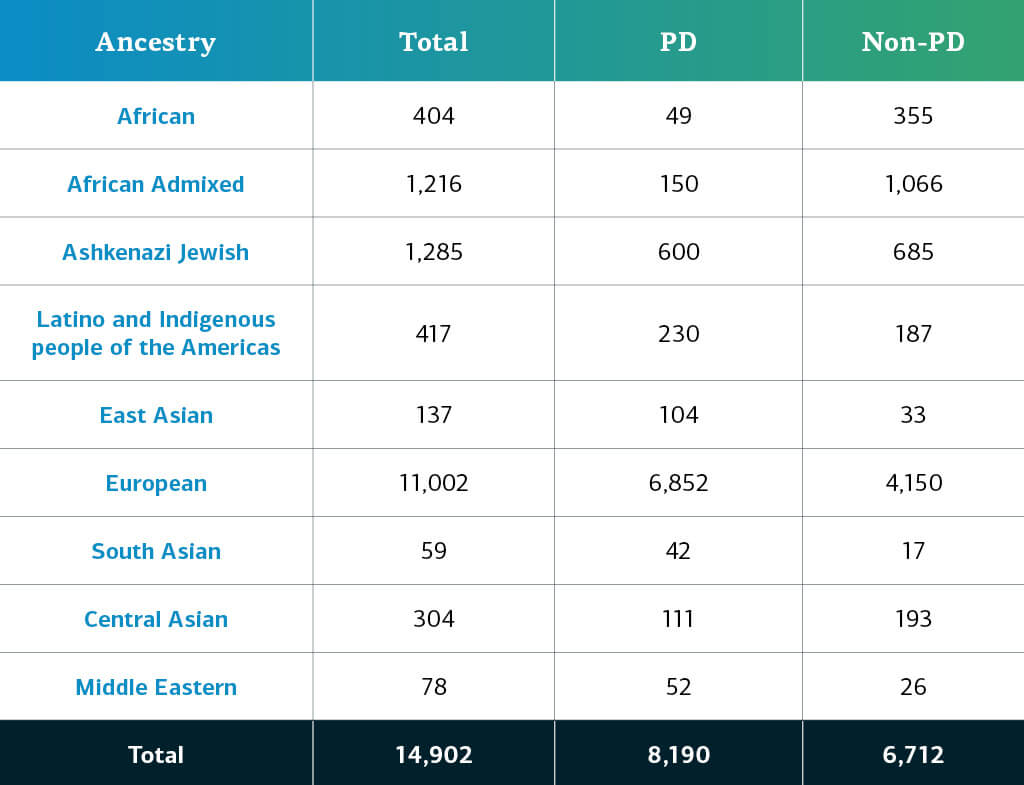 未来的数据发布还将继续提高参与者的多元性。您可以在我们的Dashboard上查看我们的进展。
未来的数据发布还将继续提高参与者的多元性。您可以在我们的Dashboard上查看我们的进展。
这个数据集的主要成果是包含了更多样本,包括基于阵列的和推定的基因数据,以及新增的临床和基因组元数据。该数据已使用定制的GP2基因型聚类文件进行了聚类(可在1级和2级数据访问下的utils目录中查看)。这个聚类文件是基于6个血统组的2793个样本, 这些样本代表了组成这次GP2数据发布的不同遗传血统,以及420个戈谢病病例的富集,以更好地捕获GBA风险基因中的相关变体。如果您希望将集群文件应用于GP2以外的数据,可通过GP2 GitHub存储库 下载该集群文件。
我们还更新了所有通过质量控制的基因型样本的拷贝数变异(CNV)调用(基因级加250kb侧翼区域)。CNV是指某段DNA重复次数的变化。这种变异可能是通过缺失、插入或其它事件产生的,并且可能提供更多关于结构变异如何影响疾病风险的信息。用于概率拷贝数变异(CNV)分析的管道可在GP2 Github上找到。这些结果在2级访问工作区中按遗传血统进行分层。这些概率CNV是一个很好的起点,可以优先考虑携带潜在插入、缺失或重复的感兴趣的基因的样本,以便开展后续研究和进一步分析。
更多关于复杂疾病基因型和临床数据详见题为“GP2第一次数据发布内容”’ 的博客文章,README格式已根据本次发布更新,可在GP2官方Terra工作空间中可获取。同一个README文件中也包含单基因PD WGS数据的详细信息。下次发布将提供更多单基因WGS数据。
下次发布预览:(计划2023年第一季度假日季发布):
- 更多基因型数据
- 更多WGS数据
- 更多临床元数据
- 最新的分析代码和工具
- 更多关于风险和发病年龄GWAS研究的汇总统计数据
本篇博文由GP2复杂疾病数据分析工作组成员撰写:Hampton Leonard、Mike Nalls、Dan Vitale和Mary Makarious;美国国立卫生研究院的Matthew Korestsky;Data Tecnica International公司/美国国立卫生研究院的Kristin Levine;以及单基因-数据分析工作组成员Zih-Hua Fang和Peter Heutink