2023年5月にGP2はAMP® PDとの協力のもとTerraプラットフォームにおいて第五回目のデータリリースを行ったことを発表しました。
本リリースには、7462人の複合疾患参加者と、487人のモノジェニック参加者が新たに追加され、複合・モノジェニックネットワークからのこれまでリリースに追加されました。
- 複合疾患データ(遺伝子型)は現在、合計24935名の遺伝子型同定参加者(PD12728人、対照群10533人、その他1674人)で構成されています。
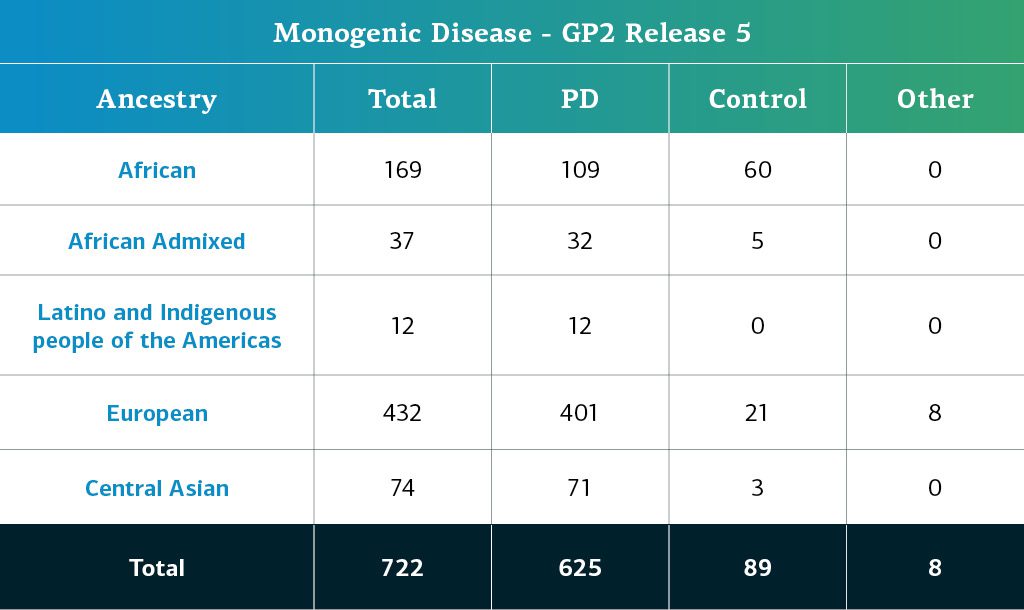
- モノジェニック疾患データ(全ゲノム配列)は、現在、計722人の配列同定された参加者で構成されています。
個人レベルデータ:本リリースでGP2に追加された新しいコホートは次の通りです:
- アリゾナブレインバンク 脳と身体寄付プログラム(BBDP)、米国を拠点とするブレインバンク
- パーキンソン病における自然免疫と適応免疫(IMMUNEPD)、アラバマ大学バーミンガム校の米国ベースの研究
- IPDGCアフリカ(IPDGCAF-NG)、ナイジェリアのコホート
- PDGENEration (PDGNRTN)、パーキンソン財団のコホート
- パーキンソン病および関連する運動障害(STELLENBOS)の遺伝学的研究、ステレンボッシュ大学の南アフリカのコホート
- マレーシアパーキンソン病遺伝学コホート(UMKLM)、マラヤ大学のマレーシアのコホート
- パーキンソン病および関連運動障害における遺伝子型-表現型相関(KUL)、マレーシア大学のモノジェニックコホート
- パーキンソン病および運動障害センターバイオリポジトリ(PDMDC)、ノースウェスタン大学のモノジェニックコホート
複合疾患GP2参加者の遺伝的祖先は、10の祖先グループ(以下の9つのグループと少数のフィンランドヨーロッパ人)に分けられています;以下の表は、このリリースにおける複合疾患参加者の遺伝的祖先を詳細に示し、品質管理に合格し、帰属されたものです。これらのデータには、新しいクラスターファイルを使用して再クラスター化され、現在のリリースに固有の新しくジェノタイピングおよび共有されたサンプルとともに品質管理を行った、以前のリリースサンプルが含まれています。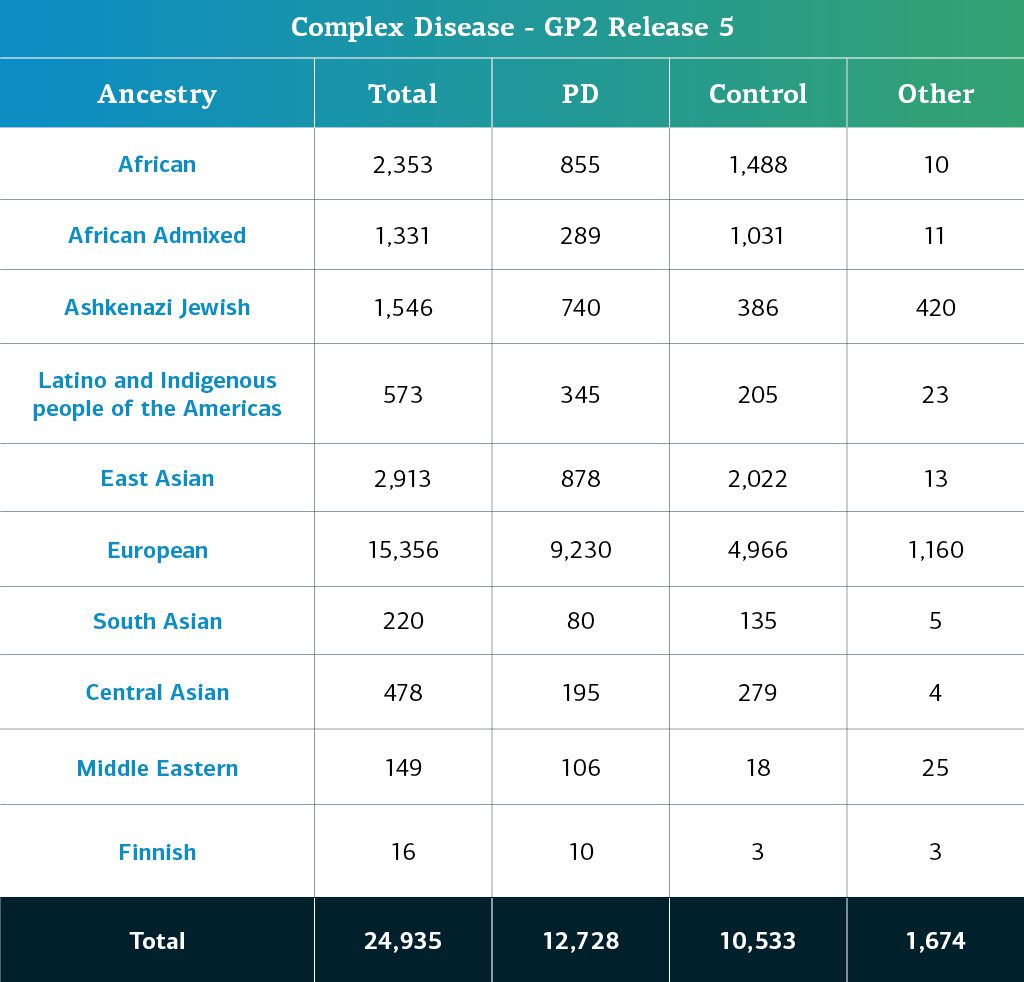
本リリースにおける重要な点:STELLENBOSは南アフリカのコホートで、地域とその歴史を反映した遺伝的祖先の混合を表します。この研究のメンバーに対する祖先予測の一部は、将来のリリースで新しい人口群の調整によって変化する可能性があります。本リリースでは、研究者がTier 2データの一部として分析に高混和性を含めるかどうか、その方法を選択できるように、すべての参加者に混和性推定値を提供しました。また、混和性が高いと現在のモデルからの祖先予測の信頼性が低下するため、この段階で分析から削除することを推奨するサンプルのリストも含まれています。
モノジェニック疾患GP2参加者の遺伝的に同定された祖先は、複合参加者と同じパイプラインから推定されます。以下の表は、GP2リリース5におけるモノジェニック疾患参加者の遺伝的に同定された祖先を詳述しています。
今後のデータリリースにより、参加者の多様性が増します。をチェックして進捗状況を確認できます。本リリースの重要な更新は、どの参加者が血縁関係があるかを判断する方法に小さな変更が加えられたことです。私たちは現在、KINGソフトウェアを使用しています。この変更により、以前にリリースされた参加者の一部の関係性推定値が更新された可能性があります。この変更により、推定関連性の閾値を詳述したファイルがリリースで利用可能になり、研究者は独自のカスタムフィルタリングの目的で使用できるようになります。予測される主要素に加え、下流の分析に利用可能な祖先ごとに計算された主要素が含まれるようになりました。
統計まとめ:
本リリースでは、新しい統計まとめもティア1アクセスを通じて利用できるようになりました。Kim et al 2023も、神経変性疾患知識ポータルとの継続的なコラボレーションを通じて利用できます。
- Nalls et al 2019 23andme in hg38を除く(欧州最大のPD GWAS)
- Loesch et al 2021(ラテンアメリカ・アメリカンインディアン最大のPD GWAS)
- Kim et al 202 23andmeを除く(ヨーロッパ、東アジア、ラテンアメリカ・アメリカンインディアン、アフリカ系アメリカ人を含むPDリスクの多祖先メタアナリシス)
- Rizig et al 2023 23andme in hg38を除く(アフリカとアフリカ混合最大のPD GWAS;GP2リリース5データを使用)
コピー番号バリアント:
品質管理に合格したすべての遺伝子型サンプル(遺伝子レベルと250kbの隣接領域)のコピー番号バリアント(CNV)コールは、リリース5の全サンプルを含むように更新されました。このデータは、カスタムGP2遺伝子型クラスタリングファイル(Tier 1 と Tier 2 の両方のデータ アクセスの下にある utils ディレクトリで利用可能)を使用してクラスター化されています。確率的CNVコールを予測するために使用されるクラスターファイルとパイプラインは、どちらもGP2外のデータで使用するためGP2 Githubにあります。カスタムGP2ジェノタイピングクラスタリングファイルを使ったクラスタリングとコピー番号バリアント確率コールの詳細については、GP2ブログの「GP2の第3次データリリースのコンポーネント」を参照してください。複合疾患の遺伝子型と臨床データの構造に関するより詳細な情報は、このリリースで更新され、公式GP2テラワークスペースで入手可能なREADMEと同様、ブログ記事で詳述されています。モノジェニックPD WGSデータも同じREADMEに詳述されています。
これまで通りパイプライン、データ、および分析の詳細については、各GP2リリースに付属のREADMEを参照してください。
GP2複合疾患データ解析、モノジェニックデータ解析、データ・コード展開ワーキンググループ代表








