Programme mondial sur la génétique de la maladie de Parkinson (GP2) et Partenariat pour l’accélération des traitements (Accelerating Medicines Partnership) : les membres de Parkinson’s Disease (AMP PD) unissent leurs forces pour devenir le guichet unique des données sur la génétique parkinsonienne (GP2) et génomique (AMP PD) et vous permettre de dynamiser vos travaux de recherche sur les nouvelles substances pharmaceutiques et les biomarqueurs, accélérant ainsi la découverte de nouveaux médicaments contre la maladie de Parkinson. A cela s’ajoute la collaboration informatique basée sur le nuage que nous avons créée afin de connecter entre elles ces deux bases de données de très grande envergure.
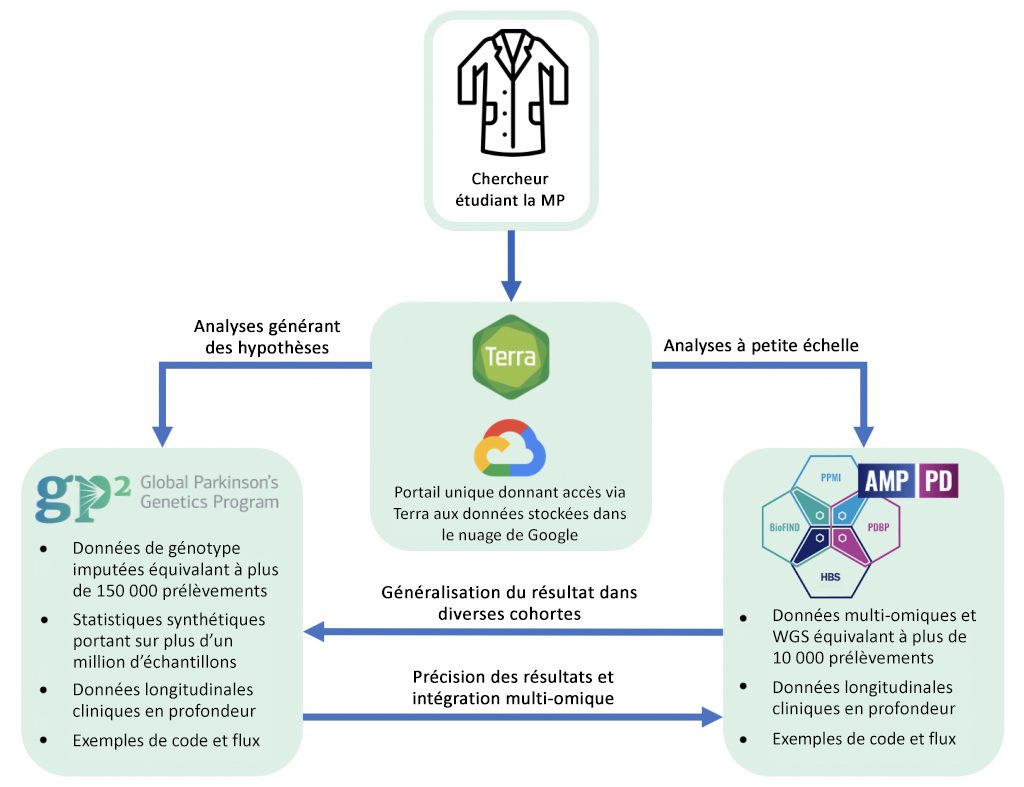
Nous offrons un point d’accès à la fois unique et sûr à une quantité massive de données stockées sur le nuage, en utilisant Terra comme squelette d’analyse de données, stockage de données et authentification de chercheur. Terra fait passer les opérations informatiques à une dimension supérieure grâce aux espaces collaboratifs qui comprennent blocs notes et flux de travail originaux sur le nuage. L’utilisation de l’informatique en nuage signifie que vous pouvez analyser les données dans un cadre plus sécurisé, sans les inconvénients des transferts de données onéreux et chronophages. Terra utilise la structure intuitive du bloc note de Jupyter pour une analyse claire et interactive. Terra vous permet aussi de publier votre espace de travail ainsi que vos résultats pour garantir une recherche reproductible et transparente. Les développements à venir incluent l’interopérabilité avec d’autres espaces de travail et plateformes consacrés aux maladies neurodégénératives pour mieux optimiser les valeurs des données.
Pourquoi situer ces deux programmes au même endroit ? Parce qu’ils sont complémentaires. Si vous voulez vous immerger dans dix mille échantillons ayant diverses logiques mutliomiques harmonisées, comprenant le séquençage d’ARN et le séquençage génomique, AMP PD est la base de données qu’il vous faut. Si vous cherchez à explorer les associations génétiques de populations diverses à l’échelle d’une biobanque, le GP2 est ce qu’il vous faut. Le GP2 et l’AMP PD incluent également l’accès aux données cliniques longitudinales pertinentes sur la plupart des participants.De façon générale, nous utilisons le GP2 comme base de données de recherche pour les analyses à grande échelle (générant des hypothèses) et poursuivons avec une fine analyse exploratoire de ces découvertes sur les cohortes harmonisées d’AMP PD.
L’avantage majeur tient au fait que la procédure d’accès et le point d’entrée sont les mêmes pour accéder aux deux bases de données, avec une architecture unique et la vaste documentation disponible pour réduire la courbe d’apprentissage. Nous avons développé ce graphique pour illustrer les cas d’utilisation des données du GP2 et AMP PD.
Ce blog a été écrit par les membres du GP2 et de l’AMP PD :
Mike A. Nalls, docteur ès sciences
Data Tecnica International | Etats-Unis .
Mike a fondé Data Tecnica au début de l’année 2017 après avoir passé plus de dix ans à étudier méthodes et analyses de données dans le domaine de la santé et autres domaines scientifiques. Il compte à son actif plus de 350 publications scientifiques (avant l’âge de 40 ans) portant sur les statistiques appliquées aux grands jeux de données, aux maladies cérébrales et à la génomique. C’est un fervent défenseur de la science ouverte, du travail collaboratif et de la transparence dans les sciences.
Hampton Leonard, master en sciences
Data Tecnica International / National Institutes of Health | Etats-Unis .
Hampton a une formation en science des données et d’apprentissage machine qu’elle applique à de grands jeux de données « multi-omiques » dans le domaine des maladies neurodégénératives. Elle se passionne pour la recherche portant sur les différences entre les niveaux cliniques et « omiques » et sur l’incidence de ces divergences sur les résultats des essais.
Matt Bookman
Verily Life Sciences | Etats-Unis .
Matt est concepteur de solutions pour l’équipe Terra chez Verify. Vice-président du groupe de travail AMP PD, il a travaillé aux côtés des autres collaborateurs du groupe au traitement des données génomiques et transcriptomiques pour les mettre à la disposition des chercheurs qualifiés à Terra. Matt est diplômé en mathématiques appliquées de l’UCLA et a un Master en sciences de l’informatique et biologie informatique de l’Université de Stanford.
Eline Appelmans
Foundation for the National Institutes of Health | Etats-Unis
Au titre de directrice des Partenariats pour la recherche en neurosciences, le prof. Appelmans travaille en coordination avec les National Institutes of Health, organismes à but non-lucratif, responsables de secteurs et de projets FNIH sur la mise en œuvre de programmes tels que l’Accelerating Medicines Partnership (AMP) for Alzheimer’s Disease (AMP-AD) (Partenariat pour accélérer la recherche médicale sur la maladie de Parkinson), AMP for Parkinson’s Disease (AMP-PD), AMP for Schizophrenia (AMP-SCZ), Alzheimer’s Disease Neuroimaging Initiative 3 Private Partner Scientific Board (ADNI3 PPSB) et le Biomarkers Consortium Neuroscience Steering Committee (BC NSC), ainsi que d’autres projets en cours de développement.



