Das Global Parkinson’s Genetics Program (GP2) und Accelerating Medicines Partnership: Parkinson-Krankheit ( AMP® PD ) haben sich zusammengeschlossen, um Ihnen als zentrale Anlaufstelle für genetische (GP2) und genomische (AMP PD) Daten zur Parkinson-Krankheit zu dienen und Ihnen die Entdeckung neuer Wirkstofftargets und Biomarker zu erleichtern und so die Entwicklung neuer Medikamente gegen Parkinson zu beschleunigen. Wir setzen kollaboratives Cloud Computing ein, um diese beiden riesigen Datenbanken miteinander zu verbinden.
Wir stellen einen einzigen sicheren und unkomplizierten Zugangspunkt zu einer riesigen Datenmenge, die in der Cloud gespeichert ist. Dabei nutzen wir Terra als Backbone für die Datenanalyse, Datenspeicherung und Authentifizierung von Forscher*innen. Mit Terra erreicht die Computerarbeit ein neues Niveau. Es bietet kollaborative Arbeitsbereiche mit Cloud-nativen Notebooks und Workflows. Cloud Computing bedeutet, dass Sie die Daten an einem besser gesicherten Ort analysieren können und keine zeitaufwändigen und teuren Datenübertragungen notwendig sind. Die Verwendung der intuitiven Jupyter-Notebookstruktur bei Terra ermöglicht eine klare und interaktive Analyse. Sie können Ihre Arbeitsbereiche zusammen mit Ihren Ergebnissen veröffentlichen, wodurch eine reproduzierbare und transparente Forschung gewährleistet wird. Für die Zukunft geplant ist die Interoperabilität mit Arbeitsbereichen und Plattformen für andere neurodegenerative Erkrankungen, um den Nutzwert der Daten weiter zu optimieren.
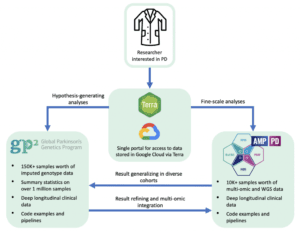
Warum sollten diese beiden Programme an einem Ort zusammengeführt werden? Weil sie sich hervorragend ergänzen. Wenn Sie tief in zehntausend Proben mit verschiedenen harmonisierten Multi-Omik-Auswertungen, einschließlich RNAseq und genomeSeq, eintauchen möchten, dann ist der Datenbestand von AMP PD genau das Richtige für Sie. Wenn Sie genetische Assoziationen in vielfältigen Populationen im Biobank-Maßstab erforschen möchten, so sind Sie bei GP2 an der richtigen Adresse. Sowohl bei GP2 als auch bei AMP PD sind auch kuratierte relevante klinische Longitudinaldaten zu den meisten Teilnehmer*innen enthalten. Grundsätzlich nutzen wir GP2 als Forschungsdatenbestand für groß angelegte Analysen (Hypothesenbildung), um dann Entdeckungen möglicherweise weiter zu verfeinern und in den vereinheitlichten AMP-PD-Kohorten genauer zu untersuchen.
Und das Beste dabei: Mit einem einzigen Zugangspunkt und denselben Formalitäten erhält man Zugang zu beiden Datenbeständen, mit einer einheitlichen Architektur und ausführlicher Dokumentation, um den Einarbeitungsaufwand gering zu halten. Zur Illustration der Anwendungsfälle für GP2- und AMP-PD-Daten haben wir diese Übersichtsgrafik entwickelt.
Dieser Blog wurde von den folgenden Mitgliedern von GP2 und AMP PD geschrieben:
Mike A. Nalls, PhD
Data Tecnica International | USA
Mike gründete Data Tecnica Anfang 2017, nachdem er über ein Jahrzehnt Erfahrung in der Analyse großer Datensätze und der Methodenforschung im Gesundheitswesen und anderen wissenschaftlichen Bereichen gesammelt hatte. Er hat über 350 von Experten begutachtete Publikationen (vor dem 40. Lebensjahr) auf dem Gebiet der angewandten Statistik bei großen Datenbeständen, Gehirnkrankheiten und Genomik auf seinem Konto. Er ist ein großer Fürsprecher von Open Science, Zusammenarbeit und Transparenz in der Wissenschaft.
Hampton Leonard, MS
Data Tecnica International / National Institutes of Health | USA
Hampton verfügt über einen Hintergrund in Datenwissenschaft und maschinellem Lernen, den sie auf große multiomische Datensätze im Bereich neurodegenerativer Erkrankungen anwendet. Ihre Leidenschaft gilt der Erforschung von Unterschieden auf klinischer und omischer Ebene und der Frage, wie diese Unterschiede die Ergebnisse klinischer Studien beeinflussen können.
Matt Bookman
Verily Life Sciences | USA
Matt ist Solutions Architect für das Terra-Team bei Verily. Als Ko-Vorsitzender der AMP PD Data Working Group arbeitete er mit anderen Wissenschaftler*innen daran, genomische und transkriptomische Daten zu verarbeiten und sie qualifizierten Forscher*innen in Terra zur Verfügung zu stellen. Matt erwarb seinen BS in angewandter Mathematik an der UCLA und seinen MS in Computerwissenschaften und Computerbiologie an der Stanford University.
Eline Appelmans
Stiftung der National Institutes of Health | USA
Als Direktor für neurowissenschaftliche Forschungspartnerschaften arbeitet Dr. Appelmans in Abstimmung mit den NIH, gemeinnützigen Organisationen, Branchenführern und Projektleitern des FNIH an der Umsetzung der Accelerating Medicines Partnership (AMP) für die Alzheimer-Krankheit (AMP-AD), der AMP für die Parkinson-Krankheit (AMP PD), der AMP für Schizophrenie (AMP-SCZ), des privaten wissenschaftlichen Gremiums der Alzheimer’s Disease Neuroimaging Initiative 3 (ADNI3 PPSB) und des Biomarkers Consortium Neuroscience Steering Committee (BC NSC) sowie an anderen in der Entwicklung befindlichen Projekten.



