Aperçu général
En décembre 2023, le GP2 a annoncé la sixième publication des données sur les plateformes Terra et Verily® Workbench , en collaboration avec l’AMP® PD. Cette édition comprend plus de 20 000 participants supplémentaires qui viennent s’ajouter aux éditions précédentes des réseaux de maladies complexes et monogéniques.
- Les données sur les maladies complexes (génotypes), y compris les échantillons restreints localement, comptent désormais un total de 44 831participants génotypés (24 709 atteints de Parkinson, 17 246 cas témoins et 2 876 « autres » phénotypes).
- Si l’on soustrait les échantillons restreints, les participants atteignent un total de 33 436 (17 129 atteints de Parkinson, 13 872 cas témoins et 2 435 « autres » phénotypes).
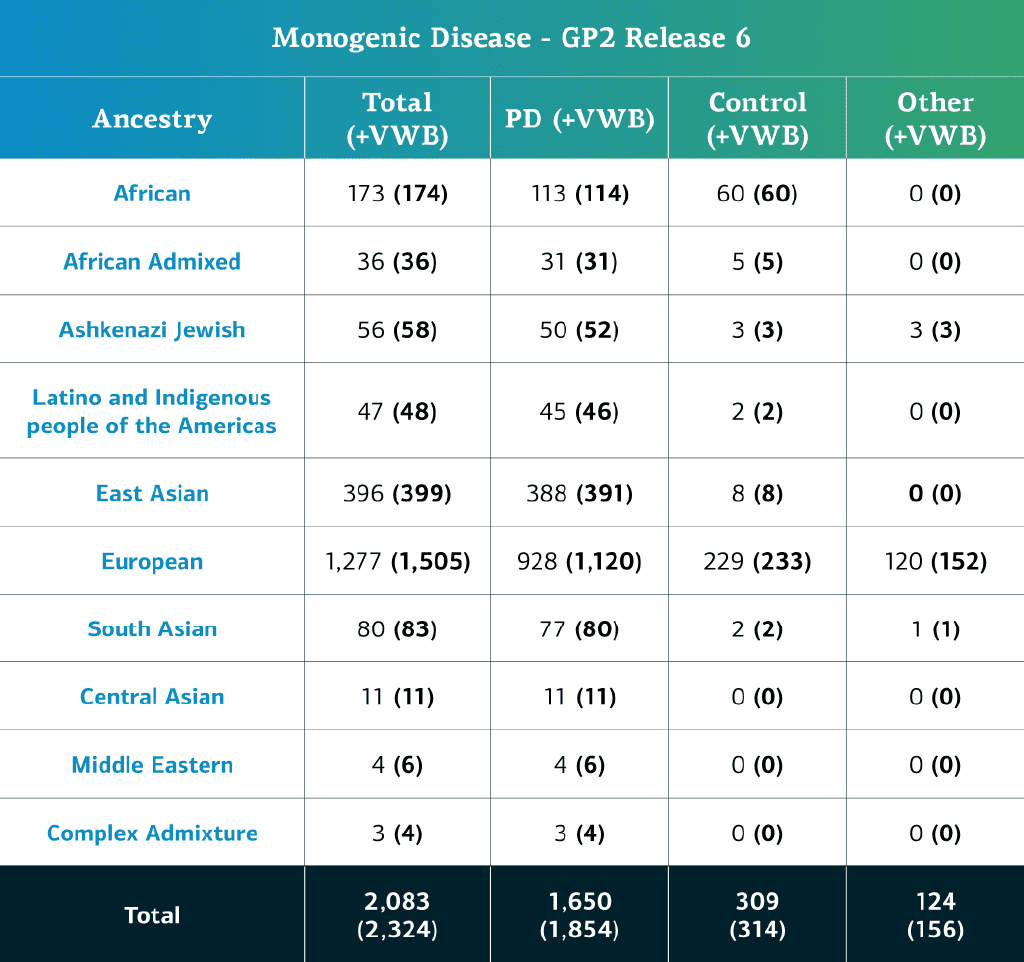
- Les données sur les maladies monogéniques (séquences du génome complet) comprennent désormais un total de 2 324 participants séquencés (1 854 atteints de Parkinson, 314 cas témoins et 156 « autres » phénotypes).
- Lorsque l’on retire les échantillons restreints localement, le nombre de participants s’élève à 2 083 (1 650 atteints de Parkinson, 309 cas témoins et 124 « autres » phénotypes).
- 12 585 personnes qui disposent d’une information profonde du phénotypage clinique ont également une information génétique.
Quoi de neuf dans cette publication?
- Echantillons additionnels de maladies complexes (à génotype) et maladie monogéniques (génome complet)
- Introduction d’échantillons restreints localement par le RGDP via Verily Viewpoint Workbench
- Introduction des données cliniques pour près de 12 000 personnes.
- Introduction d’un nouveau groupe d’ascendance → Histoire de la complexité du métissage (CAH)
- Mises à jour de mesures de contrôle de qualité pour la publication des données de génotypage.
- Mise à jour de l’appel de variant, désormais avec DeepVariant pour la publication de donnes du génome complet.
Mise à jour du contrôle de qualité
En résumé, GenoTools (v1.0.0) réalise les étapes de contrôle de qualité suivantes : recherche de doublons, discordance de sexe, élagage du taux cible, vérification et indication des personnes de la même famille et vérification du taux d’hétérozygotie.
Contrairement aux précédentes publications, nous ne réalisons plusle filtrage de variant suivant : fréquence de l’allèle mineur (MAF), Hardy-Weinberg (HWE), ou comptage d’allèle mineur (MAC). Si vous souhaitez filtrer selon le procédé utilisé lors des éditions précédentes, nous vous invitons à consulter le document README pour obtenir plus de détails et une indication des seuils utilisés.
Histoire complexe du métissage (CAH)
CAH ou Histoire complexe du métissage, est un nouveau groupe d’ascendance créé dans le GP2 pour la sixième publication des données. Sa création est une réponse au grand nombre d’échantillons de personnes d’Afrique du Sud ou à fort métissage ayant été classées, à tort, dans le groupe d’ascendance asiatique (CAS) dans la cinquième édition des données. Dans la sixième édition, le groupe d’ascendance CAH comporte surtout des échantillons de l’université de Stellenbosch (Le Cap, Afrique du Sud),de l’Institut Coriell (Camden, New Jersey, Etats-Unis) et de la Fondation Parkinson (Miami, Florida, Etats-Unis). Nous considérons que les échantillons classés CAH sont trop métissés pour être mélangés aux analyses des autres groupes d’ascendance.
Echantillons RGPD restreints localement via Verily Viewpoint Workbench
Nous sommes heureux d’annoncer que certains utilisateurs pourront avoir accès aux échantillons restreints localement, également dénommés échantillons régis par le Règlement général sur la protection des données (RGPD), grâce à notre collaboration avec Verily Viewpoint Workbench. Workbench est un environnement sécurisé pour la gestion et l’analyse des données biomédicales visant à renforcer la collaboration en matière de recherche et de reproduction des données grâce à l’intégration par le nuage. Il encourage le partage de l’espace de travail, y compris Python et le code R, et offre un ensemble de services sur le nuage pour la gestion et l’analyse des données. Workbench soutient l’intégration sécurisée de l’utilisation des données et l’authentification personnalisée, ce qui en fait un environnement de recherche idéal, sûr et évolutif pour que le GP2 y héberge ses échantillons restreints.
A ce stade, alors que le GP2 continue de déployer ses solutions de partage de données pour les données protégées par le RGPD, la sixième édition ne sera disponible que pour les membres du consortium GP2 et ses partenaires. Au fur et à mesure que les tests et la mise en œuvre se poursuivront en 2024, ces solutions seront mises à la disposition d’un plus grand nombre de chercheurs. Tous les échantillons de la sixième édition se trouvent sur Workbench, tandis que tous les échantillons de la sixième édition non régis par le RGPD sont disponibles sur Terra (comme pour les éditions passées). Pour avoir accès à la totalité de l’édition sur Verily Viewpoint Workbench, vous devez :
- Disposer d’une autorisation d’accès GP2 de catégorie 2
- Être membre du consortium du GP2 (contribution de cohorte, partenaire GP2 ou équipe de projet d’analyses).
- Veuillez remplir le formulaire de demande d’échantillons régis par le RGPD. Une fois le formulaire rempli, vous recevrez les instructions d’accès à Workbench.
Données cliniques
Nous sommes également très heureux d’annoncer que cette édition comprend l’accès aux données de phénotype profond de 12 585 personnes. Cette information comprend les informations suivantes :
- Âge au moment du diagnostique
- Diagnostique primaire, actuel et suivant
- Examens cognitifs tels que le Mini-Mental State Examination (MMSE) et le Montreal Cognitive Assessment (MoCA)
- Révision de l’échelle UPDRS parrainée par la Movement Disorder Society
- Détail des «autres» phénotypes, tels que la démence à corps de Lewy
Dans cette édition, chacune des 12 585 personnes ayant un dossier clinique a également le dossier génétique correspondant.
Données individuelles
Nous disposons désormais des données de 74 cohortes, dont 46 sont nouvelles dans cette édition. Veuillez consulter le tableau de bord des cohortes du GP2 pour obtenir des informations complémentaires au sujet des cohortes qui ont été partagées.
L’ascendance génétiquement déterminée des participants à maladie complexe du GP2 est répartie en 11 groupes d’ascendance. Le tableau ci-dessous répertorie l’ascendance génétiquement déterminée des participants à maladie complexe pris en compte dans cette édition et ayant passé le contrôle de qualité. Ces chiffres inclus les échantillons des éditions précédentes qui ont été réorganisés selon une nouvelle méthode de classement des dossiers et ont été soumis à un contrôle qualité, ainsi que de nouveaux échantillons de génotypes, spécifiques à cette édition. 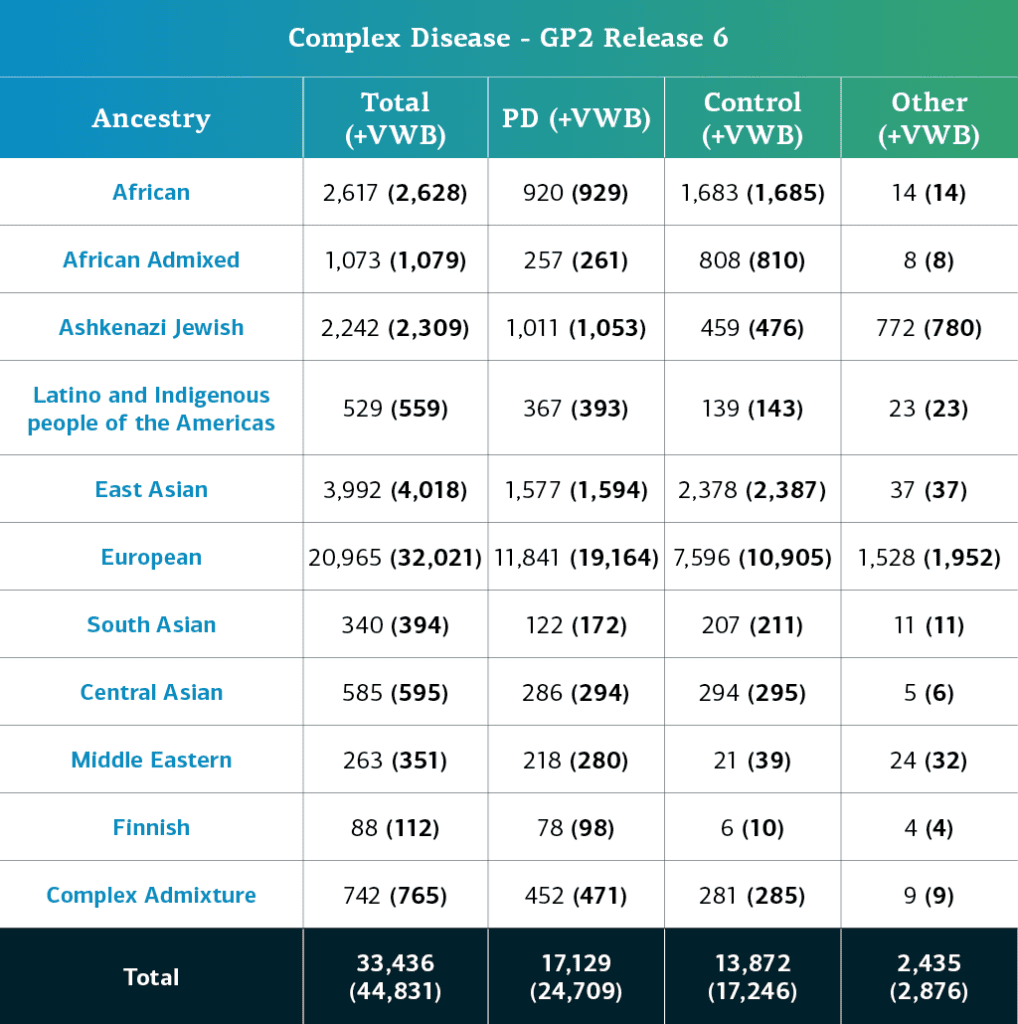
Séquençage du génome entier à haut-débit grâce à DeepVariant-GL nexus
Dans cette édition, contrairement aux précédentes, nous utilisons le fluxGoogle’s DeepVariant associé à GLnexus pour l’appel des variants au niveau des cohortes. DeepVariant est un outil d’appel des variants à apprentissage profond qui dépasse les outils les plus sophistiqués actuels en faisant un appel de variant génétique ciblé. Cela simplifie également le processus, tout en en renforçant la précision et la fiabilité.
Les futures éditions de données contribueront à renforcer la diversité des participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès. Les utilisateurs ayant un accès de catégorie 2 peuvent d’ores et déjà explorer les données sur notrenavigateur de cohortes, dont nous avons parlé dans un précédent blog.
Comme toujours, veuillez consulter le document README qui accompagne chaque publication du GP2 pour plus d’informations sur les canaux, les données et les analyses !











