2023年5月,GP2与AMP® PD一起在Terra平台进行了第五次数据发布。
此次发布包括7462名新的复杂疾病参与者和487名新增的单基因疾病参与者,对之前发布的复杂基因和单基因网络进行了补充。
- 复杂疾病数据(基因类型)现在包括总共 24935 名基因分型的参与者(PD组12728人 ,对照组10533人,“其他”组1674名其他表型)。
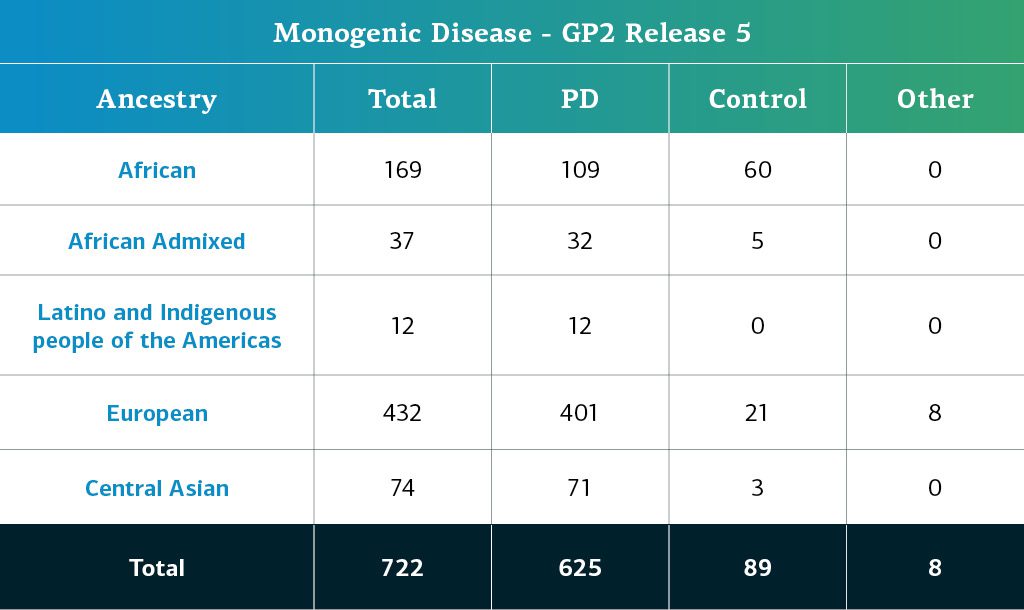
- 单基因疾病数据(全基因组序列)现在总共由722名测序参与者构成。
个人级别的数据:
此次发布的GP2的新增队列有:
- 总部设在美国的脑库——亚利桑那州脑库大脑和身体捐赠计划(BBDP)。
- 美国阿拉巴马大学伯明翰分校的一项研究:帕金森病患者的先天免疫和获得性免疫。
- 尼日利亚队列——IPDGC非洲(IPDGCAF-NG)。
- 帕金森基金会队列–PDGENEATION(PDGNRTN)
- StellenBosch大学的南非队列研究帕金森病及相关运动障碍(STELLENBOS)的遗传学研究。
- 马来亚大学的马来西亚队列——马来西亚帕金森遗传队列(UMKLM)
- 马来西亚大学的单基因队列研究中帕金森病和相关运动障碍(KUL)的基因-表型相关性
- 西北大学的单基因队列——帕金森病和运动障碍中心生物信息库(PDMDC)
GP2复杂疾病参与者根据他们基因决定的血统分为十个血统组;下表详细介绍了本次数据发布中复杂疾病参与者的基因决定的血统,这些参与者已通过质量控制并被推算。这些数字包括以前版本的样本,这些样本已经使用新的集群文件重新聚集,并通过了质量控制,同时还包括本次发布的新基因型和共享样本。
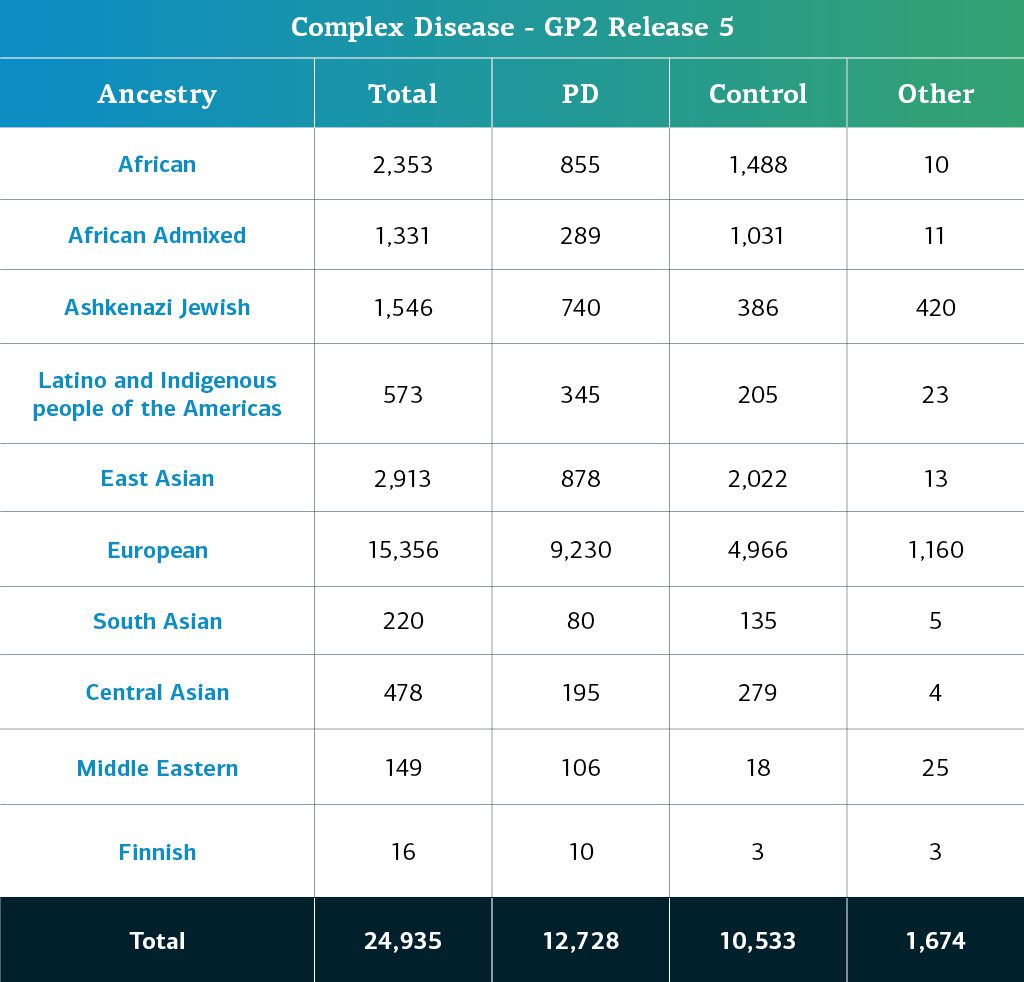
本次数据发布中值得注意的是:STELLENBOS是南非的队列,反映了该地区及其历史的遗传血统的混合。随着在未来数据发布中不断适应这一新增队列,我们对这项研究成员的一些血统预测可能会改变。在本次发布中,我们提供了所有参与者的混合估计,便于研究人员决定是否以及如何将高混合纳入分析,作为2级数据的一部分。我们还包括了一个样本列表,但建议目前在这个阶段从分析中删除这些样本,因其高混合水平会降低我们对当前模型的血统预测的信心。
单基因疾病GP2参与者的由基因决定的血统是根据与复杂参与者相同的管道估计得来的。下表详细介绍了GP2第5次发布中单基因疾病参与者的基因决定的血统。
未来的数据发布还将继续提高参与者的多元性。您可以在队列dashboard中查看进展。
此次发布的一个重要更新是对我们确定哪些参与者是亲属的方式进行了微调。我们现在使用的软件是King。这一变化可能会导致我们之前发布的一些参与者的相关性估计的改变。由于出现了这一变化,现在将在发布数据时提供一个详细说明估计相关性阈值的文件,供研究人员根据自身需求进行筛选。除了预测的主成内容,我们现在还包括按血统计算的主要内容,可用于下游分析。
统计信息摘要:
我们很高兴地宣布,本次发布还将通过1级读取提供新的摘要统计数据。Kim等人2023年的数据也可通过我们与神经退行性疾病知识门户的持续合作获得。
- Nalls等人2019年的数据不含23andme的hg38(最大的欧洲PD GWAS)
- Loesch等人2021年的数据(最大的拉美/美洲PD GWAS)。
- Kim等人2023年的数据,不含23andme(包括欧洲人、东亚人、拉丁美洲/美洲人和非裔美国人在内的PD风险的多血统元分析)。
- Rizig等人2023年的数据不含23andme的hg38(最大的非洲和非洲混合体的PD GWAS;使用GP2第5版数据)
拷贝数变异:
我们更新了所有通过质量控制(基因水平加上250kb侧翼区域)的基因型样本的拷贝数变异(CNV)检出,以囊括版本5中的所有样本。该数据已使用定制的GP2基因型聚类文件进行了聚类(可在1级和2级数据访问下的utils目录中查看)。用于预测概率CNV检出的聚类文件和管道均可在GP2 Github上找到,可与GP2以外的数据一起使用。有关使用定制的GP2基因分型集群文件和拷贝数变量概率检测进行聚类的更多信息,请参阅GP2博客中 “GP2第三次数据发布内容”。更多关于复杂疾病基因型和临床数据详见题为的博客文章,README格式已根据本次发布更新,可在GP2官方Terra工作空间中获取。同一个README文件中也包含单基因PD WGS数据的详细信息。
关于管道、数据和分析的更多详细信息,请参考每个GP2版本所附带的README!
代表GP2复杂疾病数据分析、单基因数据分析和数据和代码传播工作组。











