En février 2023, le GP2 a annoncé le publication de la quatrième édition de données sur la plateforme Terra, en collaboration avec AMP® PD. Cette dernière édition regorge de nouvelles données et ressources.
Cette édition comprend 2 583 participants supplémentaires dans la catégorie des maladies complexes, qui viennent s’ajouter aux éditions précédentes des réseaux de maladies complexes et monogéniques. Les données sur les maladies complexes totalisent désormais 17 485 participants au génotype référencé (9 429 MP ; 6 648 sujets de contrôle et 1 408 «autres»). Les nouvelles cohortes qui viennent s’ajouter au GP2 dans cette édition sont :
- Le Canadian Open Parkinson Network (C-OPN), un réseau collaboratif regroupant neuf universités et centres de recherche canadiens.
- Le Movement Disorders Genotypes and Phenotypes – Edinburgh Brain Bank (MDGAP-EBB), une banque de cerveaux basée au Royaume-Uni.
- Le Sydney Brain Bank (SBB), une banque de cerveaux basée en Australie.
Le lignage génétiquement déterminé de la maladie complexe chez les participants du GP2 se divise en dix lignées (les neuf lignées ci-dessous et un petit nombre de Finlandais d’Europe). Le tableau ci-après présente la lignée génétiquement déterminée des participants à cette édition, atteints de maladies complexes, qui ont passé le contrôle de qualité et ont été pris en compte. Ces chiffres comprennent les échantillons des éditions précédentes, reclassés selon une nouvelle nomenclature et soumis à un contrôle de qualité, ainsi que de nouveaux échantillons génotypés et exclusifs à cette édition. 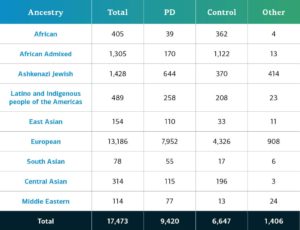 Les futures publications de données contribueront à renforcer la diversité des participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès.
Les futures publications de données contribueront à renforcer la diversité des participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès.
Une des mises à jour importante de cette édition est le petit changement introduit dans les seuils qui s’appliquent à notre chaîne de contrôle de qualité. Afin de partager un plus grand nombre d’échantillons, nous avons retiré les variants génotypés qui affichent systématiquement un niveau de qualité insuffisant avant de subir le contrôle de qualité. Une liste des variants peu performants est disponible sur le répertoire de métadonnées de la liste de niveau 2 sur Github. Nous avons également utilisé une simulation des taux d’échantillonnage moins exigeante, passant de 0,98 à 0.95. Ainsi, vous pourrez utiliser un plus grand nombre d’échantillons dans vos analyses, sans porter atteinte à la qualité.
Autre mise à jour de cette édition : la mise à jour des données cliniques. La colonne de phénotype («Phénotype») a été mise à jour pour n’inclure que les étiquette «MP», «contrôle» ou «Autres». Une colonne supplémentaire a été ajoutée, «autre_phéno», qui fournit une vignette de diagnostique plus détaillée sur les participants de la catégorie «Autres». Certaines de ces labels de diagnostiques plus détaillés incluent des sujets atteints et non atteints, passés au crible du recrutement génétique de la MP, SWEDD, MP prodormale ainsi que d’autres troubles tels que les tremblements essentiels (TE), la paralysie supranucléaire progressive (PSP), la démence à corps de Lewy (DCL) et l’atrophie multisystématisée (AMS).
Dans cette quatrième édition, nous avons également mis à jour la variabilité du nombre de copies d’un gène (NCV) pour tous les échantillons génotypés soumis au contrôle de qualité (niveau de gène plus 250kb de séquençage des régions flanquantes). Ces données ont été regroupées par génotype dans un fichier de classement propre au GP2 (disponible dans les paramètres des répertoires sous accès aux données de niveau 1 et 2). Le dossier de clusters et la procédure utilisée pour les probabilités des appels NCV sont disponibles sur le Githubdu du GP2 pour l’utilisation des données hors du GP2. Pour de plus amples informations sur les regroupements utilisant le fichier de génotypage spécifique au GP2 et les appels de variabilité du nombre de copies d’un gène, veuillez consulter «Les éléments de la troisième édition des données du GP2» sur le blog du GP2.
Des informations complémentaires sur la structure du génotype des maladies complexes et sur les données cliniques sont disponibles dans l’article «Les éléments de la première édition des données du GP2»’ ainsi que dans le document README, mis à jour pour cette édition et disponible dans les espaces de travail officiels du GP2 sur Terra. Les données monogéniques sur la maladie de Parkinson-séquençage de génome complet (PD WGS) sont également disponibles dans ce même document README.










