Im Februar 2023 kündigte GP2 die vierte Datenfreigabe auf der Terra-Plattform in Zusammenarbeit mit AMP® PD an. Diese Version enthält eine Fülle von neuen Daten und Ressourcen.
Zusätzlich zu den Beständen der früheren Freigaben des Netzwerks Komplexe Krankheit und des Monogenic-Netzwerks umfasst diese Version 2583 zusätzliche neue Teilnehmer*innen mit komplexen Erkrankungen. Die Daten zur komplexen Krankheit umfassen nun insgesamt 17.485 genotypisierte Teilnehmer*innen (9429 mit Parkinson-Erkrankung, 6648 Kontrollpersonen und 1408 „Sonstige“). Folgende Kohorten sind mit dieser Freigabe zu GP2 hinzugekommen:
- Canadian Open Parkinson Network (C-OPN), ein Kooperationsnetzwerk aus neun kanadischen Universitäten und Forschungszentren
- Movement Disorders Genotypes and Phenotypes – Edinburgh Brain Bank (MDGAP-EBB), eine im Vereinigten Königreich ansässige Gehirnbank
- Sydney Brain Bank (SBB), eine in Australien ansässige Gehirnbank
Die genetisch determinierte Abstammung von GP2-Teilnehmer*innen mit komplexer Krankheit wird in zehn Abstammungsgruppen gegliedert (die neun Gruppen unten plus eine kleine Anzahl finnischer Europäer*innen); die Tabelle unten gibt Aufschluss über die genetisch determinierte Abstammung der Teilnehmer*innen mit komplexer Krankheit in dieser Freigabe, welche die Qualitätskontrolle durchlaufen haben und imputiert wurden. Diese Zahlen beinhalten Proben aus früheren Freigaben, die unter Verwendung der neuen Cluster-Datei neu geclustert wurden und die Qualitätskontrolle durchlaufen haben, sowie die neu genotypisierten und gemeinsam genutzten Proben, die nur in dieser aktuellen Freigabe enthalten sind.
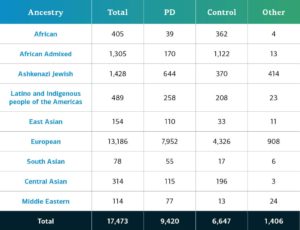
Durch zukünftige Datenfreigaben wird sich die Vielfalt der verfügbaren Teilnehmenden weiter erhöhen. Auf unserem Dashboard können Sie sich über den Fortschritt unserer Arbeiten informieren.
Eine wichtige Änderung bei dieser Freigabe betrifft einen der Schwellenwerte, die in unserer QC-Pipeline verwendet werden. Um mehr Proben teilen zu können, haben wir genotypisierte Varianten mit einem durchgängig niedrigen Qualitätsniveau vor Durchlaufen des QC-Prozesses entfernt. Eine Liste der weniger guten Varianten finden Sie im Metadatenverzeichnis der Tier-2-Buckets und auf Github. Wir haben auch eine weniger strenge Call Rate für die Proben verwendet und diese von 0,98 auf 0,95 gesenkt. So konnten mehr Proben für unsere Analysen verwendet, ohne Abstriche bei der Qualität machen zu müssen.
Eine weitere Änderung bei dieser Freigabe betrifft eine Aktualisierung bei den klinischen Daten. Die Phänotyp-Spalte wurde aktualisiert und enthält jetzt nur noch die Kennungen „Parkinson-Erkrankung“, „Kontrolle“ und „Sonstige“. Eine zusätzliche Spalte mit der Bezeichnung „other_pheno“ wurde hinzugefügt; diese enthält eine detailliertere Diagnosekennzeichnung für Teilnehmende mit der Bezeichnung „Sonstige“ (Other). Einige dieser detaillierteren Diagnosekennzeichnungen umfassen nicht betroffene und betroffene Personen über gezielte genetische Parkinson-Rekrutierung, SWEDDs und prodromales Parkinson sowie andere Störungen wie essentiellen Tremor (ET), progressive supranukleäre Lähmung (PSP), Lewy-Körper-Demenz (DLB) und Multisystematrophie (MSA).
Die Aufrufe der Kopienzahlvarianten (CNV) für alle genotypisierten Proben, welche die Qualitätskontrolle durchlaufen haben (Gen-Ebene plus 250kb flankierende Abschnitte), wurden aktualisiert und alle Proben in der 4. Freigabe einbezogen. Diese Daten wurden mit Hilfe einer benutzerdefinierten GP2-Genotyp-Clustering-Datei geclustert (verfügbar in den utils-Verzeichnissen beim Tier-1- wie auch beim Tier-2-Datenzugang). Die Cluster-Datei und die Pipeline für die Vorhersage der probabilistischen CNV-Aufrufe finden sich auf dem GP2 Github zur Nutzung mit Daten außerhalb von GP2. Weitere Informationen zum Clustering mit Hilfe der speziellen GP2-Genotypisierungs-Cluster-Datei und den probabilistischen CNV-Aufrufen finden sich in dem Beitrag „Die Komponenten der dritten Datenfreigabe von GP2“ auf dem GP2 Blog.
Nähere Informationen zur Struktur der Genotyp- und klinischen Daten zur komplexen Krankheit finden sich im Blogbeitrag „Die Komponenten der ersten Datenfreigabe von GP2“’ sowie in der README-Datei, die für diese Freigabe aktualisiert wurde und in den offiziellen GP2-Terra-Workspaces verfügbar ist. Die WGS-Daten zur monogenen Parkinson-Erkrankung sind ebenfalls in dieser README-Datei enthalten.









