2023年2月,GP2与AMP® PD一起在Terra平台进行了第四次数据发布。此次发布包含了海量的新数据和新资源。
本次发布包括2583个新增的复杂疾病参与者,对之前发布的复杂基因和单基因网络进行了补充。复杂疾病数据现在包括总共 17,485 名基因型参与者(PD组9429 人;控制组6648人,“其他组”1408 人)。此次发布的GP2的新增队列有:
- 加拿大开放帕金森网络(C-OPN),这是由九所加拿大大学和研究中心组成的合作网络
- 运动障碍的基因型和表型–爱丁堡脑库(MDGAP-EBB),一家来自英国的脑库
- 悉尼脑库(SBB),一家总部位于澳大利亚的脑库
GP2复杂疾病参与者根据他们基因决定的血统分为十个血统组(以下九个组增加了少量的芬兰欧洲人);下表详细介绍本次数据发布中复杂疾病参与者的基因决定的血统,这些参与者已通过了质量控制并被推算。这些数字包括以前版本的样本,这些样本已经使用新的集群文件重新聚集,并通过了质量控制,同时还包括本次发布的新基因型和共享样本。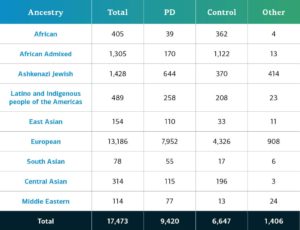 未来的数据发布还将继续提高参与者的多元性。您可以在操作面板上查看我们的进展。
未来的数据发布还将继续提高参与者的多元性。您可以在操作面板上查看我们的进展。
此版本的一个重要更新是对我们的QC渠道中使用的一个阈值进行了微调。为分享更多样本,我们已在开展QC流程之前移除了质量持续不佳的基因变异体。在Tier 2存储桶的元数据目录和Github上可找到表现较差的变异体列表。我们还采用了不太严格的样本检出率,从0.98降为0.95。如此一来,便能在不牺牲质量的情况下使用更多的样品进行分析。
此版本的另一大更新是对临床数据的更新。表型列(“表型”)已更新为标签“PD组”、“控制组”或“其他组”。新增的‘其他表型’一栏,为参与者提供了更详细的“其他”诊断标签。其中一些更详细的诊断标签包括通过靶向PD基因招募到的未受影响和受影响的个人、SWEDDS和前驱PD,以及其他疾病,如特发性震颤(ET)、进行性核上性麻痹(PSP)、路易体痴呆(DLB)和多系统萎缩(MSA)。
我们更新了所有通过质量控制(基因水平加上250kb侧翼区域)的基因型样本的拷贝数变异(CNV)检出,以囊括版本4中的所有样本。该数据已使用定制的GP2基因型聚类文件进行了聚类(可在1级和2级数据访问下的utils目录中查看)。用于预测概率CNV检出的聚类文件和管道均可在GP2 Github上找到,可与GP2以外的数据一起使用。有关使用定制的GP2基因分型集群文件和拷贝数变量概率检测进行聚类的更多信息,请参阅GP2博客中 “GP2第三次数据发布内容”。
更多关于复杂疾病基因型和临床数据详见题为“GP2第一次数据发布内容”’ 的博客文章,README格式已根据本次发布更新,可在GP2官方Terra工作空间中获取。同一个README文件中也包含单基因PD WGS数据的详细信息。










