Global Parkinson’s Genetics Program (GP2) と Accelerating Medicines Partnership in Parkinson’s Disease (AMP® PD) の両方で作成されたデータは、Broad InstituteがMicrosoftおよびVerilyと共同で開発したバイオインフォマティクス用クラウドプラットフォーム、Terraでホストしています。Terraでは、クラウド上で直接解析を行うことができるため、データを個人のパソコンにダウンロードする必要がありません。これにより、高いデータ保護とプライバシー基準を守ることができ、構造化された共有可能なコードと結果を通じて、オープンサイエンスと再現性を促進します。Terraは2種類の解析に対応しています。JupyterノートブックとWDLワークフローです。ノートブックは、コードスニペットを含むセルに分解されたドキュメントで、解析を順次実行することができます。ノートブックは、Pythonを含む多くの言語をサポートし、さまざまな分析に役立ち、可視化や文書化に最適です。最終的には、ノートブックが提供できる以上の計算能力や、より長い分析時間が必要になることがあります。このような場合には、Terraワークフローを使用することができます。ワークフローはWDL(「ウィドル」と発音します)言語を使用して構築され、クラウドサーバー上のバッチジョブと同じように送信されます。ワークフローは、サンプルサイズの大きなアライメント(整合化)やGWASなどの高出力解析に有効です。
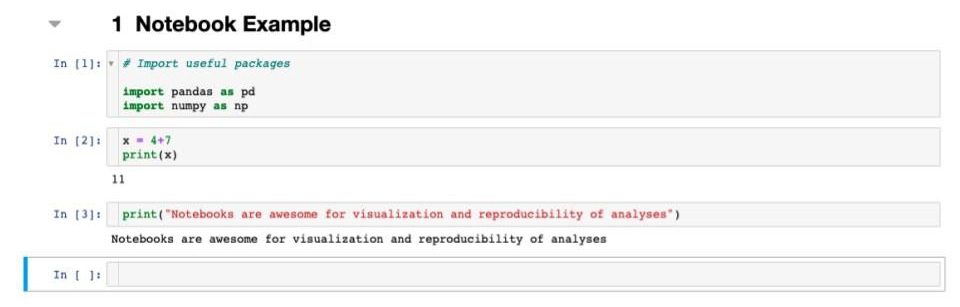
ノートブックの構成例
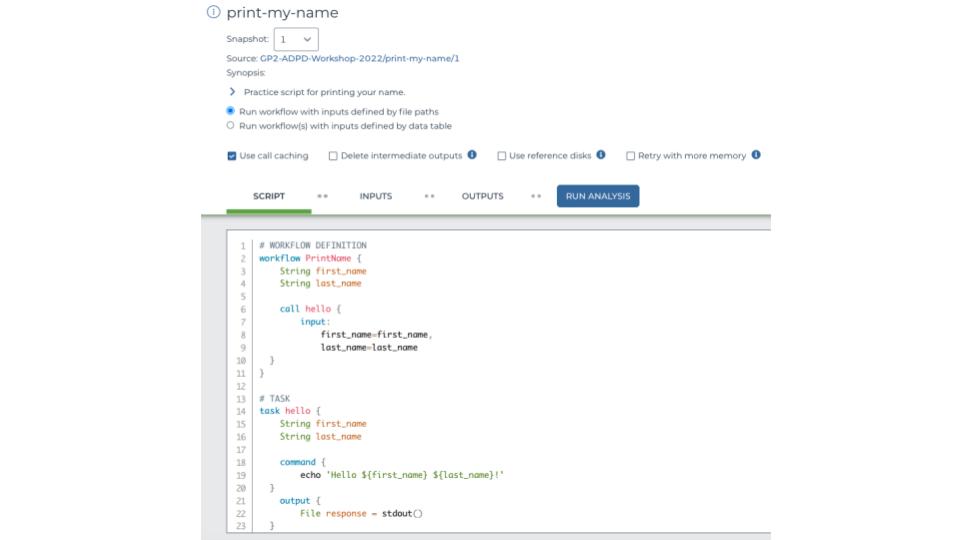
WDL構造例
 GP2データにアクセスすると、GP2 Terraの公式ワークスペースにもアクセスすることができます。Tier 1 と Tier 2 のワークスペースには、最新の GP2 データリリースの README ファイルがあります。各リリースに含まれるすべてのファイルの説明が記載されていますので、ぜひ一読されることをお勧めします。GP2データに関連するコードリソース、ノートブック、ワークフローが開発されると、これらのワークスペースにホストされるようになるので、頻繁にチェックしてください。
GP2データにアクセスすると、GP2 Terraの公式ワークスペースにもアクセスすることができます。Tier 1 と Tier 2 のワークスペースには、最新の GP2 データリリースの README ファイルがあります。各リリースに含まれるすべてのファイルの説明が記載されていますので、ぜひ一読されることをお勧めします。GP2データに関連するコードリソース、ノートブック、ワークフローが開発されると、これらのワークスペースにホストされるようになるので、頻繁にチェックしてください。 まず、プロジェクトのアイデアをGP2に提出し、審査を受ける必要があります。承認されると、あなたの機関が課金プロジェクトとワークスペースをセットアップするか、GP2があなたに代わってセットアップします。
まず、プロジェクトのアイデアをGP2に提出し、審査を受ける必要があります。承認されると、あなたの機関が課金プロジェクトとワークスペースをセットアップするか、GP2があなたに代わってセットアップします。
ワークスペースがセットアップされたら、分析用の新しいノートブックを作ることから始めましょう。ノートブックを実行するには、クラウド環境を作成する必要があります。
クラウド環境は必要に応じてカスタマイズできますが、現在利用可能なすべての帰属複合病GP2データを使用する予定であれば、少なくとも100GBの持続的なディスクスペースを要求して、リリース2.0時点で82GBのデータで作業するための大まかなスペースを確保することを確認してください。
ただし、リソースが多いほどコストも高くなることを覚えておいてください。
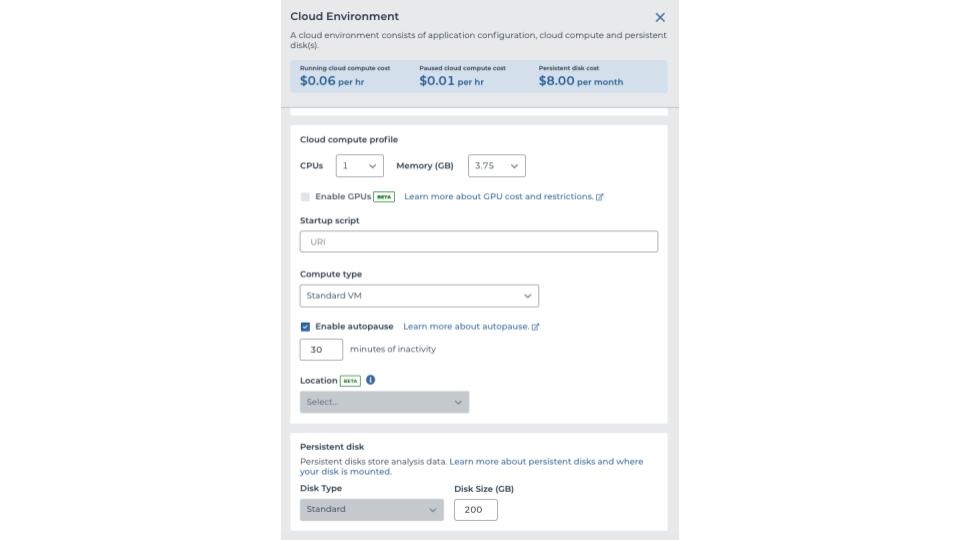
クラウド環境の例
 クラウド環境が稼働し、新しいノートブックがセットアップされたら、分析を開始する準備が整います。
クラウド環境が稼働し、新しいノートブックがセットアップされたら、分析を開始する準備が整います。
ノートブックをセットアップするために、下記やGP2公式ワークスペースにある便利なパッケージなどを利用するとよいでしょう。
便利なパッケージ
環境設定
利用可能なデータを確認したり、クラウドからワークスペースにデータを移動したりするには、gsutilツールセットを使用します。例えば、gp2tier2バケットにあるデータを一覧表示するには、次のようなコマンドを使用します 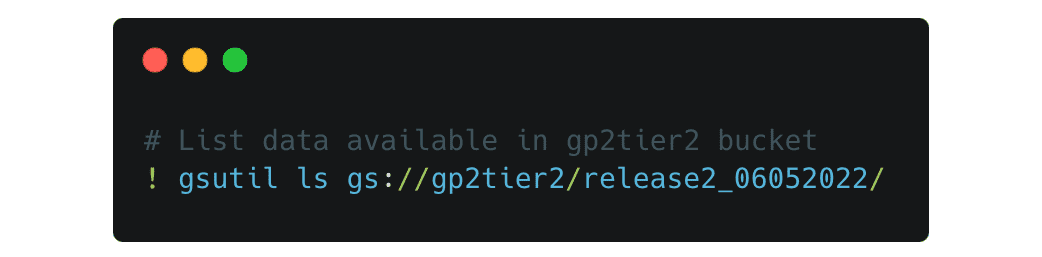 。これは、release 2 フォルダにある利用可能なすべてのファイルをリストアップします
。これは、release 2 フォルダにある利用可能なすべてのファイルをリストアップします 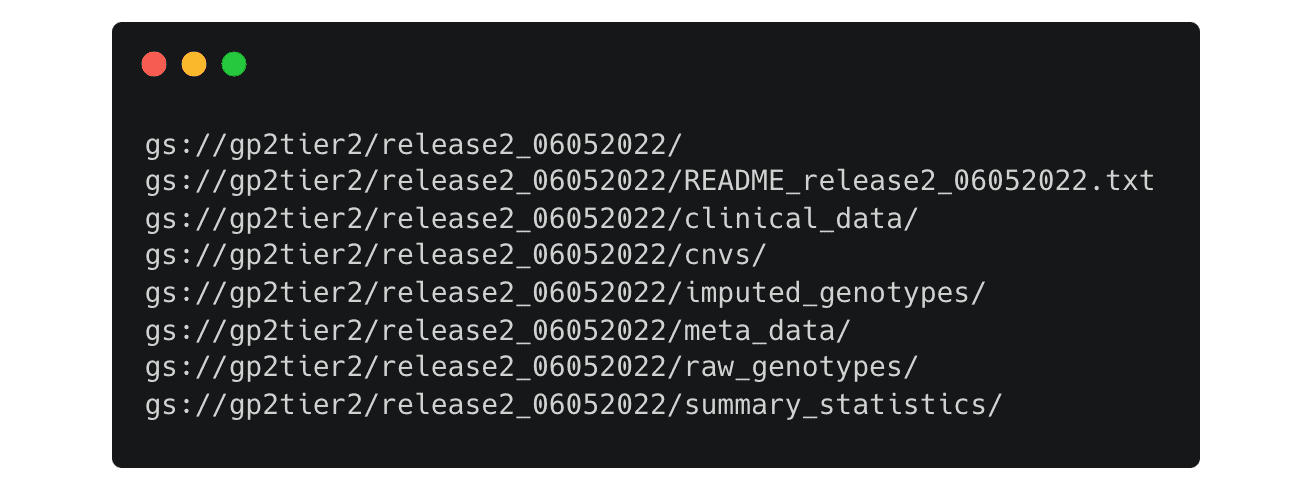 。各ディレクトリのパスは変更可能です。例えば、
。各ディレクトリのパスは変更可能です。例えば、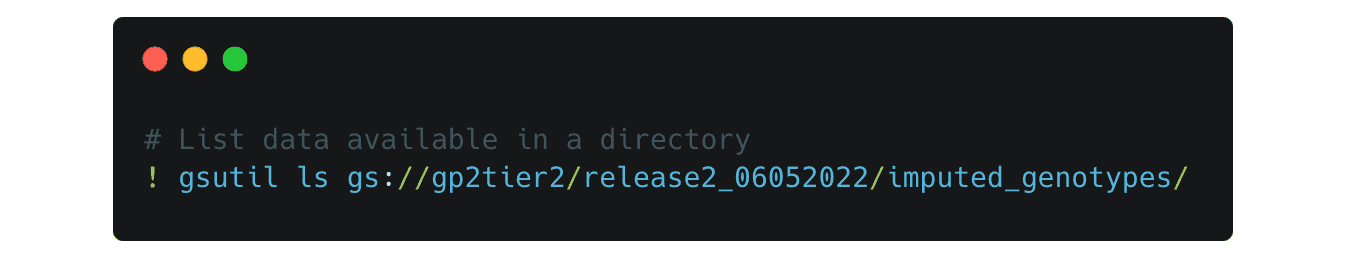 のように変更すると 帰属遺伝子型データのフォルダが予測される祖先別にリストアップされます
のように変更すると 帰属遺伝子型データのフォルダが予測される祖先別にリストアップされます 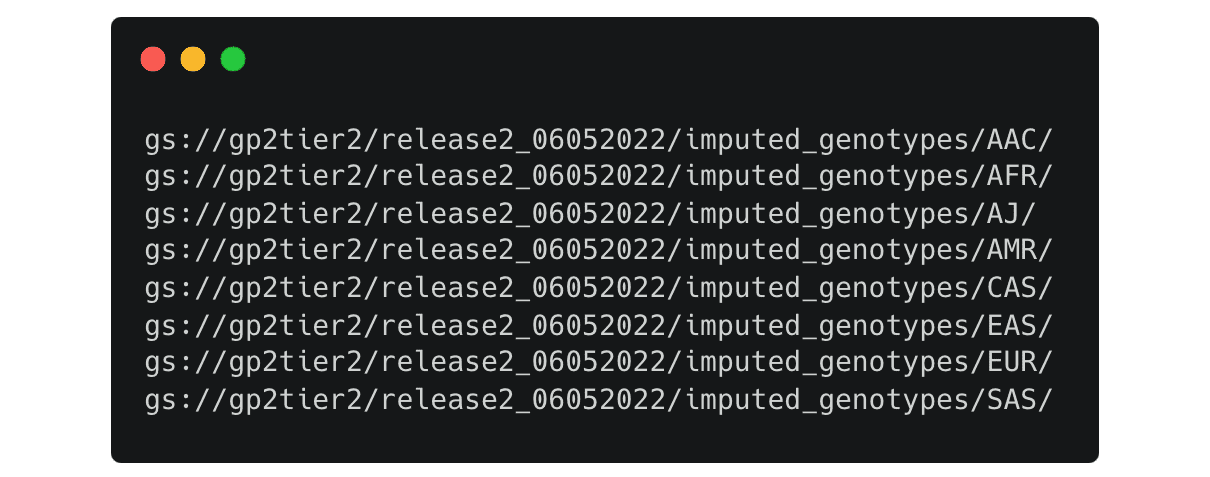 。必要なファイルが決まったら、このようにワークスペースにコピーします。まず、ファイルをコピーするためのディレクトリを作成します。
。必要なファイルが決まったら、このようにワークスペースにコピーします。まず、ファイルをコピーするためのディレクトリを作成します。 次に、新しいディレクトリにファイルをコピーします。この例では、Nalls et al.のPD要約統計をコピーしています。2019から23andMeのデータを除いたもの。
次に、新しいディレクトリにファイルをコピーします。この例では、Nalls et al.のPD要約統計をコピーしています。2019から23andMeのデータを除いたもの。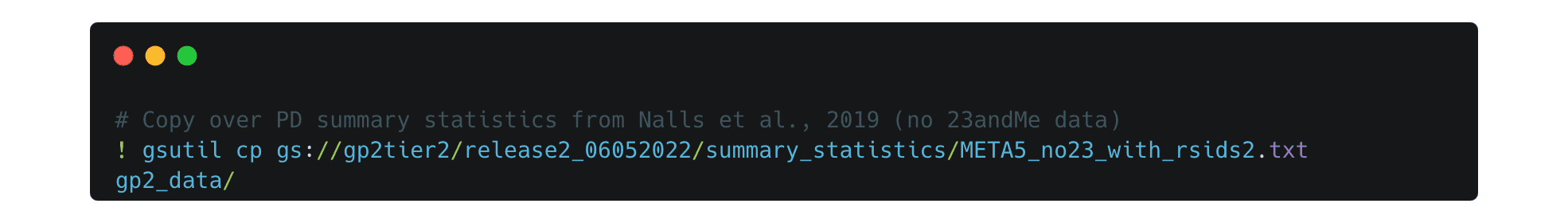 ファイルを見て、すべてが正しくコピーされたことを確認します。
ファイルを見て、すべてが正しくコピーされたことを確認します。
 これで、データを分析する準備ができました。
これで、データを分析する準備ができました。
以上がGP2データにおけるワークスペースとノートブックの使い方の短い例の説明となります。より詳細なTerraのチュートリアルについては、GP2 Learning Management Systemを参照して、’Course 1:を完了させてください。Terraを使用したデータアクセス&分析。




