Die Daten, die im Rahmen des Global Parkinson’s Genetics Program (GP2) sowie der Accelerating Medicines Partnership in Parkinson’s Disease (AMP® PD) generiert werden, werden auf Terra gehostet, einer Cloud-Plattform für Bioinformatik, die das Broad Institute gemeinsam mit Microsoft und Verily entwickelt hat. Mit Terra lassen sich Analysen direkt in der Cloud durchführen, so dass man keine Daten mehr auf den eigenen Computer herunterladen muss. So können wir die hohen Anforderungen an Datenschutz und Vertraulichkeit erfüllen und mittels strukturiertem, gemeinsam nutzbarem Code und Ergebnisbestand eine offene Wissenschaft und Reproduzierbarkeit ermöglichen.
Terra unterstützt zwei Arten von Analysen: Jupyter Notebooks und WDL Workflows. Notebooks sind in Zellen unterteilte Dokumente, die Codeschnipsel enthalten, mit denen man sequenzielle Analysen durchführen kann. Notebooks unterstützen verschiedene Sprachen, darunter Python. Sie eignen sich für eine Reihe von Analysen und bieten hervorragende Möglichkeiten zur Visualisierung und Dokumentation. Eventuell stellen Sie jedoch irgendwann fest, dass Sie mehr Rechenleistung oder eine längere Laufzeit für Ihre Analysen benötigen, als ein Notebook ermöglicht. In diesem Fall können Sie alternativ einen Terra-Workflow nutzen. Diese Workflows werden in der Sprache WDL (ausgesprochen „widdle“) erstellt und genau wie Batch-Aufträge an einen beliebigen Cloud-Server übermittelt. Sie sind nützlich für rechenintensive Analysen wie Alignments oder GWAS mit umfangreichen Stichproben.
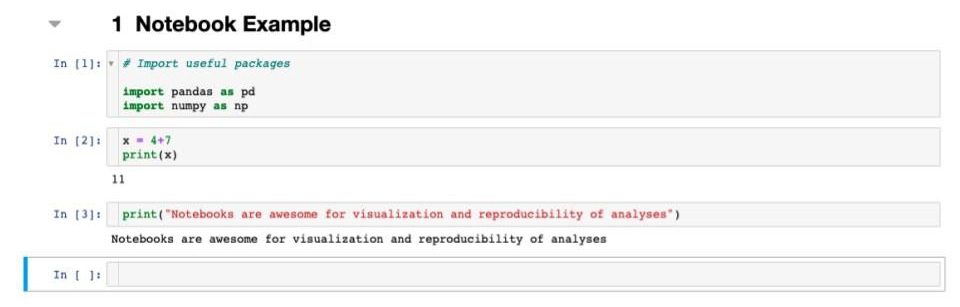
Beispiel einer Notebook-Struktur
Beispiel einer WDL-Struktur
![Ein Screenshot einer Codierungsschnittstelle für einen Workflow mit dem Titel „print-my-name“. Das angezeigte Skript ist ein einfaches Beispiel, das einen Workflow namens PrintName und eine Aufgabe namens Hallo definiert, die einen Vor- und Nachnamen als Eingabe verwendet und „Hallo [Vorname] [Nachname]!“ ausgibt.](https://gp2.org/wp-content/uploads/2022/09/03-1.jpeg)
Wenn Sie Zugriff auf GP2-Daten erhalten, erhalten Sie auch Zugriff auf die offiziellen GP2 Terra-Arbeitsbereiche. In beiden Workspaces – Tier 1 und Tier 2 – findet sich die README-Datei zur jeweils aktuellsten GP2-Datenversion (Release). Es empfiehlt sich sehr, diese zu lesen, da sie Erläuterungen zu allen Dateien aus der jeweiligen Version enthält. Schauen Sie regelmäßig in diese Workspaces, denn dort werden nach und nach neue entwickelte Code-Ressourcen, Notebooks und Workflows zu GP2-Daten eingestellt.

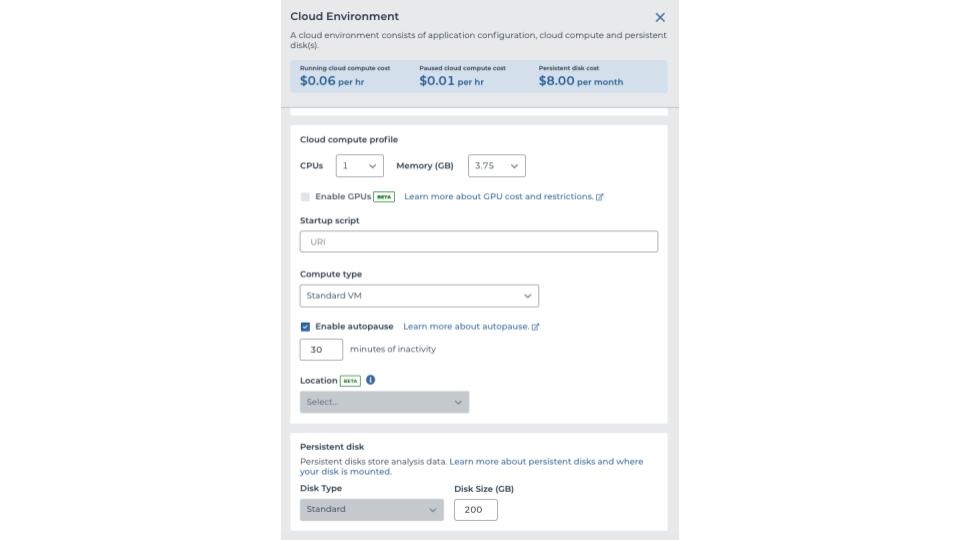
Um zu beginnen, müssen Sie eine Projektidee zur Prüfung an GP2 senden. Wird diese genehmigt, so erstellt Ihr Institut ein Abrechnungsprojekt und einen Workspace. Alternativ erledigt GP2 das in Ihrem Namen. Wenn der Workspace einmal eingerichtet ist, können Sie beginnen und ein neues Notebook für Ihre Analyse anlegen. Um ein Notebook auszuführen, müssen Sie eine Cloud-Umgebung einrichten. Diese Cloud-Umgebung können Sie nach Ihren Bedürfnissen anpassen. Wenn Sie jedoch planen, alle derzeit verfügbaren imputierten GP2-Daten zu komplexen Krankheiten zu verwenden, sollten Sie mindestens 100 GB persistente Speicherkapazität anfordern, damit Sie über genügend Platz verfügen, um mit den 82 GB Daten (ab Release 2.0) zu arbeiten. Man kann immer ein wenig mit den Optionen spielen, um zu sehen, was am besten funktioniert. Bedenken Sie aber bitte, dass mehr Ressourcen auch mit höheren Kosten verbunden sind.
Beispiel einer Cloud-Umgebung
Sobald Ihre Cloud-Umgebung läuft und ein neues Notebook eingerichtet ist, können Sie mit der Analyse beginnen. Für die Einrichtung Ihres Notebooks finden Sie weiter unten bzw. in den offiziellen GP2-Workspaces einige praktische Pakete und Variablen, die Sie nutzen können.
Nützliche Pakete
Umgebungs-Setup
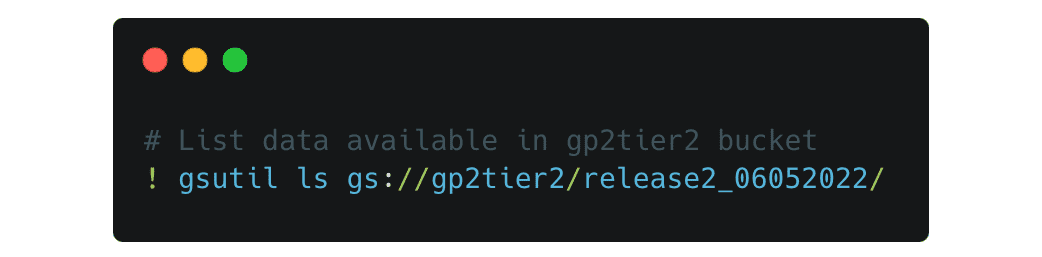
Verfügbare Daten anzeigen und Daten aus der Cloud in Ihren Arbeitsbereich übertragen können Sie mit dem Toolset gsutil. Um beispielsweise die im Bucket gp2tier2 verfügbaren Daten aufzulisten, können Sie einen Befehl wie diesen verwenden
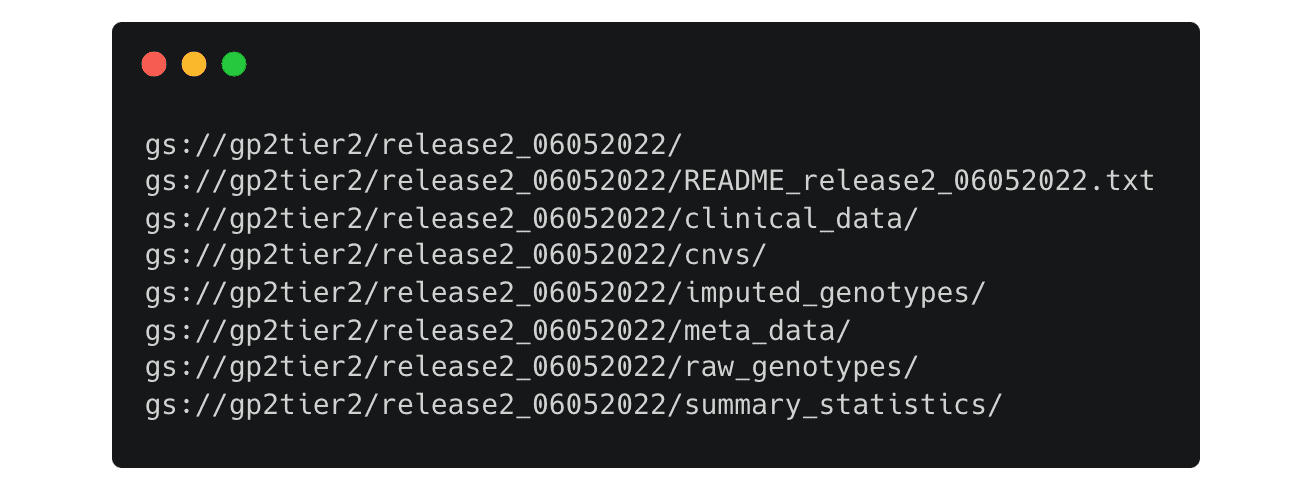
Hier werden alle verfügbaren Dateien im Release 2-Ordner aufgelistet.
Sie können die Pfade ändern, um in jedes Verzeichnis zu schauen. Wenn Sie beispielsweise den Pfad zu diesem ändern,
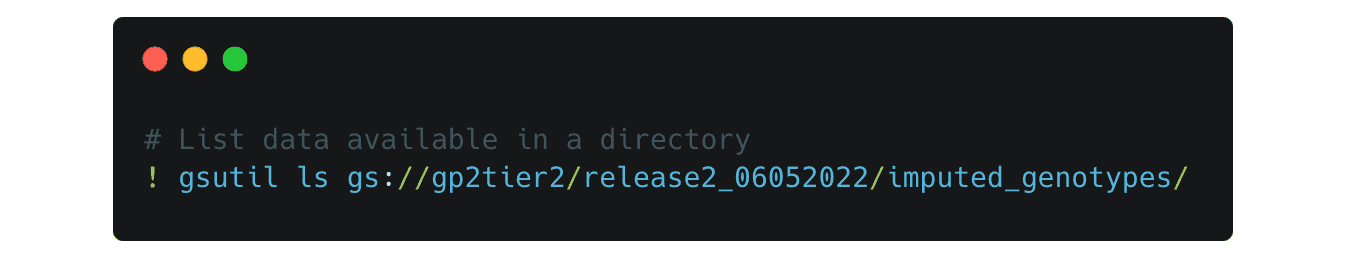
Es listet die Ordner für die imputierten Genotypdaten auf, getrennt nach der vorhergesagten Abstammung
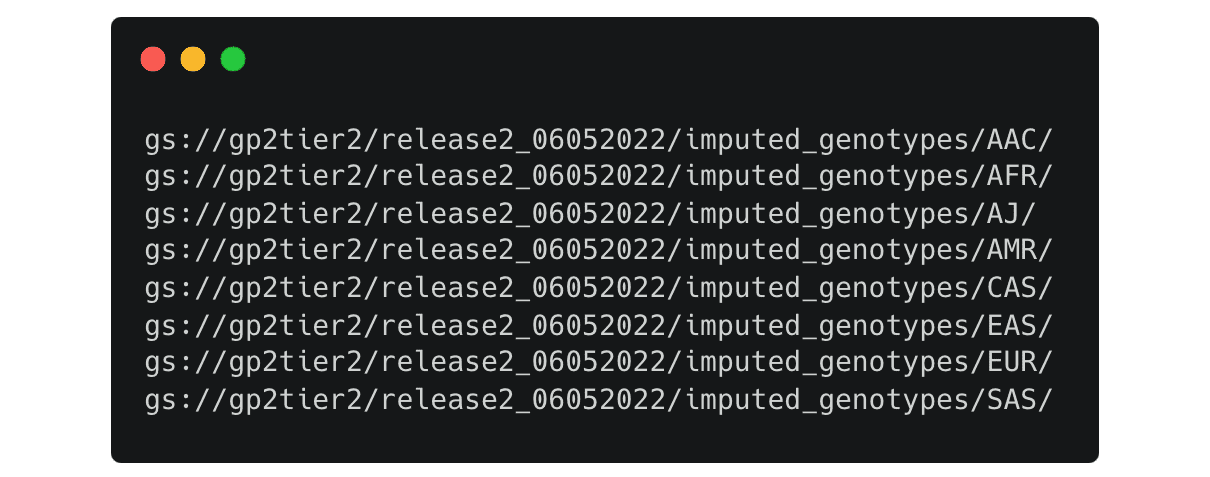
Wenn Sie sich für die benötigten Dateien entschieden haben, können Sie diese wie folgt in Ihren Arbeitsbereich kopieren:
Erstellen Sie zunächst ein Verzeichnis, in das Sie die Dateien kopieren möchten.

Kopieren Sie sie dann in Ihr neues Verzeichnis. In diesem Beispiel kopieren wir PD-Zusammenfassungsstatistiken von Nalls et al. 2019 ohne 23andMe-Daten.
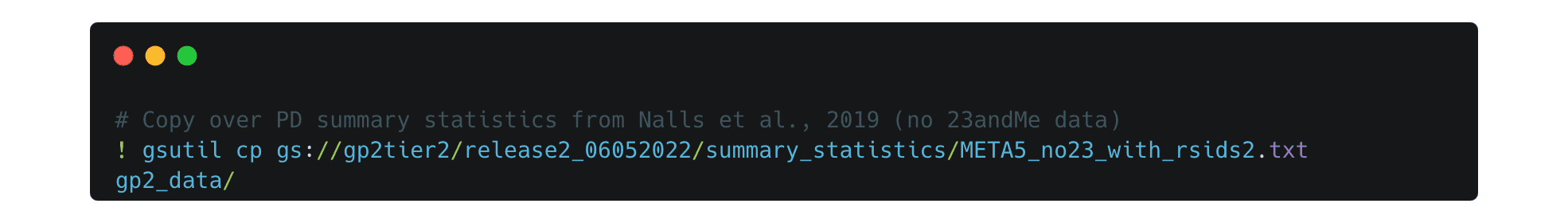
Überprüfen Sie anhand der Datei, ob alles richtig kopiert wurde:
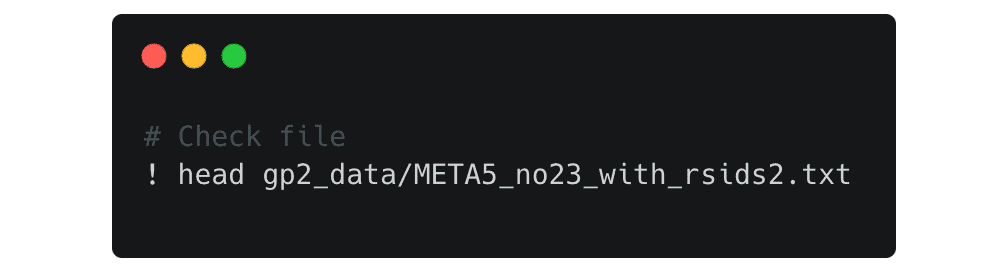

Jetzt sind Ihre Daten zur Analyse bereit!
Dies war nur ein kurzes Beispiel für das Arbeiten in Workspaces und Notebooks mit GP2-Daten. Ein ausführlicheres Terra-Tutorial finden Sie im GP2 Learning Management System. Dort können Sie Kurs 1 absolvieren: Einsatz von Terra für Datenzugriff und Durchführung von Analysen




