Présentation de l’utilisation des espaces de travail et des données du GP2

Les données issues du Programme mondial sur la génétique de la maladie de Parkinson (GP2) et du Partenariat pour l’accélération des traitements contre la maladie de Parkinson (AMP® PD) sont hébergées sur Terra, une plateforme en nuage consacrée à la bioinformatique développée par le Broad Institute avec le concours de Microsoft et Verily. Terra permet d’effectuer des analyses directement sur le nuage et élimine le besoin de télécharger les données sur votre ordinateur. Cela nous permet d’adhérer à des normes élevées de protection des données et de confidentialité et favorise la science ouverte et la reproductibilité grâce à un code et des résultats structurés et partageables. Terra prend en charge deux types d’analyse : Bloc-notes Jupyter et flux de travail WDL. Les blocs-notes sont des documents composés de cellules contenant des extraits de code qui vous permettent d’exécuter une analyse de manière séquentielle. Les blocs-notes sont compatibles avec un certain nombre de langages, dont Python, et sont utiles pour toute une série d’analyses, ainsi que pour les visualisations et la documentation. Il se peut que vous ayez besoin d’une plus grande puissance de calcul ou d’une plus longue durée d’exécution pour vos analyses qu’un ordinateur portable ne peut fournir. Dans ce cas, vous pouvez utiliser un flux de travail Terra. Ils sont construits à l’aide du langage WDL et sont proposés comme des travaux par lots sur n’importe quel serveur en nuage. Les flux de travail sont utiles pour les analyses à haute puissance telles que l’alignement et les GWAS avec des échantillons à grand effectif.
Exemple de structure de bloc-notes
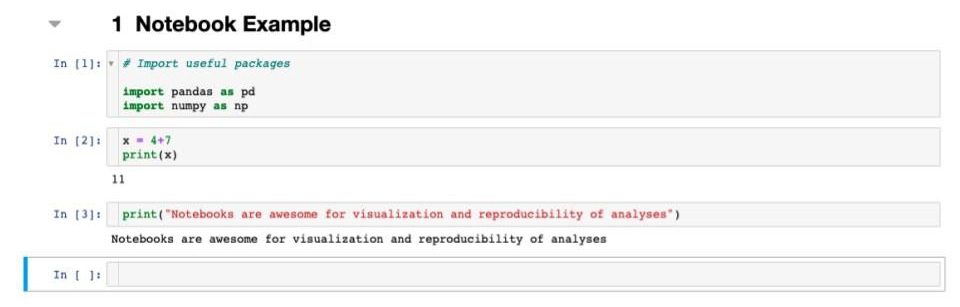
Exemple de structure de WDL
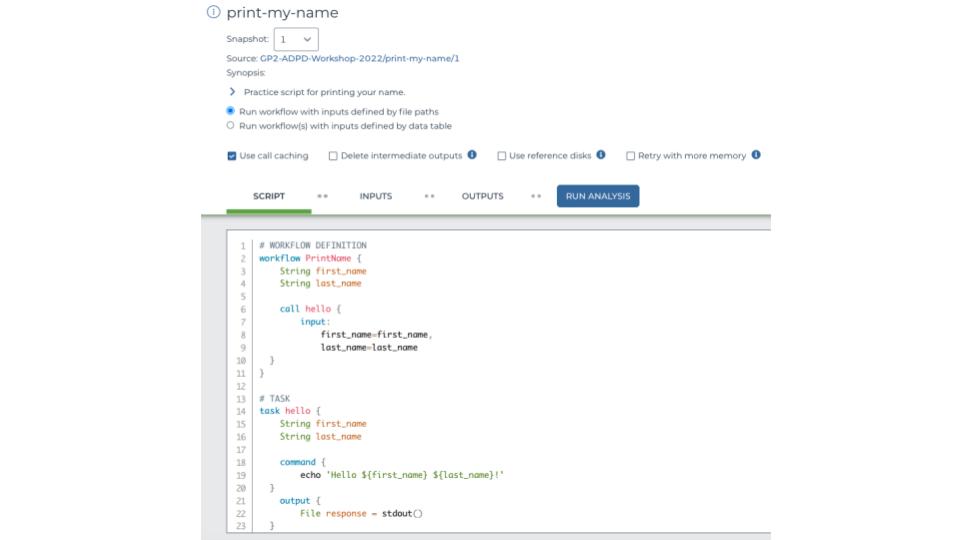 Avec l’accès au données du GP2, vous avez aussi accès aux espaces de travail Terra officiels du GP2. Les espaces de travail de niveau 1 et de niveau 2 contiennent le fichier README de la version la plus récente des données GP2. Nous vous recommandons vivement de lire ces informations car elles fournissent des explications pour tous les fichiers présents dans chaque version. Consultez régulièrement ces espaces de travail, car d’autres ressources de code, blocs-notes et flux de travail liés aux données GP2 y seront hébergés au fur et à mesure qu’ils sont développés.
Avec l’accès au données du GP2, vous avez aussi accès aux espaces de travail Terra officiels du GP2. Les espaces de travail de niveau 1 et de niveau 2 contiennent le fichier README de la version la plus récente des données GP2. Nous vous recommandons vivement de lire ces informations car elles fournissent des explications pour tous les fichiers présents dans chaque version. Consultez régulièrement ces espaces de travail, car d’autres ressources de code, blocs-notes et flux de travail liés aux données GP2 y seront hébergés au fur et à mesure qu’ils sont développés.  Pour commencer, vous devez soumettre un concept de projet au GP2 pour examen. Une fois que la demande est acceptée, votre institution mettra en place un projet de facturation et un espace de travail, ou GP2 le fera pour vous en votre nom. Dès que l’espace de travail est prêt, vous pouvez créer un bloc-notes pour vos travaux d’analyses. Pour exécuter un bloc-notes, vous devez créer un environnement en nuage. Vous pouvez personnaliser votre environnement en nuage en fonction de vos besoins, mais si vous prévoyez d’utiliser toutes les données GP2 relatives aux maladies complexes actuellement disponibles, veillez à demander au moins 100 Go d’espace disque permanent afin de disposer de l’espace nécessaire pour travailler avec les 82 Go de données, à partir de la version 2.0. Vous pouvez tester les différentes options pour trouver ce qui vous convient le mieux, mais n’oubliez pas que plus de ressources sont associées à des coûts plus élevés.
Pour commencer, vous devez soumettre un concept de projet au GP2 pour examen. Une fois que la demande est acceptée, votre institution mettra en place un projet de facturation et un espace de travail, ou GP2 le fera pour vous en votre nom. Dès que l’espace de travail est prêt, vous pouvez créer un bloc-notes pour vos travaux d’analyses. Pour exécuter un bloc-notes, vous devez créer un environnement en nuage. Vous pouvez personnaliser votre environnement en nuage en fonction de vos besoins, mais si vous prévoyez d’utiliser toutes les données GP2 relatives aux maladies complexes actuellement disponibles, veillez à demander au moins 100 Go d’espace disque permanent afin de disposer de l’espace nécessaire pour travailler avec les 82 Go de données, à partir de la version 2.0. Vous pouvez tester les différentes options pour trouver ce qui vous convient le mieux, mais n’oubliez pas que plus de ressources sont associées à des coûts plus élevés.
Exemple d’environnement en nuage
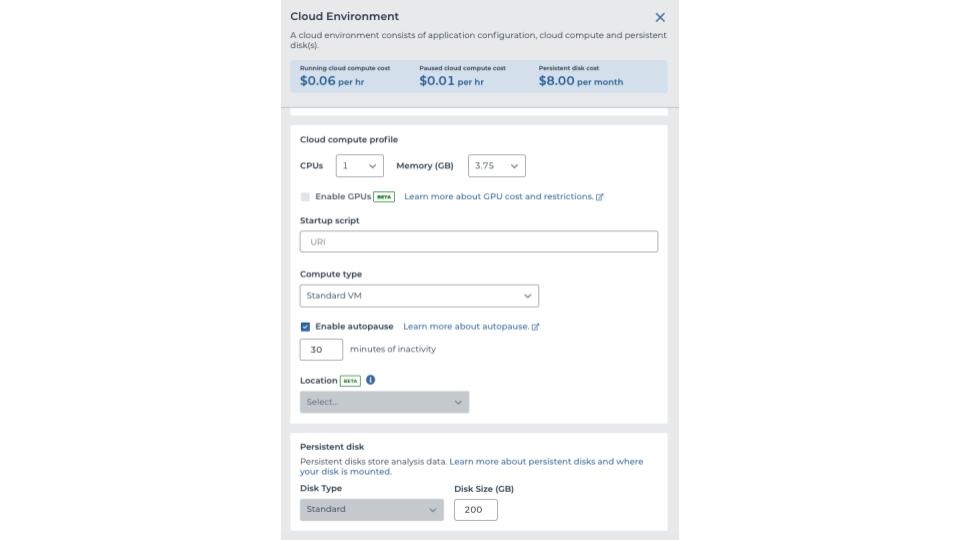 Une fois que votre environnement en nuage est opérationnel et qu’un bloc-notes a été configuré, vous êtes prêt à lancer votre analyse. Pour configurer votre bloc-notes, vous pouvez tirer parti de certains paquets et variables utiles fournis ci-dessous et sur les espaces de travail officiels du GP2.
Une fois que votre environnement en nuage est opérationnel et qu’un bloc-notes a été configuré, vous êtes prêt à lancer votre analyse. Pour configurer votre bloc-notes, vous pouvez tirer parti de certains paquets et variables utiles fournis ci-dessous et sur les espaces de travail officiels du GP2.
Paquets utiles
Configuration de l’environnement
Pour consulter les données disponibles et les transférer du cloud vers votre espace de travail, vous utiliserez le jeu d’outils gsutil. Par exemple, pour répertorier les données disponibles dans le panier gp2tier2, vous pouvez utiliser une commande comme : 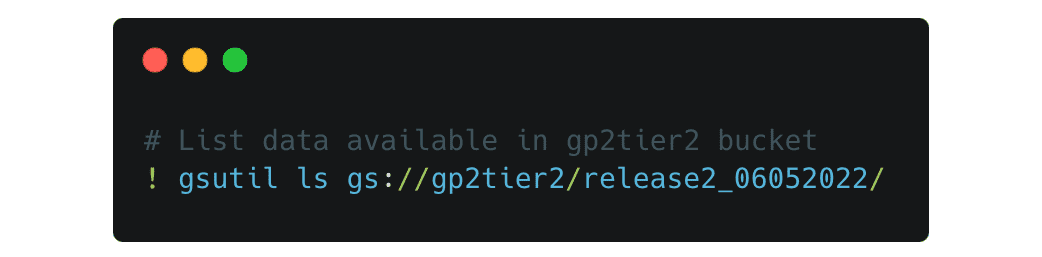 Celle-ci répertorie tous les fichiers disponibles dans le dossier de la version 2 :
Celle-ci répertorie tous les fichiers disponibles dans le dossier de la version 2 : 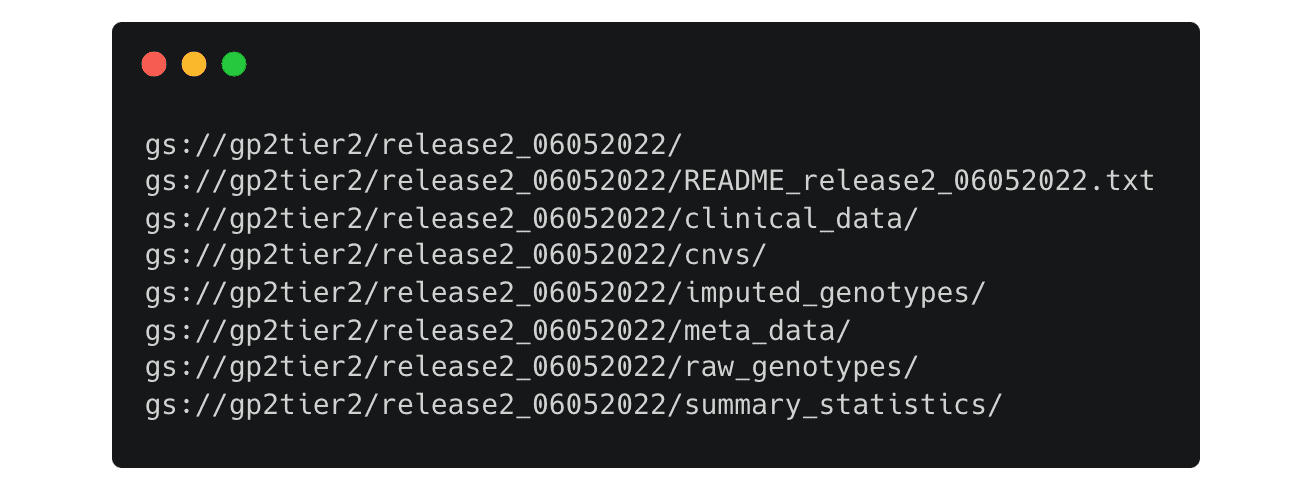 Vous pouvez modifier les chemins d’accès pour consulter chaque répertoire comme, par exemple, un chemin d’accès modifié comme suit :
Vous pouvez modifier les chemins d’accès pour consulter chaque répertoire comme, par exemple, un chemin d’accès modifié comme suit : 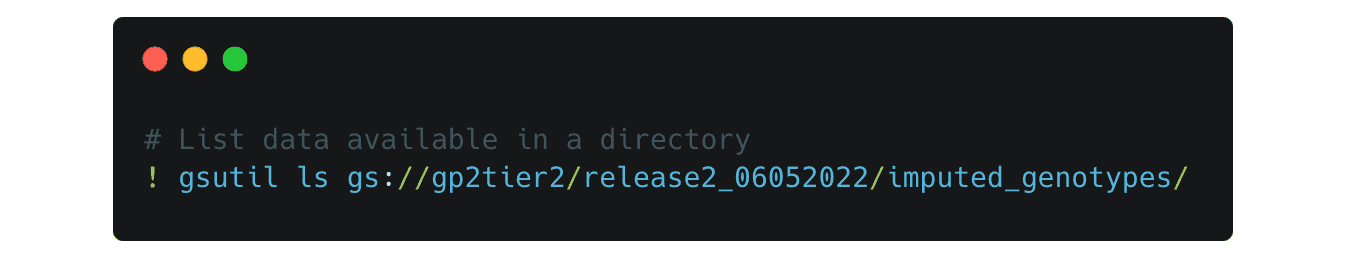 Ici, vous trouverez les dossiers pour les données de génotype imputées, séparées par l’ascendance supposée :
Ici, vous trouverez les dossiers pour les données de génotype imputées, séparées par l’ascendance supposée : 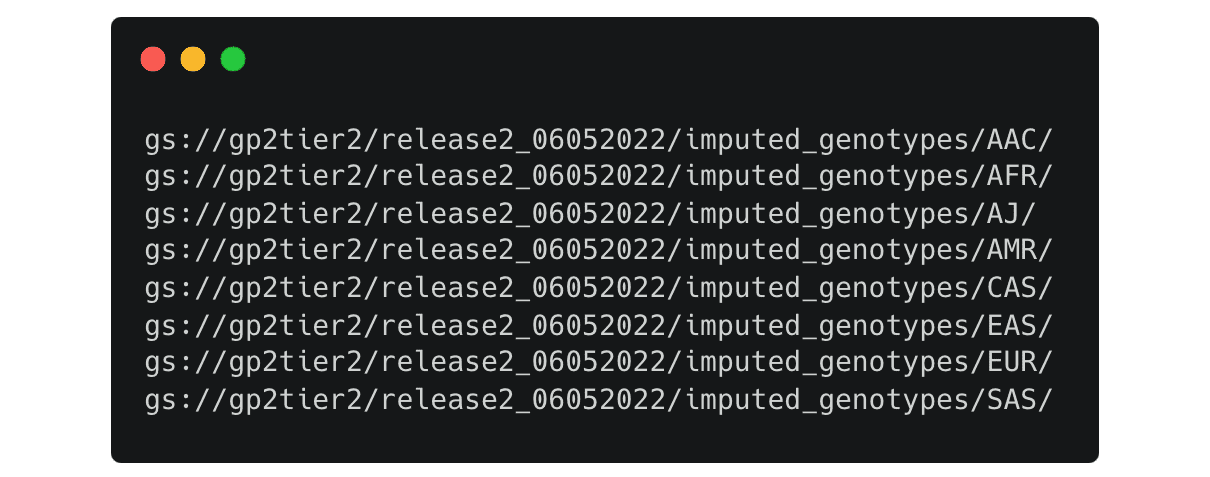 Une fois que vous avez choisi les fichiers dont vous avez besoin, vous pouvez les copier dans votre espace de travail comme ceci : Tout d’abord, créez un répertoire pour y copier les fichiers.
Une fois que vous avez choisi les fichiers dont vous avez besoin, vous pouvez les copier dans votre espace de travail comme ceci : Tout d’abord, créez un répertoire pour y copier les fichiers.  Puis, copiez-les dans votre nouveau répertoire. Dans cet exemple, nous copions les statistiques récapitulatives sur la MP de Nalls et al. 2019 sans les données 23andMe.
Puis, copiez-les dans votre nouveau répertoire. Dans cet exemple, nous copions les statistiques récapitulatives sur la MP de Nalls et al. 2019 sans les données 23andMe. 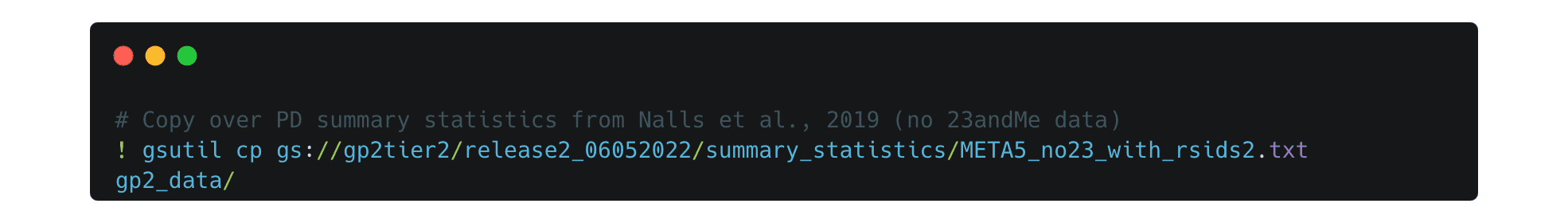 Vérifiez que tout a été copié correctement en consultant le fichier comme suit :
Vérifiez que tout a été copié correctement en consultant le fichier comme suit : 
 Vos données peuvent être analysées.
Vos données peuvent être analysées.
Ceci n’est qu’un exemple succinct de l’utilisation des espaces de travail et des blocs-notes avec les données GP2. Pour un tutoriel détaillés sur Terra, veuillez consulter le système de gestion de l’apprentissage du GP2 et suivre le « Cours Course 1 » : Utilisation de Terra pour accéder aux données et effectuer des analyses





