Resumen
En diciembre de 2024, el GP2 anunció su novena publicación de datos en las plataformas Workbench de Terra y Verily® en colaboración con AMP® PD. Esta edición incluye 17,690 participantes genotipados nuevos.
- Los datos genotipados, que incluyen muestras localmente restringidas, corresponden ahora a un total de 71,835 participantes genotipados (31,985 con EP; 18,249 de control y 21,601 «otros» fenotipos).
- Si restamos las muestras localmente restringidas, el total asciende a 55,305 muestras (23,709 con EP; 13,404 de control y 18,192 «otros» fenotipos)
- Estas 71,835 muestras con datos genotipados incluyen:
- 16,800 individuos con información de fenotipado clínico profundo (aportación de datos 8)
- 10,454 individuos con información exómica clínica (aportación de datos 8)
- 7,732 individuos con datos de genoma completo (aportación de datos 8)
¿Qué hay de nuevo en esta aportación de datos?
- Sobre los datos nuevos:
- La aportación 9 incluye el genotipado de 17,690 participantes nuevos.
- Para los investigadores que prefieren datos brutos revertidos y alineados, proporcionamos genotipos_brutos_revertidos además de lo genotipos_brutos.
- Sobre los identificadores de muestras:
- Ya no usamos el prefijo «m-» para denotar a las cohortes originalmente reclutadas a través de la red monogénica.
- Dejaremos de usar el sufijo «-s*» en la nomenclatura de muestras del GP2. El número de muestra sigue estando disponible a través de la clave maestra para facilitar el cruce de datos con los ID de aportaciones anteriores, pero los ID de muestras del GP2 ya no incluirán el sufijo «-s*» en los archivos genéticos.
- Se han actualizado los PPMI GP2ID en todos los archivos, y ahora incluyen su PATNO ID para facilitar su identificación a los investigadores que trabajan con varias plataformas.
- La aportación 9 incluye el genotipado de 17,690 participantes nuevos.
Muestras localmente restringidas sujetas al RGPD, vía Verily Viewpoint Workbench Seguimos haciendo pruebas para brindar acceso a muestras localmente restringidas; es decir, muestras sujetas al Reglamento General de Protección de Datos (RGPD), gracias a nuestra colaboración con Verily Viewpoint Workbench. Por ahora, y mientras el GP2 sigue desarrollando soluciones para la compartición de datos protegidos por el RGPD, los datos de la novena aportación con restricciones regionales solo están disponibles para miembros y socios del consorcio del GP2. A medida que las pruebas y la implementación sigan avanzando en 2024, esta solución estará también disponible para la comunidad de investigación en general. Todas las muestras de la novena aportación pueden encontrarse en Workbench, mientras que todas las muestras de la novena aportación no sujetas a los requisitos del RGPD pueden encontrarse en el Workbench comunitario de Terra (como en las publicaciones anteriores). Para obtener acceso a la aportación completa en VWB, usted debe:
- Contar con acceso de nivel 2 autorizado por el GP2
- Llenar el formulario de solicitud para muestras sujetas al RGPD
- Ser miembro del consorcio del GP2 (contribuir cohortes, ser socio del GP2 o ser miembro de un equipo de análisis de proyectos)
Datos clínicos Esta aportación contiene datos clínicos de un total de 71,835 participantes de quienes disponemos de datos genéticos y datos clínicos principales. Esta edición incluye datos clínicos fenotipados profundos y datos genéticos de 16,800 participantes. Esta información contiene los siguientes datos:
- Edad en el momento del diagnóstico y de la aparición de síntomas
- Diagnóstico principal, actual y más reciente
- Exámenes cognitivos, como el Miniexamen Cognoscitivo (MMSE) y la Evaluación Cognitiva de Montreal (MoCA)
- Revisión patrocinada por la Movement Disorder Society de la Escala de Valoración Unificada de la Enfermedad de Parkinson (MDS-UPDRS)
- «Otros» fenotipos detallados, como la demencia de cuerpos de Lewy (LBD)
- Los casos reclutados por la Red monogénica están clasificados como «otros»
Datos a nivel individual Ahora recopilamos datos de un total de 104 cohortes. Consulte el Panel de cohortes del GP2 para obtener más información sobre las cohortes compartidas. La ascendencia determinada genéticamente de los participantes genotipados del GP2 se divide en once grupos. La tabla siguiente presenta la ascendencia determinada genéticamente de los participantes genotipados de esta aportación de datos que pasaron el control de calidad y se imputaron. Estos números reflejan muestras de aportaciones anteriores, reagrupadas en el archivo clúster actualizado y sujetas a control de calidad. También incluyen las nuevas muestras genotipadas, que son exclusivas de la presente aportación de datos. 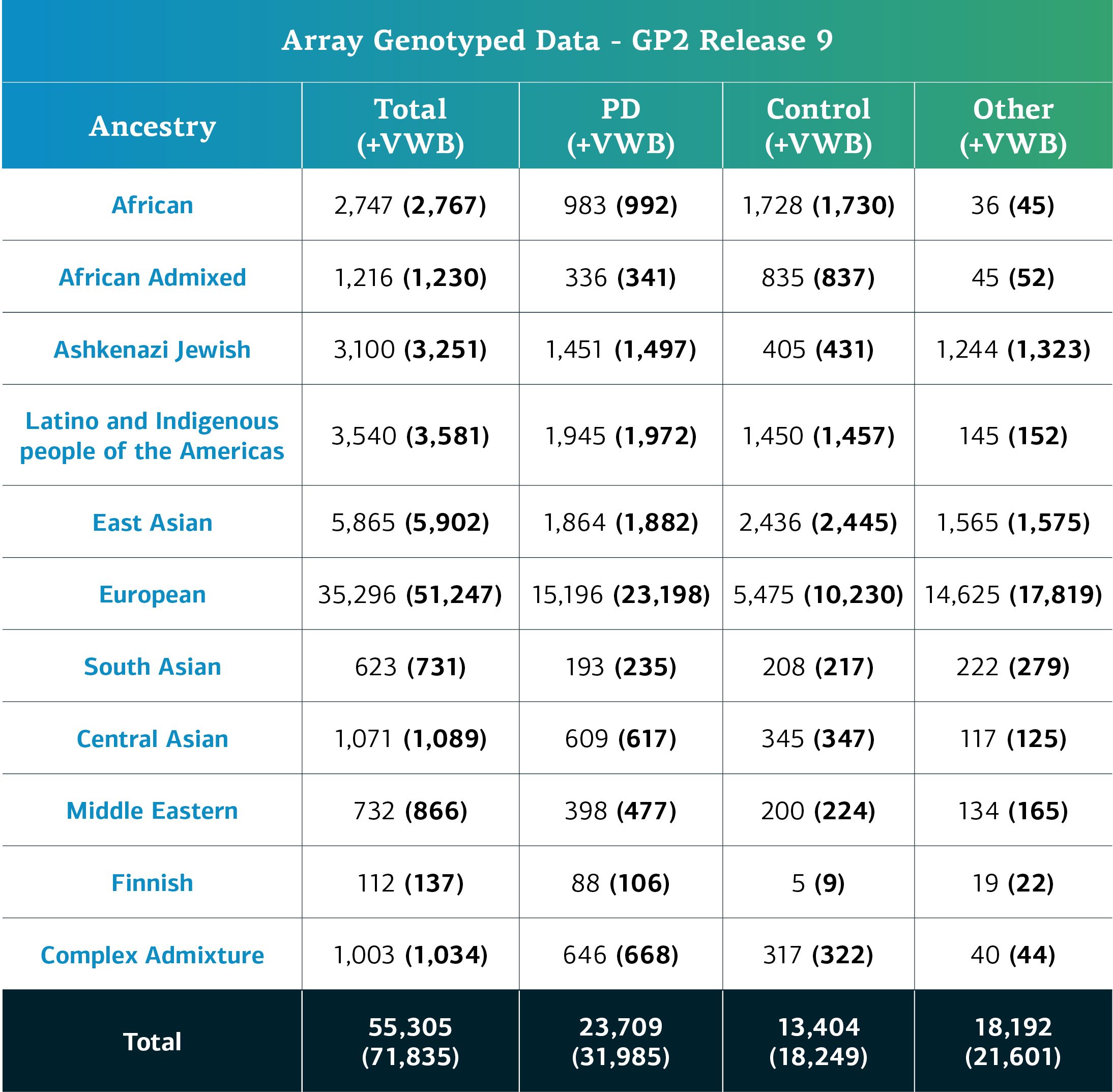 En futuras aportaciones, seguiremos ampliando la diversidad de los participantes. Los invitamos a visitar nuestro panel para conocer nuestro progreso. Los usuarios que ya tengan acceso de nivel 2 pueden explorar los datos en más profundidad en nuestro navegador de cohortes, que presentamos en un artículo de blog anterior. Como siempre, ¡consulte el README que acompaña cada aportación de datos del GP2 para obtener más detalles sobre recomendaciones de control de calidad, pipelines, datos y análisis!
En futuras aportaciones, seguiremos ampliando la diversidad de los participantes. Los invitamos a visitar nuestro panel para conocer nuestro progreso. Los usuarios que ya tengan acceso de nivel 2 pueden explorar los datos en más profundidad en nuestro navegador de cohortes, que presentamos en un artículo de blog anterior. Como siempre, ¡consulte el README que acompaña cada aportación de datos del GP2 para obtener más detalles sobre recomendaciones de control de calidad, pipelines, datos y análisis!





