في شهر تشرين الأول 2022، أعلن البرنامج العالمي حول الجينات المرتبطة بداء باركنسون GP2 عن صدور الدفعة الثالثة من البيانات على منصة تيرا بالتعاون مع AMP® PD. تتميّز هذه الدفعة بأنها تزخر بالبيانات والموارد الجديدة، وتشمل عدداً كبيراً من العيّنات يصل إلى ضعفي ما احتوت عليه الدفعة السابقة تقريباً.
تضّم هذه الدفعة 6330 مشاركاً جديداً في فئة المرض المعقد، إذ تمت إضافة هؤلاء إلى الدفعات السابقة من شبكتي المرض المعقد وأحادي الجينة. تحتوي بيانات المرض المعقد حالياً على 14902 مشاركاً تم تصنيفهم حسب النموذج الوراثي بالمجمل (8190 فرداً من مرضى الباركنسون و6712 فرداً من غير المرضى). تالياً قائمة بالمجموعات الجديدة المشاركة في هذه الدفعة:
- الدراسة الأسترالية حول الجينات المرتبطة بداء باركنسون (APGS)
- دراسة فوكس لاكتشاف المؤشرات الحيوية الجديدة (BioFIND)، وهي دراسة سريرية رصدية لداء باركنسون
- مواقع مشاركة من «الروابط بين ذوي البشرة السمراء والأمريكيين من أصول أفريقية وداء باركنسون» (BLAACPD-KPM، وBLAACPD-RUSH، وBLAACPD-UAB وBLAACPD-UC)، وهي مجموعة من الأفراد ذوي البشرة السمراء والأمريكيين من أصول أفريقية ممّن يعانون من داء باركنسون.
- مجموعة مشاركي جامعة كوتش في GP2، وهي مجموعة مشاركة من تركيا
- ائتلاف مجموعة LRRK2، وهي مجموعة من مرضى الباركنسون الذين يحملون طفرة في جين LRRK2
- النماذج الوراثية والأنماط الظاهرية لاضطرابات الحركة – كلية كينغز في لندن (MDGAP-KINGS)، وهي مجموعة من بنك أدمغة يتخذ من المملكة المتحدة مقراً له
- برنامج نيوزيلاندا حول داء باركنسون (NZP3)
- دراسة جينات داء باركنسون وبيئته (PAGE)
- مبادرة مؤشرات ترقي داء باركنسون (PPMI)
- الدراسة المنهجية لعينات سينوكلين (S4)، وهي دراسة تبحث في المؤشرات الحيوية لداء باركنسون وتحديداً بروتين الفا سينوكلين
تنقسم سلالات المشاركين في دراسة المرض المعقد في GP2، والتي تم تحديدها وفقاً للجينات، إلى عشر مجموعات (المجموعات التسعة المذكورة أدناه بالإضافة إلى عدد قليل من الأوروبيين الفنلنديين)، ويوضح الجدول أدناه خضوع السلالات المُحدّدة بحسب جينات المشاركين في دراسة المرض المعقد في هذه الدفعة لمعايير ضبط الجودة وتحديد النسب. تشمل هذه الأرقام عينات من إصدارات سابقة تمت إعادة تجميعها في فئات باستخدام ملف التجميع الجديد كما تم فحصها لضبط الجودة بالإضافة إلى العينات المتفردة لهذا الإصدار والتي تم تصنيفها وراثيا ومشاركتها في الإصدار الحالي. 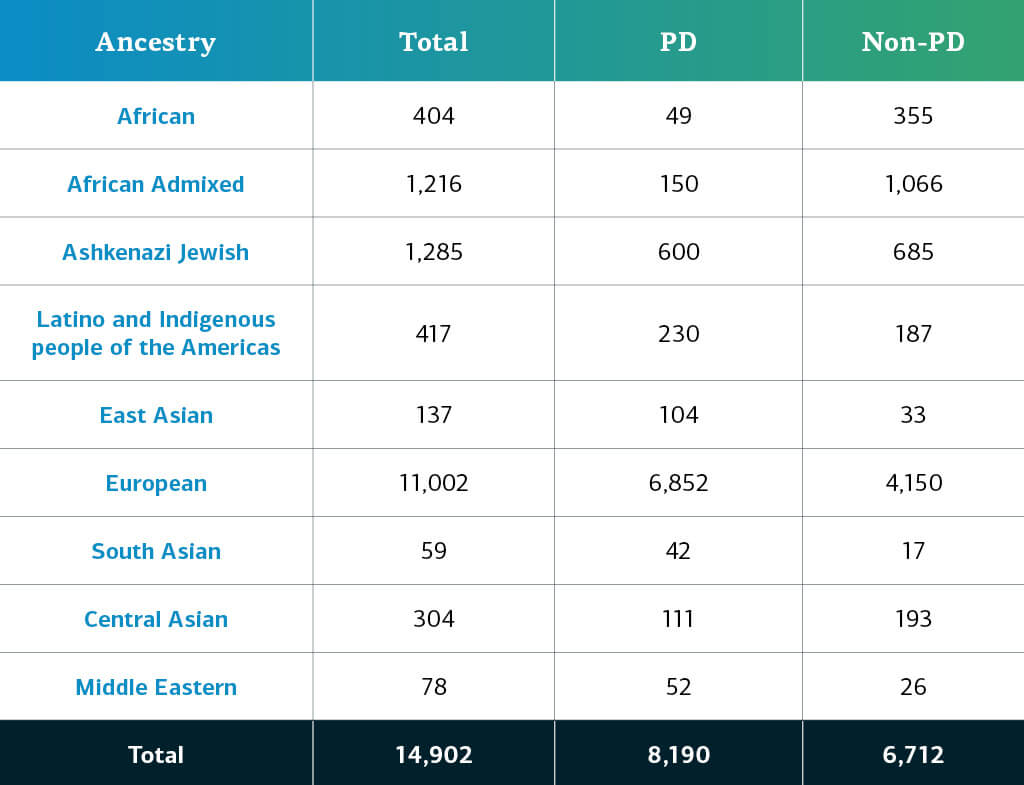 سوف نستمر في تعزيز التنوع في مجموعات المشاركين في الإصدارات المستقبلية من البيانات. يمكنكم الإطلاع على لوحة التحكم لمواكبة التقدم المحرز.
سوف نستمر في تعزيز التنوع في مجموعات المشاركين في الإصدارات المستقبلية من البيانات. يمكنكم الإطلاع على لوحة التحكم لمواكبة التقدم المحرز.
يتمثل الهدف الأساسي من إطلاق هذه المجموعة من البيانات في شمول المزيد من العينات التي تم استخلاص بيانات أولية منها بالإضافة إلى المزيد من البيانات الوصفية السريرية والجينومية. تم تجميع هذه البيانات باستخدام ملف تجميع خاص بـGP2 للنماذج الوراثية (متوفر في قسم «الأدوات المفيدة» في المستويين الأول والثاني من الوصول إلى البيانات). ويعتمد ملف التجميع هذا على 2793 عينة من ست سلالات مختلفة تعكس تنوع السلالات الجينية المكوّنة لهذه الدفعة من بيانات GP2 بالإضافة إلى 420 حالة من حالات مرض غوشر التي تمت إضافتها لتعكس بشكل أفضل المتغيرات ذات الأهمية في جين GBA الذي يشكل خطراً. يمكن تحميل ملف التجميع عبر مستودع GP2 GitHub إذا كنتم ترغبون في تطبيقه على بيانات خارج إطار GP2.
قمنا أيضا بتحديث استدعاءات متغير رقم النسخة (CNV) لكل العينات التي تم تصنيفها وراثيا والتي نجحت في اختبار ضبط الجودة (مستوى الجين +250kb مناطق متاخمة). يشير متغير رقم النسخة (CNV) إلى التغير في عدد المرات التي يتم فيها تكرار قطعة معينة من الحمض النووي الريبوزي منقوص الاكسجين (الدنا). هذا التغير يمكن أن يتأتى من خلال الشطب، الإضافة، أو أي أحداث أخرى ويمكن له أن يوفر مزيدا من المعلومات حول الكيفية التي يؤثر فيها التغيير الهيكلي على خطر الإصابة بالمرض. يمكن العثور على المسار المستخدم لإنتاج احتمالات استدعاءات متغير رقم النسخة (CNV) هنا GP2 Github. يتم تصنيف هذه النتائج حسب السلالات الجينية في مساحة العمل في المستوى الثاني tier 2 access workspace. احتمالات CNVs هي نقطة بداية ممتازة لتصنيف العينات حسب الأولوية بناء على احتمال حدوث إضافة، حذف، أو تكرار في الجينات المهمة وذلك لأغراض دراسات استكمالية وتحليلات إضافية.
يمكن الاطلاع على مزيد من المعلومات حول هيكلية النموذج الوراثي للمرض المعقد والبيانات السريرية في المدونة التالية «مكونات الدفعة الأولى من بيانات GP2» بالإضافة إلى README الذي تم تحديثه لهذا الإصدار ومتاح على الموقع الرسمي لمساحات عمل تيرا على موقع GP2. تجدون بيانات تسلسل الجينوم الكامل لداء باركنسون أحادي الجينة مفصلة في نفس ملف README. يمكن أن تتوفر مزيدا من المعلومات حول تسلسل الجينوم الكامل لأحادي الجينة في الدفعة المقبلة.
لمحة عما سوف تشتمل عليه الدفعة المقبلة من البيانات (نستهدف موسم الأعياد، الربع الأول من عام 2023)
- المزيد من بيانات النموذج الوراثي
- المزيد من بيانات تسلسل الجينوم الكامل
- المزيد من البيانات الوصفية السريرية
- تحديث شيفرات وأدوات التحليل
- ملخصات إحصائية إضافية لدراسات ارتباط الجينات ببعض الأمراض GWAS بخصوص المخاطر والعمر لدى بدء الإصابة بالمرض.
تمت كتابة هذه المدونة من قبل أعضاء مجموعة عمل تحليل بيانات المرض المعقد في GP2: هامبتون ليونارد، مايك نولز، دان فيتالي وماري مكاريوس، ماثيو كوريتسكي من المعاهد الوطنية للصحة، كريستين ليفين من داتا تكنيكا الدولية/ المعاهد الوطنية للصحة، وزي-هوا فونغ وبيتر هويتنك، أعضاء مجموعة عمل تحليل البيانات – منصة أحادي الجينة.