لمحة عامة
في ديسمبر/كانون الأول 2023، أعلن البرنامج العالمي حول الجينات المرتبطة بداء باركنسون عن إطلاق الدفعة السادسة من البيانات على منصتي terra وverily® workbench بالتعاون مع منصة AMP® PD. تضّم هذه الدفعة أكثر من 20 ألف مشارك جديد، إضافة إلى الدفعات السابقة من شبكتي المرض المُعقَّد وأحادي الجين.
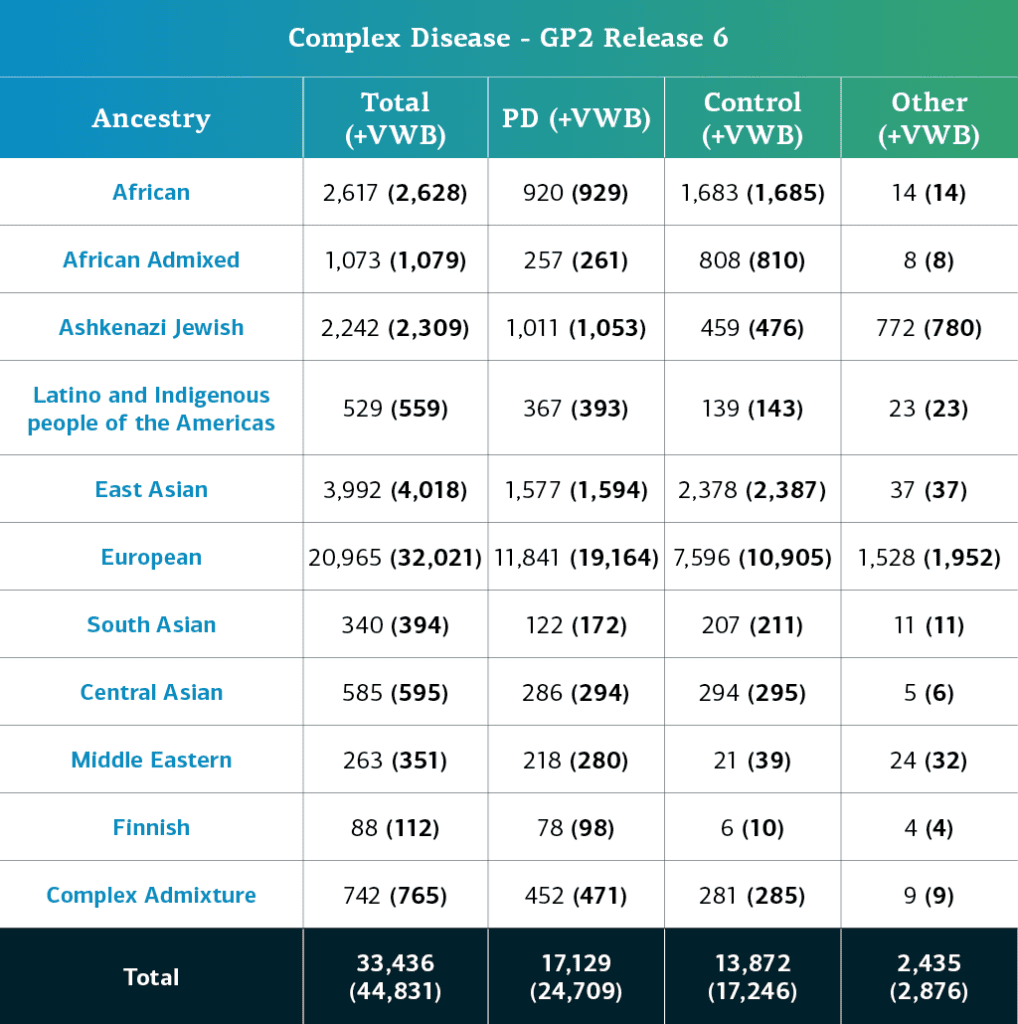
- تحتوي قاعدة بيانات المرض المُعقَّد (الأنماط الوراثية) حاليا، بما في ذلك العينات المقيدة محليا، على 44831 مشاركاً تم تصنيفهم حسب النموذج الوراثي بالمجمل (24709 فرداً من مرضى باركنسون و17246 فرداً من مجموعة الضبط و2876 فرداً من تصنيفات «أخرى»).
- بعد استبعاد العينات المقيدة محليا، تضم هذه البيانات 33436 مشاركاً (17129 فرداً من مرضى باركنسون و13872 فرداً من مجموعة الضبط و2435 فرداً من تصنيفات «أخرى»)
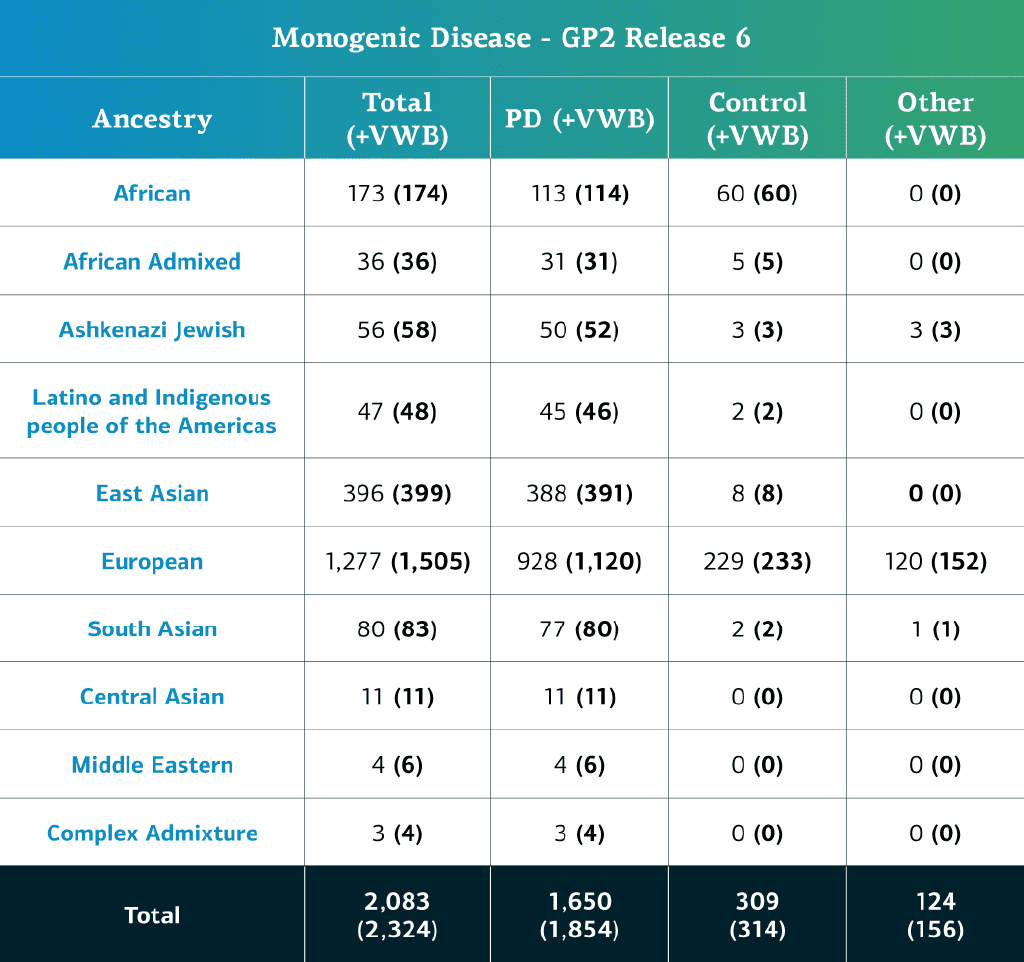
- تضم بيانات المرض أحادي الجين (تسلسل الجينوم الكامل) اليوم ما مجموعه 2324 مشاركاً تم ترتيبهم وفقاً لهذا التسلسل (1854 فرداً من مرضى باركنسون و314 فرداً من مجموعة الضبط و156 فرداً من تصنيفات «أخرى»).
- بعد استبعاد العينات المقيدة محليا، تضم هذه البيانات 2083 مشاركاً (1650 فرداً من مرضى باركنسون و13872 فرداً من مجموعة الضبط و2435 فرداً من تصنيفات «أخرى»)
- تتوفر لدينا معلومات جينية عن 12585 فرداً ممن تتوفر عنهم معلومات حول السمات النمطية السريرية العميقة
ما الجديد في هذه الدفعة؟
- عيّنات جديدة من المرض المُعقَّد (مصنفة بحسب النموذج الوراثي) والمرض أحادي الجين (الجينوم الكامل)
- إتاحة الوصول لعيّنات مقيدة محليا بواسطة Verily Viewpoint Workbench
- طرح البيانات السريرية لأكثر من 12 ألف فرد
- طرح سلالة جديدة – تاريخ مختلط مُعقَّد (CAH)
- مستجدات إجراءات ضبط الجودة للبيانات المنشورة والمصنفة بحسب النموذج الوراثي
- مستجدات التعرف على المتغيرات باستخدام DeepVariant في الوقت الحالي وتطبيقها على بيانات الجينوم الكامل المنشورة
مستجدات ضبط الجودة
باختصار، تُنفّذ GenoTools (v1.0.0) خطوات ضبط الجودة التالية: التضارب في معلومات الجنس، تشذيب معدل استدعاء المتغيرات الجينية Call rate، والتحقق من عدم وجود نسخ مكررة، والتحقق من وجود أفراد تربط بينهم صلة قرابة والإبلاغ عنهم وتدقيق معدل تجانس اللواقح.
بخلاف الدفعات السابقة، لم نعُد ننفّذ عملية الغربلة على مستوى المتغيرات التالية: وتيرة تكرار الأليل الثانوي (MAF) وهاردي-فينبيرغ (HWE) أو تعداد الأليل الثانوي (MAC). إذا كنتم ترغبون بغربلة البيانات بنفس الطريقة التي اتبعناها في الدفعات السابقة، نوصيكم بمراجعة ملف README الخاص بكل دفعة للحصول على معلومات تفصيلية والحدود الدنيا المقترحة.
إن مجموعة «التاريخ المختلط المُعقَّد»
، أو المعروفة اختصاراً باسم CAH، هي سلالة جديدة تم طرحها للدفعة السادسة من بيانات GP2. وقد أنشئت هذه المجموعة في محاولة لتفادي الخطأ الذي حدث في الدفعة الخامسة من البيانات، حيث تم نسب عدد كبير من العيّنات، التي تعود لأفراد من جنوب أفريقيا وغيرهم من الأفراد ذوي الأصول المختلطة إلى حدٍ كبير، إلى سلالة آسيا الوسطى (CAS) بشكلٍ خاطئ. وفيما يخص الدفعة السادسة، فإن مجموعة سلالة «التاريخ المختلط المُعقَّد» تحتوي بشكلٍ رئيس على عينات من جامعة ستيلينبوش (كيب تاون، جنوب أفريقيا)، ومعهد كورييل (كامدن، ولاية نيوجيرسي الأمريكية) ومؤسسة باركنسون (ميامي، ولاية فلوريدا الأمريكية). نعتبر أي عيّنة تحمل وصف (CAH) مختلطة إلى حد كبير جداً، بحيث لا يمكن شمولها مع غيرها من مجموعات سلالات GP2 في التحاليل.
العيّنات المقيدة محليا بموجب قانون حماية البيانات العامة مُتاحة بواسطة Verily Viewpoint Workbench
يسعدنا أن نُعلن أن بعض المستخدمين سيتمكنون من الوصول إلى العيّنات المقيدة محليا، والتي تُعرف بالعيّنات الخاضعة لسياسة قانون حماية البيانات العامة، وذلك من خلال تعاوننا مع Verily Viewpoint Workbench. يخلق Workbench بيئة آمنة لتنظيم البيانات الطبية الحيوية وتحليلها، حيث يهدف إلى تعزيز التشاركية في الأبحاث والقدرة على إعادة إنتاج البيانات بواسطة دمج النظم السحابية. ويدعم مشاركة مساحات العمل، بما في ذلك شيفرتا Python وR، ويقدم مجموعة من الخدمات السحابية الأصلية لإدارة البيانات وتحليلها. إلى جانب ما سبق، يدعم ًWorkbench عملية دمج البيانات الآمنة والتحقق من البيانات حسب الطلب، ما يؤهله لاستضافة بيانات GP2 المقيدة محليا، بصفته بيئة بحثية مثالية وآمنة وقابلة للتطوير.
في هذا الوقت، وبينما يستمر برنامج GP2 في تنفيذ حلول مشاركة البيانات على البيانات المحميّة بموجب قانون حماية البيانات العامة، سوف تكون الدفعة السادسة متوفرة حصراً لأعضاء ائتلاف برنامج GP2 وشركائه. ومع استمرار اختبار هذه الحلول وتنفيذها في بداية عام 2024، سوف تكون هذه الحلول متوفرة لمجتمع الباحثين الأوسع في وقتٍ لاحق. يمكن إيجاد كل عيّنات الدفعة السادسة على ًWorkbench بينما توجد كل عيّنات الدفعة التي لا تخضع لقانون حماية البيانات العامة على مساحة العمل الجماعية على Terra (تماماً مثل كل الدفعات السابقة). حتى تتمكن من الدخول إلى الدفعة الكاملة علىVerily Viewpoint Workbench، يجب أن:
- يكون لديك تصريح للدخول إلى المستوى الثاني من بيانات GP2
- تكون عضواً في ائتلاف GP2 (مساهم بمجموعة من المشاركين، أو شريك لـGP2 أو عضو في فريق إجراء التحاليل)
- تملأ نموذج طلب عيّنة خاضعة لقانون حماية البيانات العامة. وبعد الانتهاء من تعبئة النموذج، سوف تصلك رسالة بتعليمات الدخول إلى Workbench.
البيانات السريرية
نحن متحمسون أيضاً للإعلان عن احتواء هذه الدفعة على تصنيف سريري عميق وشامل لبيانات 12585 فرداً بحسب الأنماط الظاهرية. وتشمل هذه المعلومات:
- العمر عند التشخيص وبداية المرض
- التشخيصات الأولية والحالية والأحدث
- الاختبارات الإدراكية مثل اختبار الحالة العقلية المُصغَّر (MMSE) وتقييم مونتريال الإدراكي (MoCA)
- مراجعة مقياس تصنيف داء باركنسون الموحد برعاية جمعية اضطرابات الحركة (MDS-UPDRS)
- أنماط ظاهرية «أخرى» مُفصّلة، مثل خرف أجسام ليوي
كل فرد من الأفراد البالغ عددهم 12585 لديه في هذه الدفعة معلومات سريرية ترتبط بمعلومات جينية أيضاً.
بيانات على المستوى الفردي
نعمل في الوقت الحالي على جمع البيانات من 74 مجموعة من المشاركين، حيث تُعدّ 46 مجموعة منها جديدة وتُعرض للمرة الأولى في هذه الدفعة. يُرجى مراجعة لوحة متابعة معلومات المشاركين في GP2 للمزيد من المعلومات حول مجموعات المشاركين التي تمت مشاركتها.
تنقسم سلالات المشاركين في دراسة المرض المُعقَّد في GP2، والتي تم تحديدها وفقاً للجينات، إلى11 مجموعة. ويوضح الجدول أدناه خضوع السلالات المُحدّدة بحسب جينات المشاركين في دراسة المرض المُعقَّد في هذه الدفعة لمعايير ضبط الجودة وتحديد النسب. تشمل هذه الأرقام عيّنات من دفعات سابقة تمت إعادة تجميعها في فئات باستخدام ملف التجميع الجديد وتم فحصها لضبط الجودة، بالإضافة إلى العينات المتفردة لهذه الدفعة والتي تم تصنيفها وراثياً ومشاركتها في الدفعة الحالية.
تحديد المتغيرات في التسلسل الكلي للجينوم باستخدام DeepVariant-GLnexus
في هذه الدفعة، مقارنة مع الإصدارات السابقة، قمنا باستخدام Google’s DeepVariantكمسار بمصاحبة GLnexus لتحديد المتغيرات على مستوى المجموعات المشاركة. DeepVariant هو أداة لتحديد المتغيرات يستند إلى التعلّم المتعمق ويتفوق على أحدث الأدوات التي تقوم بتحديد المتغيرات الجينية على المستوى الفردي بشكل دقيق. كما يُسهّل العملية ويُعزّز الدقة والمصداقية.
سوف نعمل على تعزيز التنوع في مجموعات المشاركين في الدفعات القادمة من البيانات. يمكنكم الإطلاع عللوحة متابعة معلومات المشاركينلمواكبة التقدم المحرز. . يُمكن للمستخدمين الذين يملكون إذن الدخول إلى المستوى الثاني من البيانات التعمق في هذه البيانات على متصفح المجموعات المشاركة، والتي تم الحديث عنها بشكل موسع في مدونة سابقة.
وكالعادة، يُرجى العودة إلى ملف README الذي يرافق كل دفعة من بيانات GP2 للمزيد من التفاصيل حول المسارات، والبيانات والتحليلات.











