Aperçu général
En décembre 2024, le GP2 a annoncé la neuvième édition des données sur les plateformes Terra et Verily®Workbench , en collaboration avec l’AMP® PD. Cette publication comprend plus de 17 690 participants supplémentaires au génotype répertorié.
- L’éventail de données de génotypes répertoriés, y compris les échantillons strictement locaux, atteignent désormais un total de 71 835 participants à génotype répertorié (dont 31 985 atteints de Parkinson, 18 249 cas témoins et 21 601 « autres » phénotypes).
- Si l’on soustrait les échantillons strictement locaux, le nombre total s’élève à 55 305 cas (soit 23 709 cas de Parkinson, 13 404 cas témoins et 18 192 « autres » phénotypes).
- Sur ce total de 71835 échantillons aux données à génotype répertorié :
- 16 800 personnes présentent également des informations exhaustives sur le phénotypage clinique (8e édition)
- 10 454 personnes présentent également des informations sur les exomes cliniques (8e édition)
- 7 732 personnes présentent également des données AGE (8e édition)
Quoi de neuf dans cette nouvelle édition des données ?
- Concernant les données supplémentaires :
- Nous avons ajouté 17 690 participants au génotype répertorié dans cette 9e édition.
- Pour les chercheurs qui préfèrent que les données brutes soient inversées et alignées, nous proposons les génotypes_bruts_inversés en plus des génotypes_bruts.
- Concernant les identifiants d’échantillons :
- Le préfixe «m» utilisé pour les cohortes recrutées au début par le réseau monogénique a été abandonné.
- Le suffixe «s» pour la nomenclature des échantillons du GP2 devient obsolète. Le chiffre de l’échantillon est encore disponible via la clé maître pour permettre d’établir des correspondances avec les identifiants émis précédemment mais, les identifiants des échantillons du GP2 dans les fichiers génétiques vont cesser d’utiliser le suffixe «s».
- Les identifiants PPMI GP2 dans tous les dossiers sont en cours de mise à jour pour inclure les identifiant PATNO et faciliter la tache aux chercheurs qui travaillent sur plusieurs plateformes.
- Nous avons ajouté 17 690 participants au génotype répertorié dans cette 9e édition.
Echantillons RGPD strictement locaux via Verily Viewpoint Workbench Nous sommes heureux d’annoncer que certains utilisateurs pourront avoir accès aux échantillons restreints localement, également dénommés échantillons régis par le Règlement général sur la protection des données (RGPD), grâce à notre collaboration avec Verily Viewpoint Workbench. A ce stade, alors que le GP2 continue de déployer ses solutions de partage de données pour les données protégées par le RGPD, la neuvième édition ne sera disponible que pour les membres du consortium GP2 et ses partenaires. Au fur et à mesure que les tests et la mise en œuvre se poursuivent, en 2024, ces solutions seront mises à la disposition d’un plus grand nombre de chercheurs. Tous les échantillons de la neuvième édition se trouvent sur Workbench, tandis que tous ceux non régis par le RGPD sont disponibles sur Terra (comme pour les éditions précédentes). Pour avoir accès à l’intégralité des publications sur VWB, vous devez :
- Disposer d’une autorisation d’accès GP2 de catégorie 2.
- Remplir leformulaire de demanded’échantillons régis par le RGPD.
- Être membre du consortium GP2 (contribution de cohorte, partenaire GP2 ou membre de l’équipe d’analyse de projets).
Données cliniques Cette édition contient les données cliniques de 71 835 personnes dont les données génétiques et les données cliniques de base sont accessibles. Cette édition comprend les données précises du phénotype et des données génétiques de 16 800 personnes. Ces données incluent :
- Âge au moment du diagnostique
- Diagnostique primaire, actuel et suivant
- Examens cognitifs tels que le Mini-Mental State Examination (MMSE) et le Montreal Cognitive Assessment (MoCA)
- Révision de l’échelle UPDRS pour la maladie de Parkinson, parrainée par la Movement Disorder Society
- Détail des «autres» phénotypes, tels que la démence à corps de Lewy
- Les cas recrutés par l’intermédiaire du réseau monogénique sont classés dans la catégorie « Autres »
Données à l’échelon individuelNous extrayons désormais les données à partir d’un total de 104 cohortes. Veuillez consulter le tableau de bord des cohortes du GP2 pour obtenir des informations complémentaires au sujet des cohortes qui ont été partagées. Les participants au GP2 à l’ascendance génétiquement déterminée sont regroupés en onze groupes ; le tableau ci-dessous détaille l’ascendance des participants à génotype répertorié dans cette édition, qui ont passé le contrôle de qualité et ont été comptabilisés. Ces chiffres sont le reflet des échantillons des éditions précédentes, réorganisés selon l’actualisation du dossier des groupes et font l’objet d’un contrôle de qualité, tout comme les nouveaux échantillons de génotype, propres à cette édition. 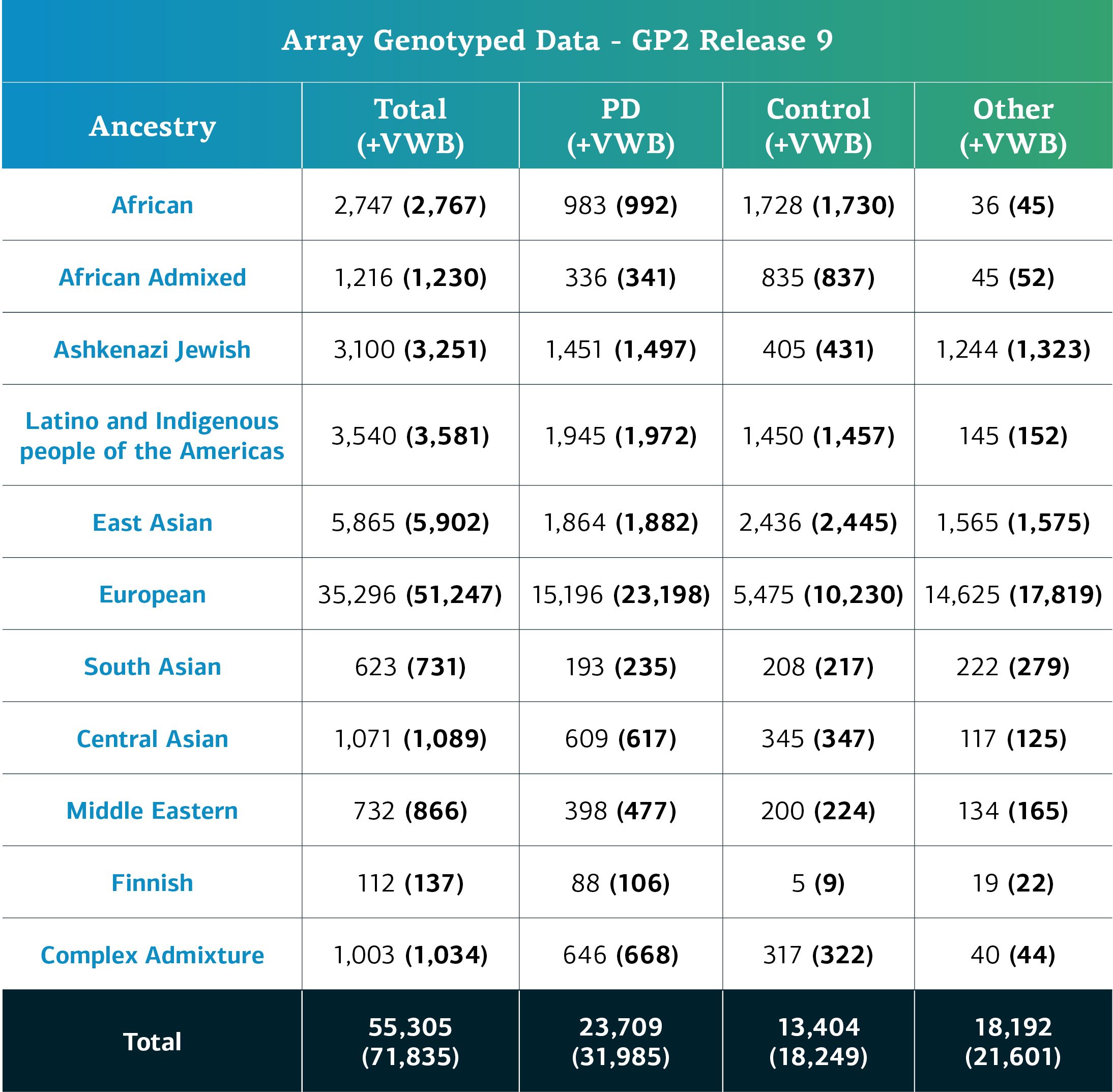 Les futures diffusions de données contribueront à renforcer la diversité du groupe de participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès. Les utilisateurs ayant un accès de catégorie 2 peuvent d’ores et déjà explorer les données sur notre navigateur de cohortes, dont nous avons parlé dans un précédent blog. Comme toujours, veuillez consulter le document README qui accompagne chaque édition du GP2 pour obtenir plus d’informations sur le contrôle de qualité, les canaux, les données et les analyses !
Les futures diffusions de données contribueront à renforcer la diversité du groupe de participants disponibles. Vous pouvez consulter notre tableau de bord pour suivre nos progrès. Les utilisateurs ayant un accès de catégorie 2 peuvent d’ores et déjà explorer les données sur notre navigateur de cohortes, dont nous avons parlé dans un précédent blog. Comme toujours, veuillez consulter le document README qui accompagne chaque édition du GP2 pour obtenir plus d’informations sur le contrôle de qualité, les canaux, les données et les analyses !











