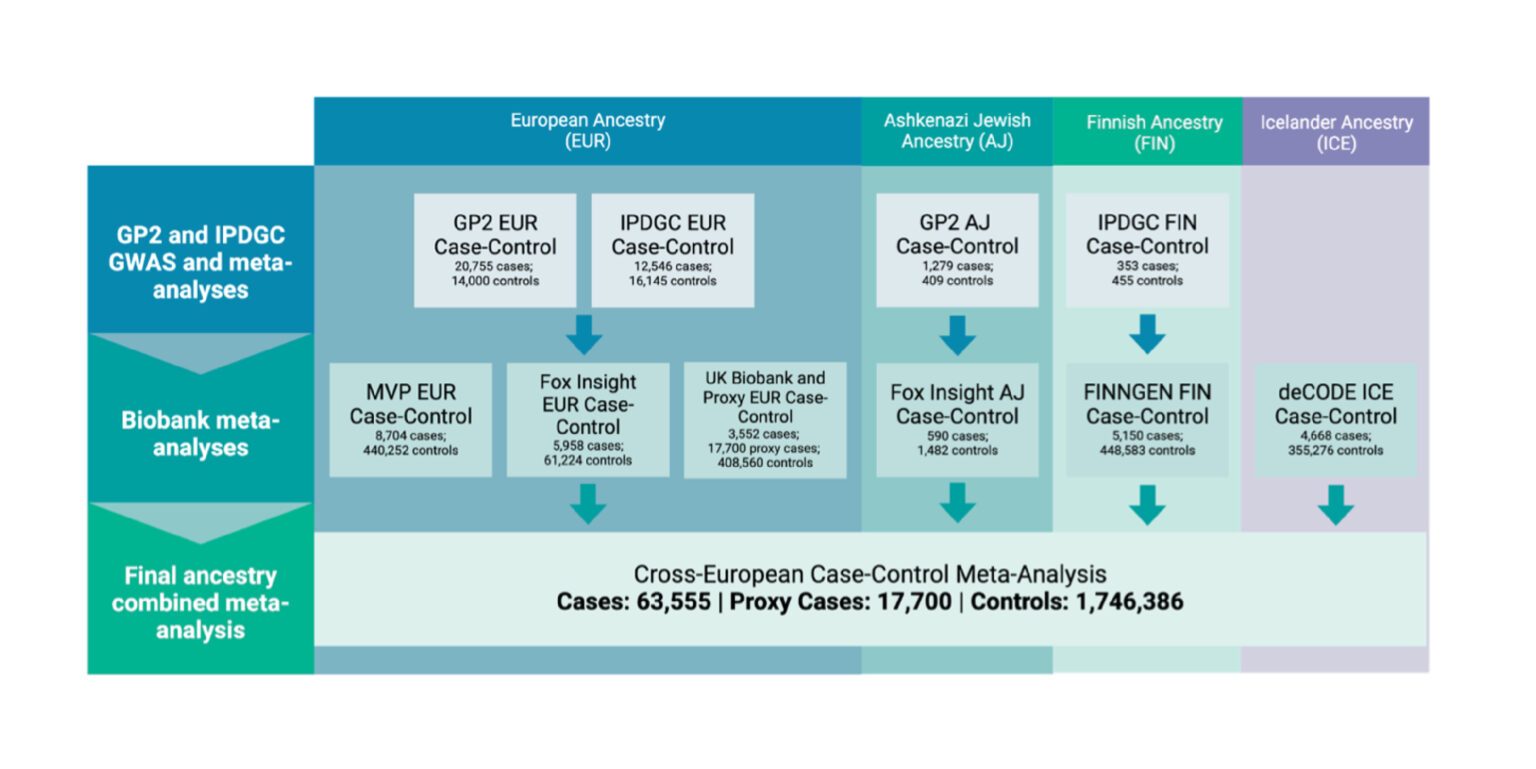
Wir freuen uns, ankündigen zu können, dass der Preprint der nächsten Generation von Meta-Analysen genomweiter Assoziationsstudien zur Parkinson-Krankheit (Meta-GWAS) von GP2 jetzt verfügbar ist. Hierbei handelt es sich um eine bahnbrechende Studie. Sie umfasst 63.555 Fälle, 17.700 Proxy-Fälle mit Parkinson in der Familiengeschichte und 1.746.386 Kontrollfälle – womit sie die bislang umfassendste Untersuchung zum genetischen Risiko für die Parkinson-Krankheit darstellt. Sie veranschaulicht, welch große Wirkung Open Science, Open Data und einheitliche Datengenerierungs- und Analyseprozesse bei globalem Einsatz in der Cloud entfalten können.
Wichtige Erkenntnisse aus der Studie
Dank unserer Erkenntnisse konnten wir das Wissen über die Ätiologie der Parkinson-Krankheit ausbauen. Diese umfassen:
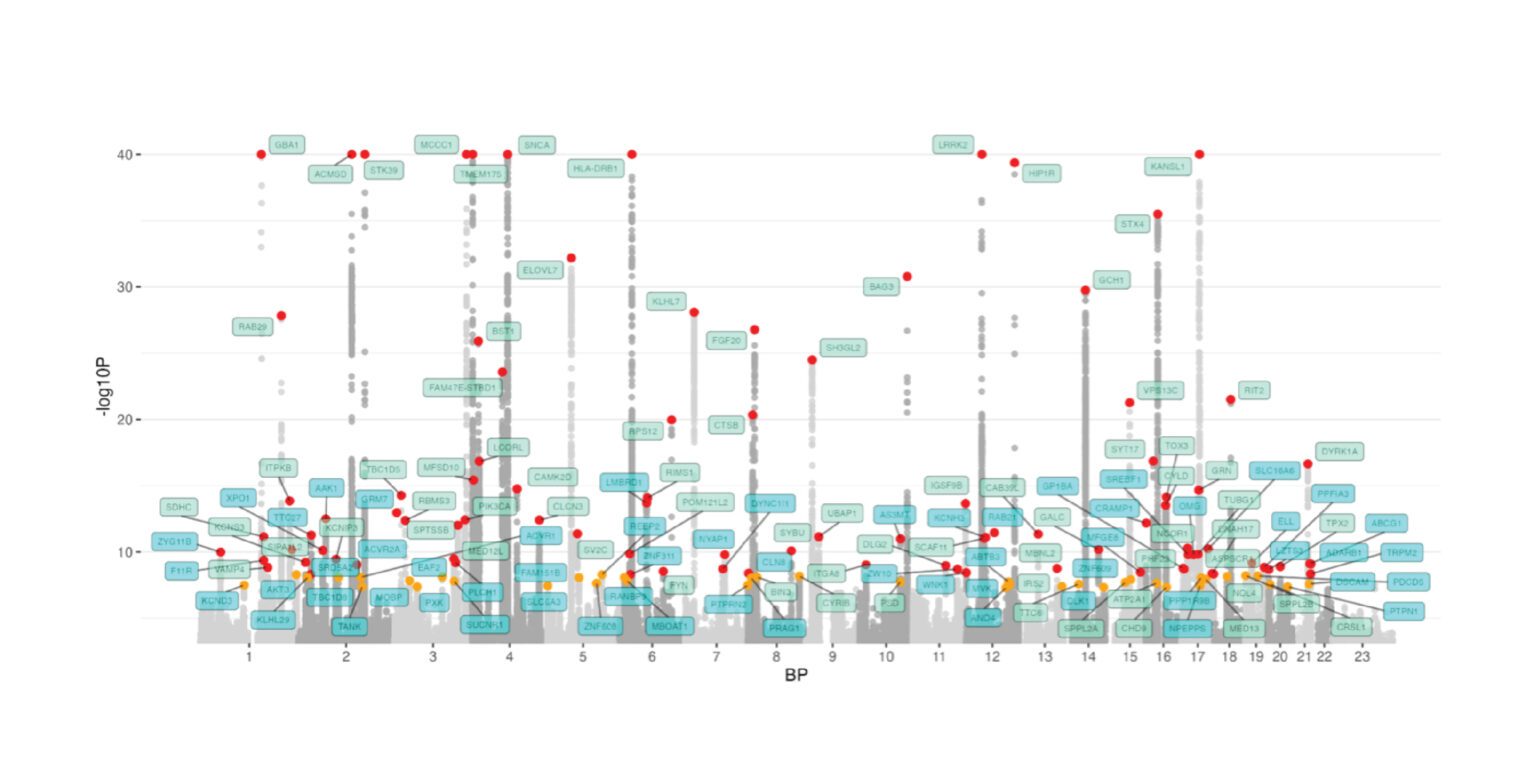
- Identifizierung von 134 Risikoloci (59 neuartigen) und insgesamt 157 unabhängigen Risikovarianten über diese Loci hinweg, wodurch unser Wissen über das Parkinson-Risiko erheblich erweitert wird.
- Entdeckung (mittels Integration von Multi-Omics-Daten) einer hochgradigen Anreicherung der Expression der nominierten Risikogene in Hirngewebe, insbesondere in neuronalen und astrozytären Zelltypen.
- Priorisierung von 33 Hochkonfidenzgenen aus den 134 Loci für zukünftige Follow-up-Studien.
Danksagung/Acknowledgements
Diese Arbeit ist das Ergebnis einer intensiven Kooperation mit Beiträgen von Hunderten von Mitwirkenden aus aller Welt. Wichtige Beiträge wurden unter anderem von folgenden Personen geleistet: Hampton L. Leonard, Mary B. Makarious, Dan Vitale und Mike A. Nalls erstellten das Analysekonzept, analysierten die Daten und verfassten das ursprüngliche Manuskript. Astros Th. Skuladottir, Vala Palmadottir, Hreinn Stefansson und Kari Stefansson erstellten das Analysekonzept und analysierten die Daten für das isländische (ICE) Abstammungs-GWAS von deCODE. Kristin Levine, Hampton L. Leonard, Nicole Kuznetsov, Mary B. Makarious, Dan Vitale und Mat Koretsky haben Beiträge zur Qualitätskontrolle der genetischen Daten von GP2 geleistet und sie der Forschungsgemeinschaft zur Verfügung gestellt. Cornelis Blauwendraat und Andrew B. Singleton übernahmen eine führende Rolle beim Studiendesign, der Datenlogistik und der Finanzierung der Studie. Huw Morris, Manuela Tan, Hirotaka Iwaki, Simona Jasaityte, Ellie Stafford, Lietsel Jones, Shannon Ballard, J. Solle und Claire Wegel (Arbeitsgruppen „Komplexe Krankheit“ und „Compliance“) erstellten das Konzept für die komplexe Studie von GP2, die Generierung und Aufbereitung klinischer und genetischer Daten sowie die Logistik im Bereich Rechtliches/Datenaustausch. Alle übrigen GP2-Mitglieder (Mitwirkende) haben Daten beigesteuert und diesen Artikel kritisch überarbeitet. Ein besonderer Dank geht an Noel Burtt und ihr Team vom Neurodegenerative Disease Knowledge Portal, die die zusammenfassenden Statistiken bereits Monate im Voraus bereitgestellt haben.