Übersicht
Im Dezember 2025 kündigte GP2 die 11. Datenfreigabe in Kooperation mit AMP® PDauf derTerra-Plattform und der Verily® Workbench an. Diese Freigabe umfasst 20.842 zusätzliche genotypisierte Teilnehmende, 17.153 zusätzliche WGS-Teilnehmende und 4.232 zusätzliche klinische Exom-Daten.
- Die Genotyp-Array-Daten (NBA), einschließlich Proben mit lokalen Einschränkungen, umfassen nun insgesamt 103.786 genotypisierte Teilnehmende (46.327 mit Parkinson-Erkrankung, 28.857 Kontrollpersonen und 28.602 „sonstige“ Phänotypen).
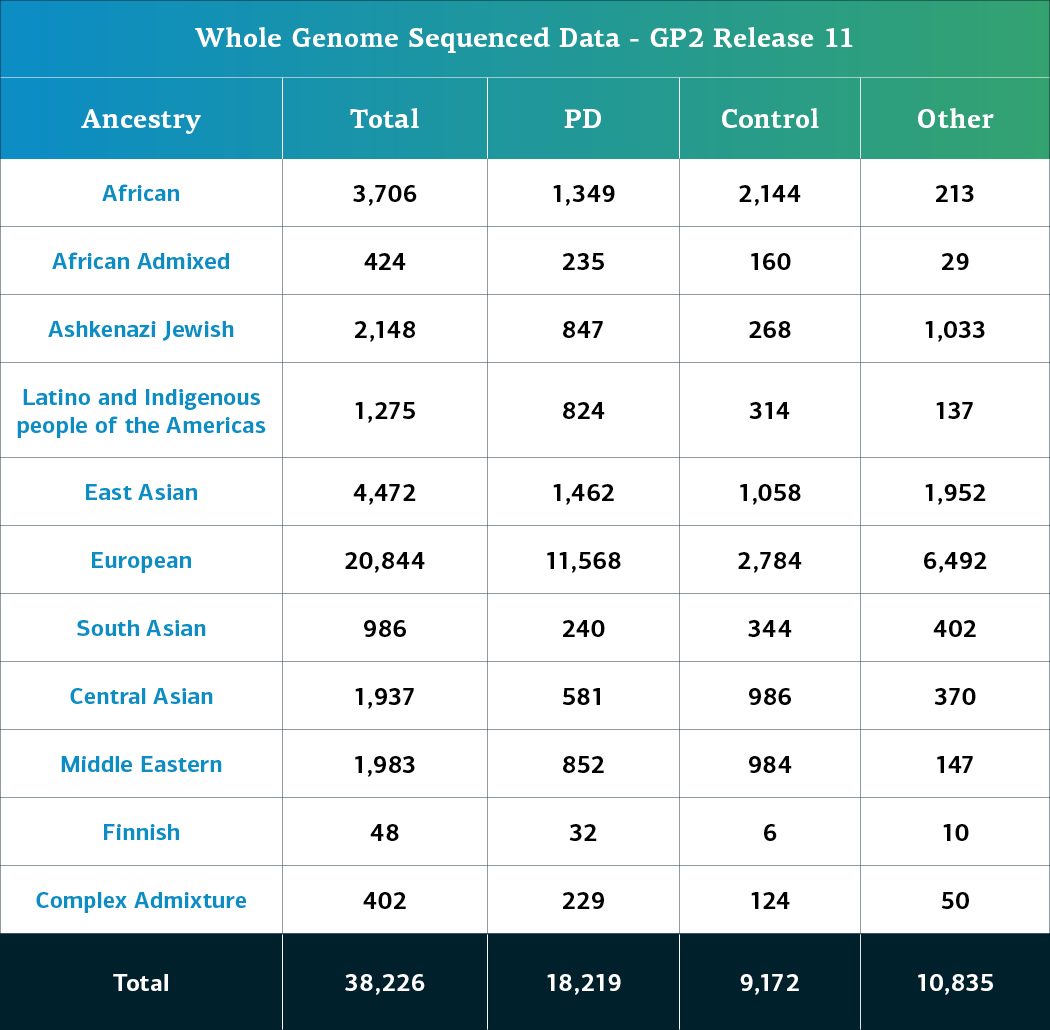
- Die WGS-Daten umfassen nun insgesamt 38.226 sequenzierte Teilnehmende (18.219 mit Parkinson-Erkrankung, 9.172 Kontrollpersonen und 10.835 „sonstige“ Phänotypen).
- Die klinischen Exom-Daten bestehen nun aus 14.686 Proben mit Parkinson-Erkrankung.
- Von den 122.317 einzigartigen Proben mit genetischen Daten (NBA, WGS oder klinische Exom-Daten) haben 32.897 Personen auch zusätzliche erweiterte klinische Informationen.
Was ist in dieser Freigabe neu?
Erweiterung der genomischen Daten Mit dieser Freigabe wird die Zahl der Teilnehmenden mit verfügbaren genetischen Daten erheblich erweitert. Folgendes wurde hinzugefügt:
- 20.842 neue Teilnehmende mit Genotyp-Array-Daten (NBA)
- 17.153 neue Teilnehmende mit WGS-Daten (Gesamtgenom-Sequenzierung)
- 5.915 neue Teilnehmende mit erweiterten klinischen Daten
- Eine Familiendatei (und ein entsprechendes Datenverzeichnis) mit Schätzwerten betreffend die paarweisen Verwandtschaftsbeziehungen zwischen Einzelpersonen innerhalb von Familien. Dies beinhaltet sowohl abgeleitete Beziehungen (mit Verwandtschaftskoeffizienten) als auch berichtete Beziehungen.
Joint-Calling beinhaltet nun auch AMP® PD-Kohorten
- Die per Joint-Calling umgesetzten WGS-Variantensätze beinhalten nun auch Proben aus den folgenden sieben AMP® Parkinson-Kohorten: BioFIND, HBS, PDBP, PPMI, LCC, STEADY-PD3 und SURE-PD3.
- Eine Verarbeitung dieser Proben gemeinsam mit GP2 statt einer unabhängigen Verarbeitung minimiert Fehlstellen und Artefakte und verbessert die Genauigkeit des Genotyps.
- Wir haben dem Master-Key eine Spalte hinzugefügt, die anzeigt, welche GP2-Proben auch im AMP-PD-Datensatz enthalten sind.
Neue zusammenfassende Statistiken jetzt verfügbar Wir haben verschiedene zusätzliche Datensätze zu den zusammenfassenden Statistiken der GWAS in Tier 1 zur Verfügung gestellt:
- Blauwendraat et al PD age at onset (AAO) meta-analysis paper
- Blauwendraat et al PD autosomal sex-stratified meta-analysis paper
- Blauwendraat et al PD GBA1-modifier meta-analysis paper
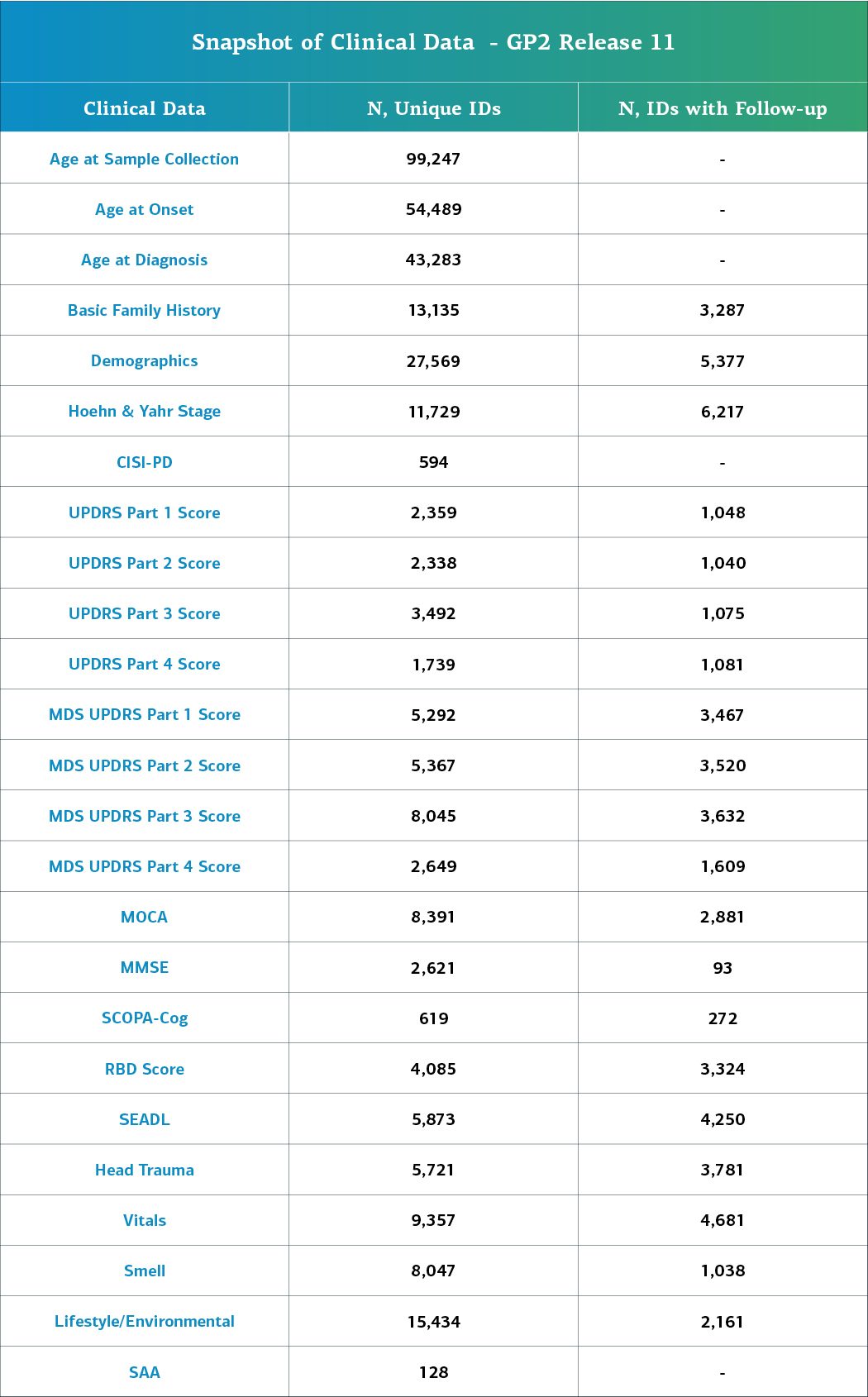
Klinische Daten Diese Freigabe enthält klinische Daten von insgesamt122.317 Personen, für die genetische und klinische Kerndaten verfügbar sind. Für 32.897 von diesen Personen stehen tiefe klinische Phänotypisierungsdaten zur Verfügung. Diese Informationen umfassen:
- Alter bei Diagnose und Ausbruch
- Primäre, aktuelle und letzte Diagnose
- Kognitive Tests wie der Mini-Mental Status-Test (MMST) und der Montreal-Cognitive-Assessment-Test (MoCa)
- Von der Movement Disorder Society gesponserte Überarbeitung der Unified Parkinson’s Disease Rating Scale (MDS-UPDRS)
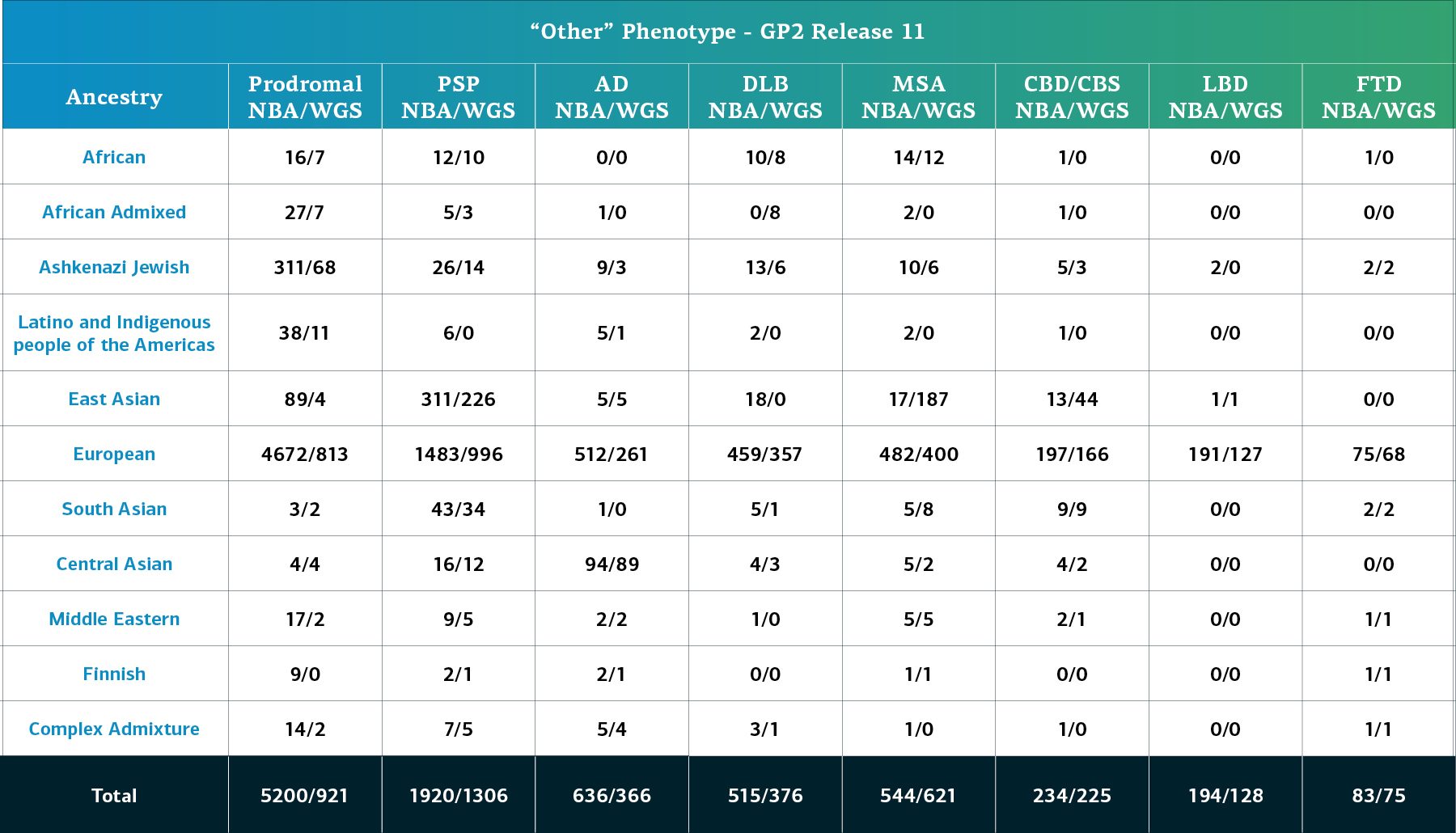
- Detaillierte „sonstige“ Phänotypen wie etwa Lewy-Körper-Demenz (LBD)
Individuen-spezifische Daten Wir erfassen nun die Daten von insgesamt 148 Kohorten. Weitere Informationen zu den bereitgestellten Kohorten finden Sie im GP2 Cohort Dashboard. Die genetisch determinierte Abstammung von Array-genotypisierten GP2-Teilnehmenden wird in 11 Abstammungsgruppen gegliedert; die Tabellen unten geben Aufschluss über die genetisch determinierte Abstammung der Teilnehmenden in dieser Freigabe, die die Qualitätskontrolle für Array-Daten und Gesamtgenom-Sequenzierungsdaten durchlaufen haben. Diese Zahlen spiegeln Proben aus früheren Freigaben wider, die unter Verwendung der aktualisierten Clusterdatei neu geclustert und einer Qualitätskontrolle unterzogen wurden, sowie neu genotypisierte Proben, die ausschließlich in dieser Freigabe enthalten sind. Die letzte Tabelle gibt Aufschluss über die genetisch determinierte Abstammung ausgewählter sonstiger Phänotypen ohne Parkinson. 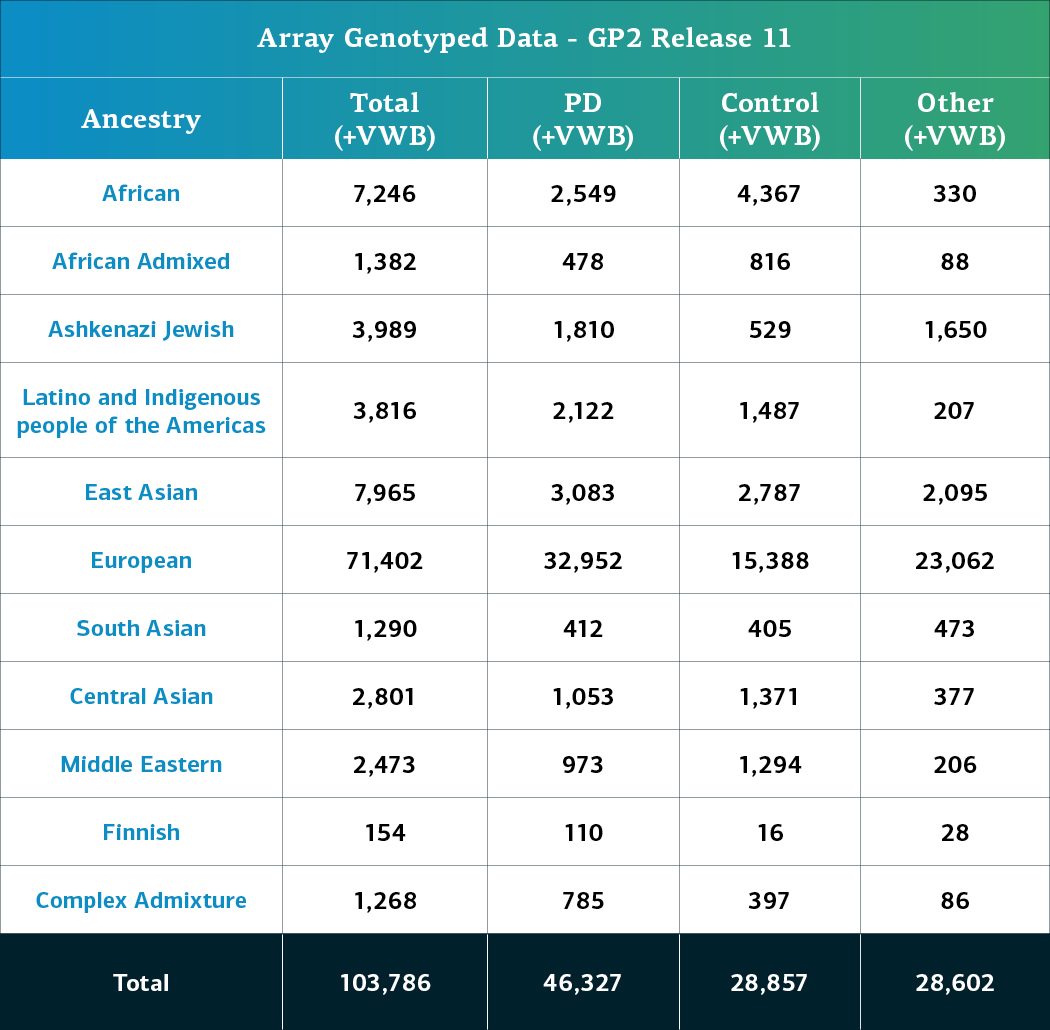
Datenzugang
DSGVO-Proben mit lokalen Einschränkungen über die Verily Viewpoint Workbench Wir stellen über unsere Zusammenarbeit mit der Verily Viewpoint Workbench weiterhin einen Pilotzugang zu lokal begrenzten Proben bereit, d. h. Proben, die der Datenschutzgrundverordnung (DSGVO) unterliegen. Wenn Sie Zugang zur gesamten Freigabe auf der VWB erhalten möchten, müssen Sie:
- über einen bewilligten GP2-Tier-2-Zugang verfügen,
- das Antragsformular für DSGVO-Proben ausfüllen,
Durch zukünftige Datenfreigaben wird sich die Vielfalt der verfügbaren Teilnehmenden weiter erhöhen. Auf unserem Dashboard können Sie sich über den Fortschritt unserer Arbeiten informieren. Wenn Sie bereits über einen Tier-2-Zugang verfügen, können Sie die Daten in unserem Cohort Browser ansehen, der bereits in einem früheren Blogbeitrag vorgestellt wurde. Wie immer finden Sie weitere Details zu den Themen Qualitätskontrolle, Pipelines, Daten und Analysen in der README-Datei zur jeweiligen GP2-Freigabe!










